遥感数据介绍—Sentinel 2A
今天介绍一下Sentinel卫星以及一些预处理的方法。
1.基本信息(成像仪/重访周期/波段数/分辨率)
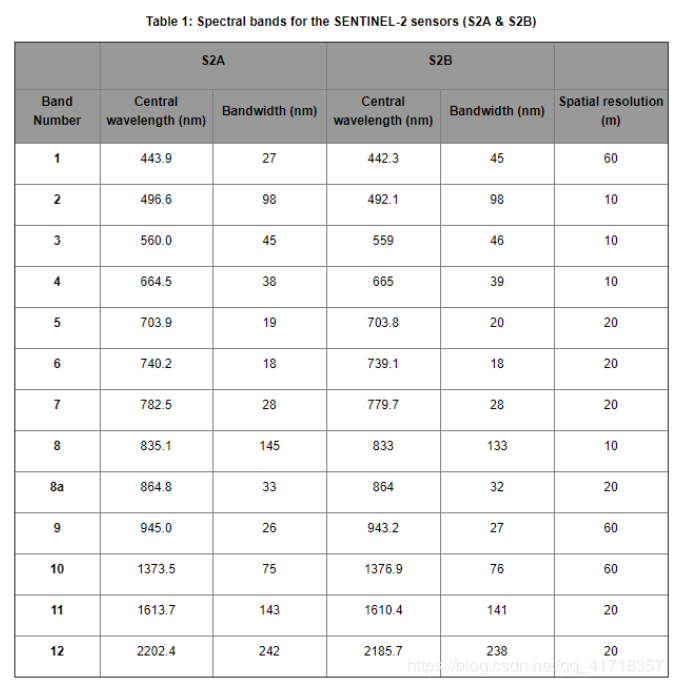
哨兵2号是高分辨率多光谱成像卫星,携带一枚多光谱成像仪(MSI),用于陆地监测,可提供植被、土壤和水覆盖、内陆水路及海岸区域等图像,分为2A和2B两颗卫星,哨兵,2B与2015年6月发射的哨兵-2A卫星为同一组。哨兵-2号卫星高度为786km,覆盖13个光谱波段,幅宽达290千米。地面分辨率分别为10m、20m和60m、一颗卫星的重访周期为10天,两颗互补,重访周期为5天。从可见光和近红外到短波红外,具有不同的空间分辨率,在光学数据中,哨兵-2号数据是唯一一个在红边范围含有三个波段的数据,这对监测植被健康信息非常有效。
下载网站:https://scihub.copernicus.eu/dhus/#/home
Ps:夜里1:00下载会很快
成像仪:MSI 重访周期:5-10天 波段数:13 分辨率:10m/20m/60m
2.产品等级及插件介绍
欧空局仅发布了哨兵2号的L1C级多光谱数据(MSI),Sentinel-2 L1C是经过正射校正和几何精校正的大气表观反射率产品,并没有进行大气校正。同时,ESA还对S2 L2A级数据进行了定义,L2A级数据主要包含经过大气校正的大气底层反射率数据(Bottom-of-Atmosphere corrected reflectance),这个数据可以通过Sen2cor插件自行生产。
插件说明:1.Sen2cor 分为2.05/2.08版本,前者用于处理290km宽幅的16年老数据,后者用于处理新数据。L2A Process + 文件名完成辐射定标+大气校正。
2.Sen2Res,提供超分辨率合成功能。这是一个nbm文件,由于Sentinel2有10/20/60m三个分辨率的遥感数据,所以在对图像进行其他处理之前,需要先统一到一个分辨率,可以用重采样或者是超分辨率合成。具体操作步骤是在Snap中的plugins里添加已下载的插件并安装,此外该nbm文件还可以通过python调用实现批处理功能。
3.文件夹介绍
介绍一下数据的内容,这里分为16年和19年的数据,由于16年的影像是290km幅宽,所以存储方式和19年有所不同。
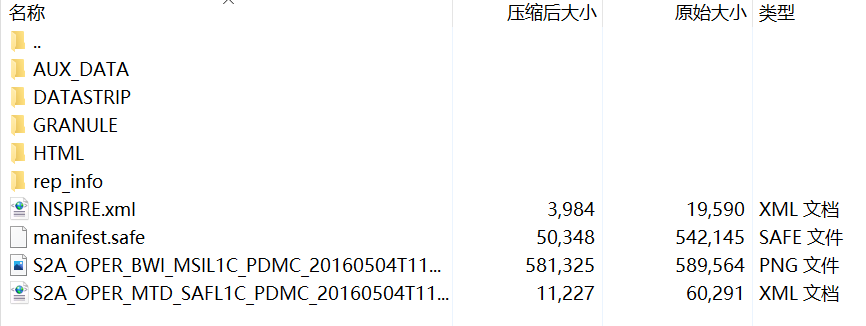
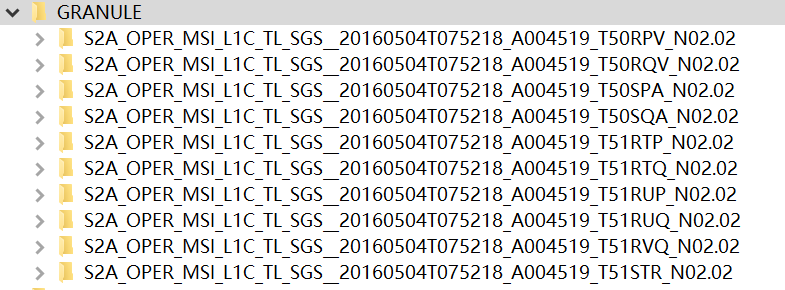
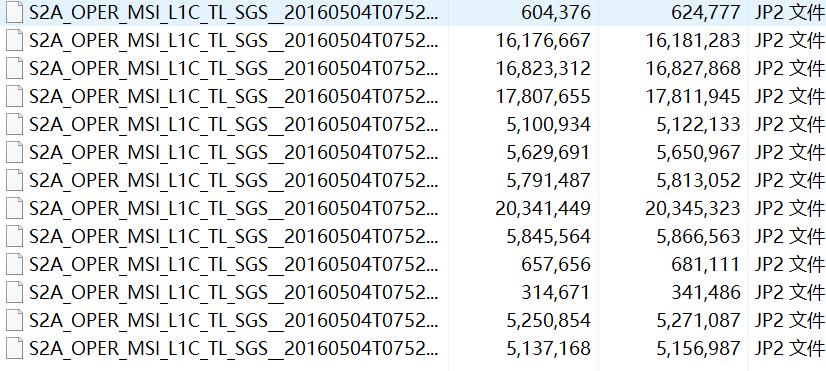
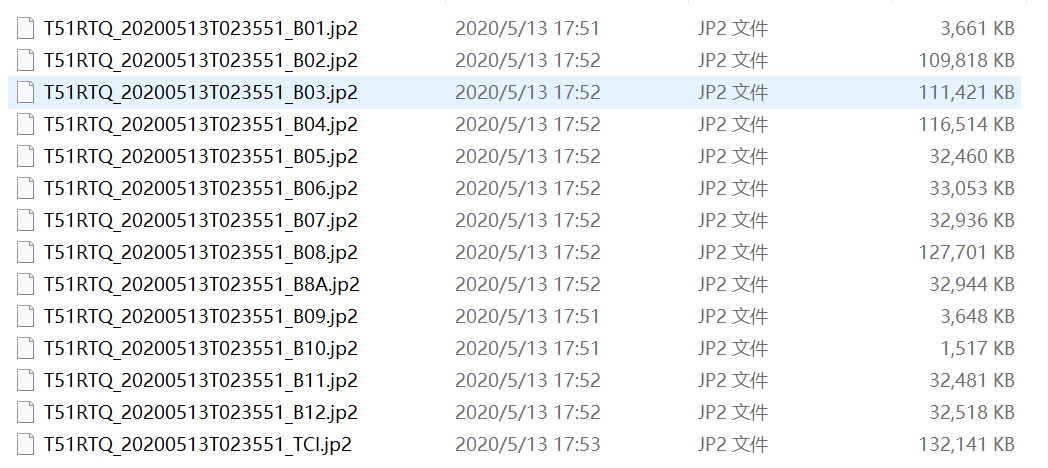
首先是16年的数据。其中xml文件是索引文件,索引到存储的数据,具体存放波段数据的文件是GRANULE文件夹,用10个文件夹把大图幅切片分别存储,打开某个图幅的IMG_DATA文件夹存储了13个波段的影像数据,jp2格式。通过最外层索引文件控制内层的文件,直接打开所有数据。也可以通过内层文件夹的xml文件打开单个图幅的数据。AUX_DATA为辅助文件,可能存储 太阳高度角、日地距离等信息。
19年的数据存储大小发生改变,减小了单幅图像的幅宽。GRANULE文件夹中只存放了一个图幅,存储所有波段数据(图4)。
接着对上述L1C级产品加工,做大气校正,得到L2A级产品,数据格式有所不同。校正后IMG_DATA把数据按照分辨率不同分成了三个文件夹进行存储。Sentinel2A数据没有全色波段,因此他提供了10m分辨率的四个波段数据,用于完成图像的超分辨率。此外10波段作为卷云波段,不进行大气校正,没有提供,需要从L1C产品获取取。
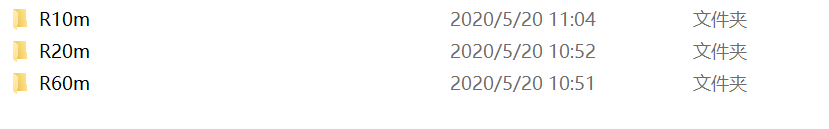
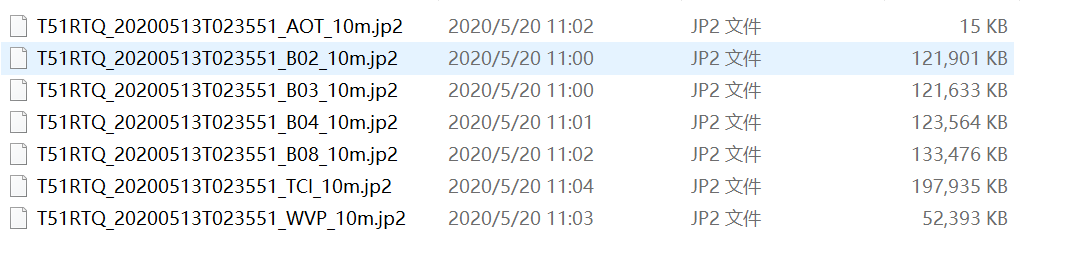
完成图像大气校正后,由于不同波段的分辨率各不相同,所以需要重采样到同一个分辨率,或者用Sen2Res完成超分辨率合成,速度较慢,不过质量很好。这样才能进行下一步处理所有波段分辨率统一了才能进行下一步处理,可以Snap保存为ENVI格式或者GTiff等等再做运算,这样计算NDVI等指数也就方便了。
L2A产品的文件里还有相关的分类文件,分类了植物、云、雪、水体等等,还有对应的index,但是精度太低。
4.数据的读取。
1)Snap软件:打开xml索引文件从而打开所有波段数据,或者打开单个文件xml来打开单个波段。
2)Python+GDAL
JP2格式的图像数据可以用GDAL读取,并且可以读取到文件的投影信息和地理参数,有需要可以用GDAL读取并且转换成Tiff文件。
这里还发现数据集ds可以直接调用ds.ReadAsArray()读取数组阵列,真是有点奇葩。。。我还一直以为ds只能ds.ReadRaster呢。
from osgeo import gdal import numpy as np filename = r'S2A.jp2' class sentinel: def read_img(self, filename): ds = gdal.Open(filename) Xsize = ds.RasterXSize Ysize = ds.RasterYSize bnum = ds.RasterCount data = ds.ReadAsArray(0, 0, Xsize, Ysize) #太奇葩了,ds都能ReadAsArray() proj = ds.GetProjection() geotrans = ds.GetGeoTransform() return proj, geotrans, data, Xsize, Ysize, bnum def write_img(self, filename, outfile): proj, geotrans, data, width, height, bands = self.read_img(filename) driver = gdal.GetDriverByName('GTiff') ds = driver.Create(outfile, width, height, bands, gdal.GDT_UInt16) ds.SetGeoTransform(geotrans) ds.SetProjection(proj) if bands==1: ds.GetRasterBand(1).WriteArray(data) else: for i in range(1, bands+1): ds.GetRasterBand(i).WriteArray(data) del ds run = sentinel() proj, geotrans, data = run.read_img(filename) print(np.shape(data))
另外贴一下16年数据的读取代码,有所不同。
from osgeo import gdal import os #这里打开了一个哨兵2A L1C zip文件 filename = (' S2A_MSIL1C_***.zip') root_ds = gdal.Open(filename) # 返回结果是一个list,list中的每个元素是一个tuple,每个tuple中包含了对数据集的路径,元数据等的描述信息 #读取所有子数据集 ds_list = root_ds.GetSubDatasets() visual_ds = gdal.Open(ds_list[0][0]) # 取出第一个图幅的第一个波段数据 visual_arr = visual_ds.ReadAsArray() # 将数据集中的数据转为ndarray del visual_arr # 获得栅格数据的一些重要信息 print(f'打开数据为:{ds_list[0][1]}') print(f'投影信息:{visual_ds.GetProjection()}') print(f'栅格波段数:{visual_ds.RasterCount}') print(f'栅格列数(宽度):{visual_ds.RasterXSize}') print(f'栅格行数(高度):{visual_ds.RasterYSize}')
以上就是本次学习Sentinel2A的一些心得,其实仔细看看发现和Landsat8都是有共通之处的。下次会介绍一下Sentinel3A卫星,这个数据的存储方式好像都是.nc文件,有点意思。





