WGDI学习记录之数据格式化处理(一)
参考链接
https://wgdi.readthedocs.io/en/latest/Introduction.html
https://blog.csdn.net/u012110870/article/details/113544271
一、原始数据
Acoerulea_322_v3.1.cds.fa
Acoerulea_322_v3.1.gene.gff3
Acoerulea_322_v3.1.protein.fa二、数据处理
#Gff Reformat
python 01.getgff.py Acoerulea_322_v3.1.gene.gff3 ac.old.gff#获取最长转录本的gff文件(并对gff文件中的基因名进行了替换,最后一列为替换前的基因名)以及lens文件
python 02.gff_lens.py ac.old.gff ac1s.gff ac1s.lens#修改*.cds.fa以及*.protein.fa的基因名(此处需要注意是依据ac1s.gff中第二列及最后一列的对应关系进行替换,也就是说需要确保序列中的名称是ac1s.gff最后一列的对应格式,否则会出错)
sed -i -e 's/\.v3\.1//' ac1s.gff
python 03.seq_newname.py ac1s.gff Acoerulea_322_v3.1.cds.fa ac1s.cds.fa
python 03.seq_newname.py ac1s.gff Acoerulea_322_v3.1.protein.fa ac1s.pep.fa#PS:如果需要修改,可以查询替换ac1s.gff中的最后一列查询列以确保和cds以及protein序列中的基因名对应
#使用sed替换.v3.1后缀为空
sed -i -e 's/\.v3\.1//' ac1s.gff三、排坑说明
查看cds序列,第一行>基因名称为Aqcoe0131s0001.1,而在原始的ac1s.gff中提取出来的最末一列的基因名多了.v3.1,因此需要去除.v3.1。而这个cds序列中显示的基因名称并非是基因ID因此会替换失败。
more *.cds.fa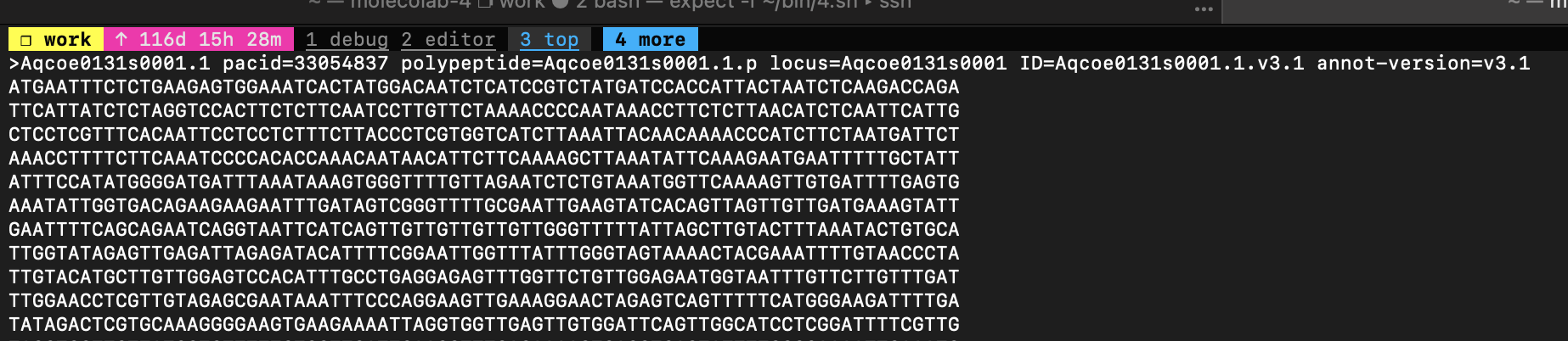
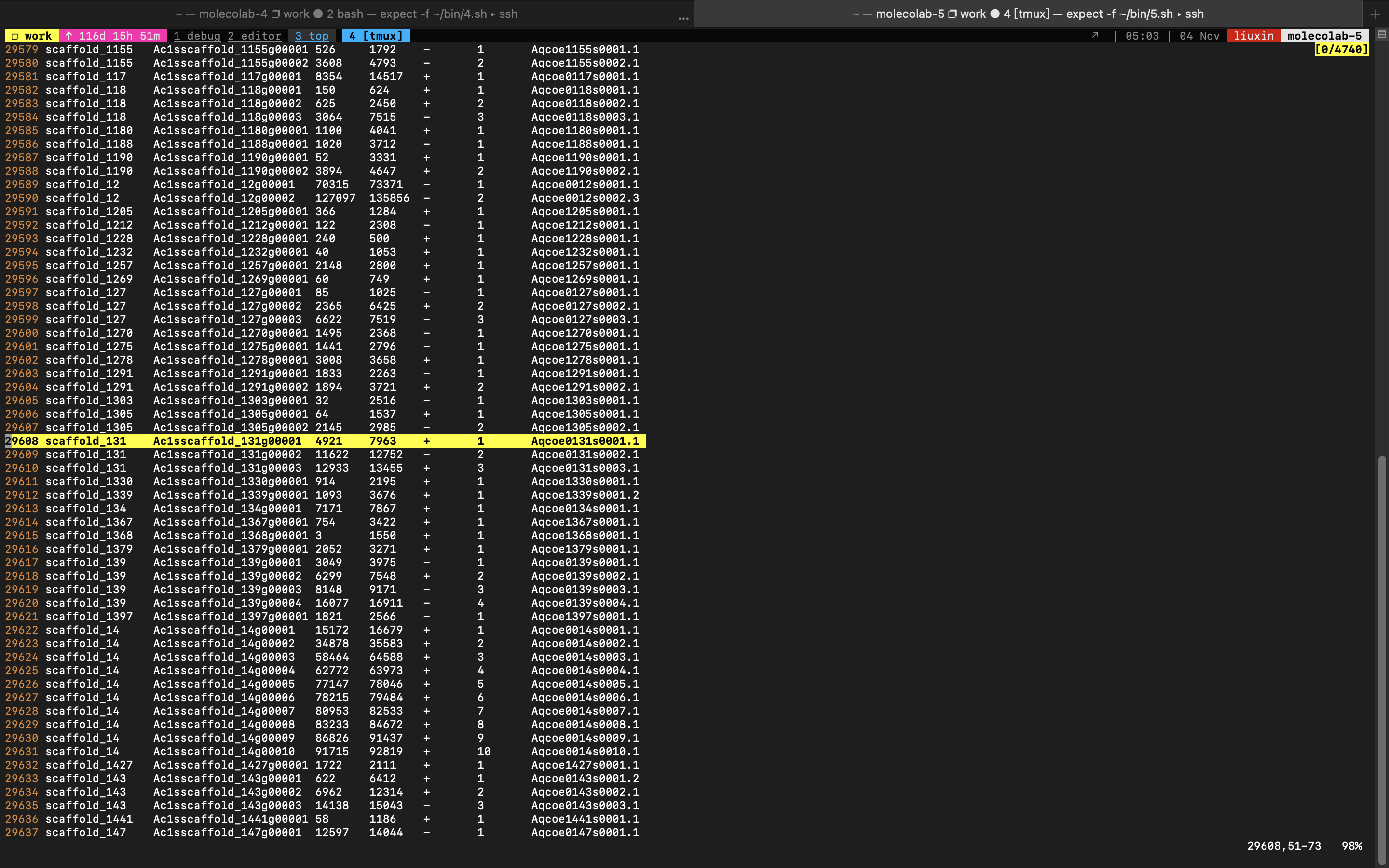
检查ac1s.gff最末一列基因名称是否对应。
vim ac1s.gff
\Aqcoe0131s0001.1