JCVI作图进阶系列(一)
尝试JCVI画多个物种间的共线性。本文尝试三物种共线性分析及作图。
参考官网教程Github官网示例流程
一、数据格式转换
###获取数据
$ python -m jcvi.apps.fetch phytozome Vvinifera,Ppersica,Tcacao
$ python -m jcvi.formats.gff bed --type=mRNA --key=Name Vvinifera_145_Genoscope.12X.gene.gff3.gz -o grape.bed
$ python -m jcvi.formats.gff bed --type=mRNA --key=Name Ppersica_298_v2.1.gene.gff3.gz -o peach.bed
$ python -m jcvi.formats.gff bed --type=mRNA --key=Name Tcacao_233_v1.1.gene.gff3.gz -o cacao.bed
$ python -m jcvi.formats.fasta format Vvinifera_145_Genoscope.12X.cds.fa.gz grape.cds
$ python -m jcvi.formats.fasta format Ppersica_298_v2.1.cds.fa.gz peach.cds
$ python -m jcvi.formats.fasta format Tcacao_233_v1.1.cds.fa.gz cacao.cds
###build .anchors
$ python -m jcvi.compara.catalog ortholog grape peach --cscore=.99 --no_strip_names --notex
$ python -m jcvi.compara.catalog ortholog peach cacao --cscore=.99 --no_strip_names --notex
###build .sample
$ python -m jcvi.compara.synteny screen --minspan=30 --simple grape.peach.anchors grape.peach.anchors.new
$ python -m jcvi.compara.synteny screen --minspan=30 --simple peach.cacao.anchors peach.cacao.anchors.new二、画图
seqids是从bed文件中获取,layout参数需要自己摸索配置。另外,若想突出某个synteny block,可以在.simple文件中添加'g*' 'r*' 'b*'
vim seqids
*******************************
chr1,chr2,chr3,chr4,chr5,chr6,chr7,chr8,chr9,chr10,chr11,chr12,chr13,chr14,chr15,chr16,chr17,chr18,chr19
Pp01,Pp02,Pp03,Pp04,Pp05,Pp06,Pp07,Pp08
scaffold_1,scaffold_2,scaffold_3,scaffold_4,scaffold_5,scaffold_6,scaffold_7,scaffold_8,scaffold_9,scaffold_10r
*******************************vim layout
*******************************
# y, xstart, xend, rotation, color, label, va, bed
.7, .1, .8, 15, , Grape, top, grape.bed
.5, .1, .8, 0, , Peach, top, peach.bed
.3, .1, .8, -15, , Cacao, bottom, cacao.bed
# edges
e, 0, 1, grape.peach.anchors.simple
e, 1, 2, peach.cacao.anchors.simple
*****************************************python -m jcvi.graphics.karyotype seqids layout --notex三、结果
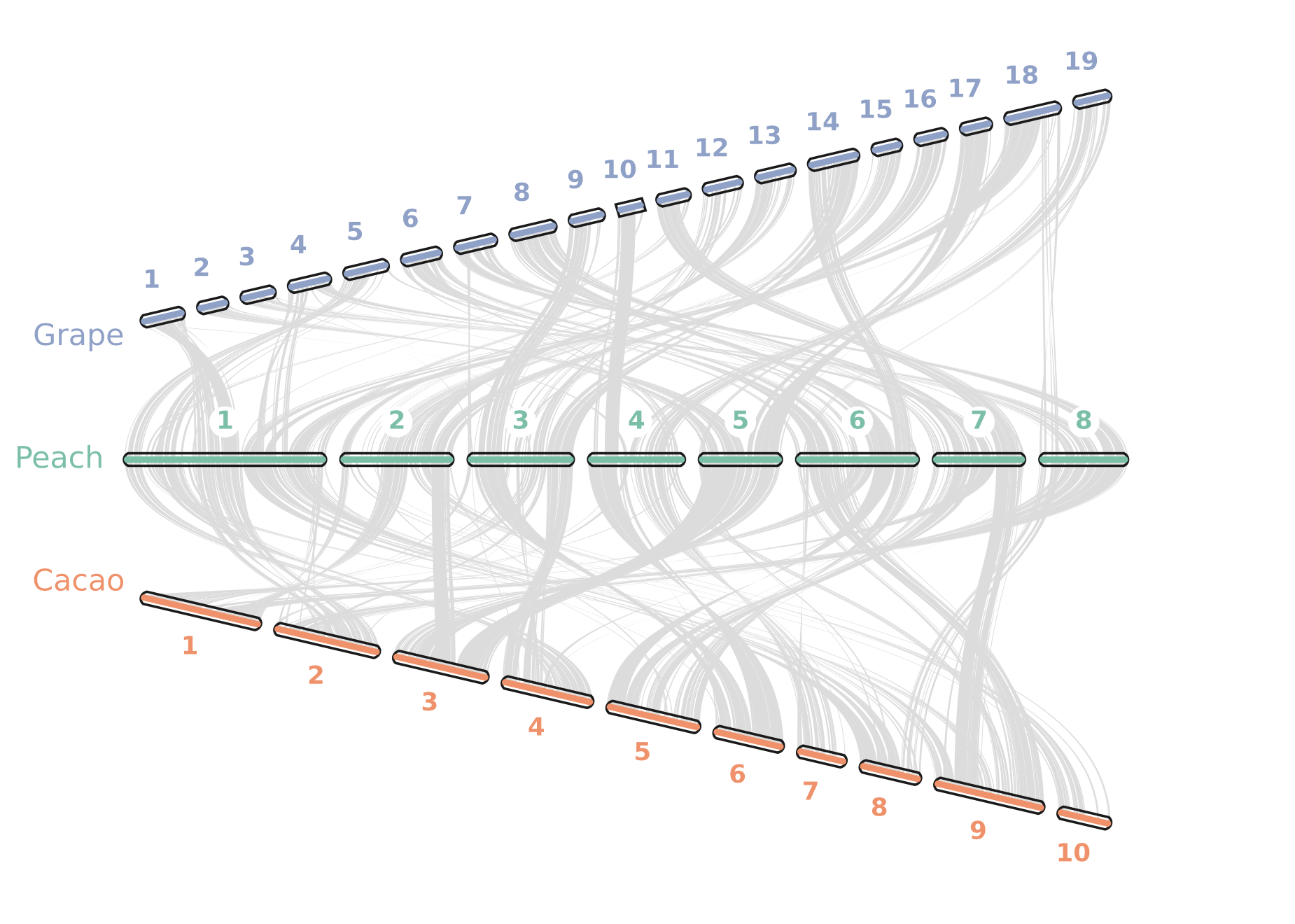
四、注意
需要注意最后画图的layout的染色体名称一定要在bed文件中可以找到,否则会出现染色体Size为0的情况,这说明layout的作图染色体在对应的bed文件中找不到。
另外,github官网的这部分教程中,给出的三物种作图存在一点小坑,第二个peach文件应该是Pp01-Pp08而不是官网给的scaffold_1-scaffold_8,经过查看bed文件才发现这一错误。(我猜测这个原因是phytome下载的数据有了更新,所以现在下载后提取出来的是id名称是Pp而不是scffold)
因此作图前需要注意,一定要检查seqids给得是否正确!(当然,实际的项目中都会注意这一点)
官网的三物种作图layout(不只是seqids的顺序要匹配layout,还有seqid中的id名也要匹配bed文件,官网这个地方第二行应该是Pp01-Pp08,否则作图会出错提示染色体size文件为空,实际上就是这个原因“seqid与bed文件中的id不匹配”)



 浙公网安备 33010602011771号
浙公网安备 33010602011771号