JCVI安装及使用
笔者在尝试JCVI安装及使用的过程中,遇到了各种各样的报错。总体来说,安装部分的报错比较好解决,官网都有解决方案。但是关于绘图相关的模块,存在各种bug,官网没有很好的解决方案。
======================================
参考地址
https://www.jianshu.com/p/214eb661fad3
https://github.com/tanghaibao/jcvi
https://github.com/tanghaibao/jcvi/wiki/MCscan-(Python-version)
======================================
一、安装
###last依赖包必须先安装
conda create -n jcvi -c bioconda last jcvi
###测试安装是否成功
conda activate jcvi
python -m jcvi.formats.fasta extract
###遇到module缺失使用pip安装
pip install more_itertools cython deap ftpretty graphviz ortools pybedtools scikit-image webcolors
###再次运行测试
python -m jcvi.formats.fasta extract###成功安装,显示如下
###还需要测试流程看每个模块是否正常工作。先跑个测试数据看一看。
二、使用
另外,需要先在phytozome官网(phytozome)注册账户,后续测试数据会用到这个数据库
conda activate jcvi
三、流程测试
1、获取测试数据,并转换格式
# 养成习惯,目录要清晰,出错也方便全部删除
mkdir ~/output/jcvi
cd ~/output/jcvi
# 载入用户名,密码完成登陆
python -m jcvi.apps.fetch phytozome
Phytozome Login: xxxxxxxx
Phytozome Password:
# 下载测试数据,下载前需先完成上面的登陆步骤
python -m jcvi.apps.fetch phytozome Vvinifera,Ppersica
# 从gff文件中提取目标bed文件
python -m jcvi.formats.gff bed --type=mRNA --key=Name Vvinifera_145_Genoscope.12X.gene.gff3.gz -o grape.bed
python -m jcvi.formats.gff bed --type=mRNA --key=Name Ppersica_298_v2.1.gene.gff3.gz -o peach.bed
# 将cds序列进行reformat
python -m jcvi.formats.fasta format Vvinifera_145_Genoscope.12X.cds.fa.gz grape.cds
python -m jcvi.formats.fasta format Ppersica_298_v2.1.cds.fa.gz peach.cds2、共线性分析
该步骤会生成*.anchors文件,这个文件包含基因位置关联信息,作图时经常用到。
# --notex不使用latex,使用latex会报错,若遇到latex调用出错的问题,可使用该参数解决
python -m jcvi.compara.catalog ortholog --no_strip_names --notex peach grape3、结果可视化
# 生成sample文件,jcvi.graphics.karyotype模块需要
python -m jcvi.compara.synteny screen --minspan=30 --simple peach.grape.anchors peach.grape.anchors.new # 配置绘制文件seqids和layout
# 将下列内容添加到seqids(vim seqids)
Pp01,Pp02,Pp03,Pp04,Pp05,Pp06,Pp07,Pp08
chr1,chr2,chr3,chr4,chr5,chr6,chr7,chr8,chr9,chr10,chr11,chr12,chr13,chr14,chr15,chr16,chr17,chr18,chr19# 配置画布 & 使用数据信息(实际应用时,该步骤的参数信息需要手动配置并多次调整才能得到最佳展示结果)
vim layout
# y, xstart, xend, rotation, color, label, va, bed
.4, .1, .8, 0, , Peach, top, peach.bed
.6, .1, .8, 0, , Grape, top, grape.bed
# edges
e, 0, 1, peach.grape.anchors.simple# 绘图
python -m jcvi.graphics.karyotype --notex seqids layout
四、结果展示(peach.grape.pdf以及karyotype.pdf)
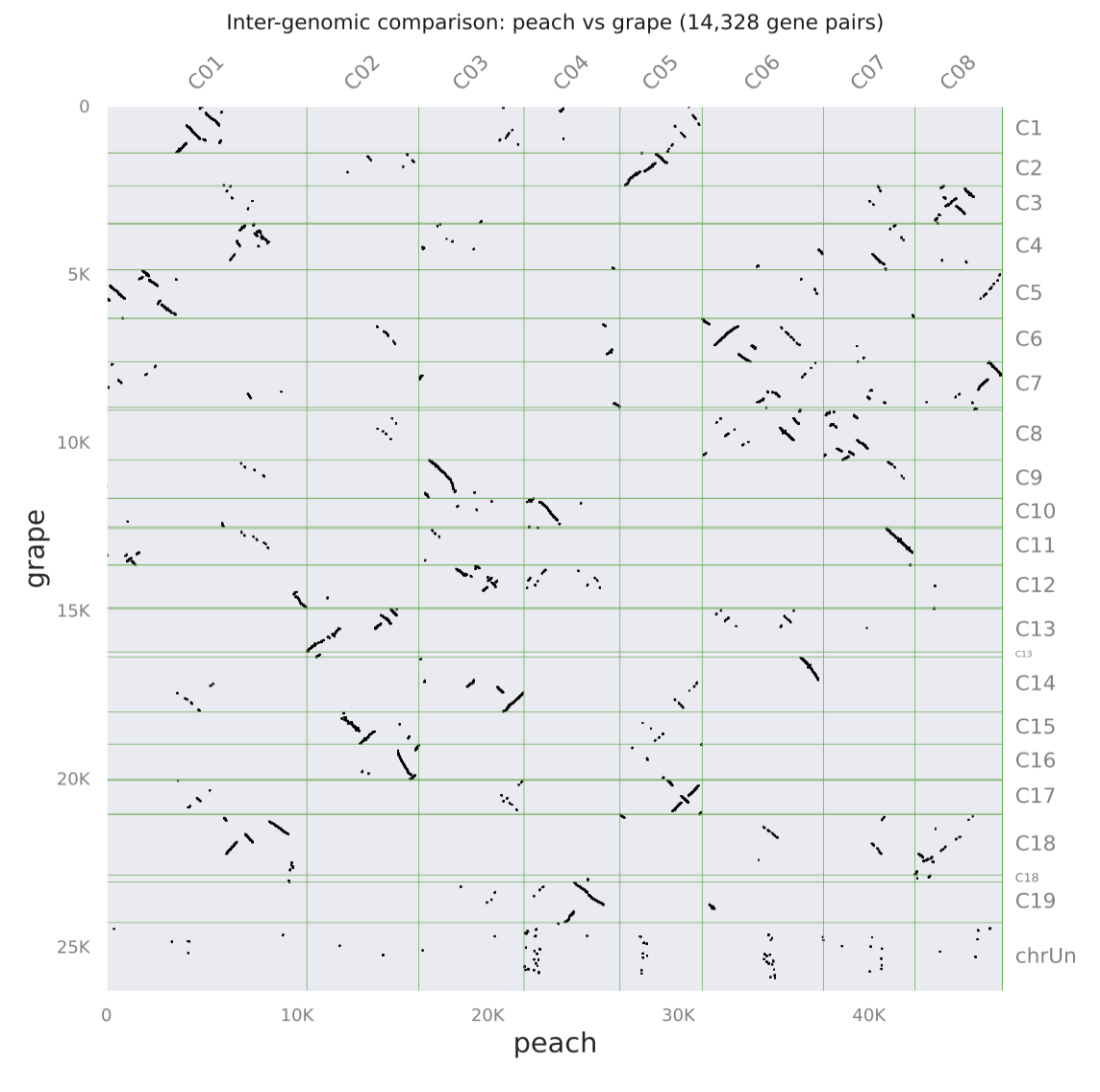
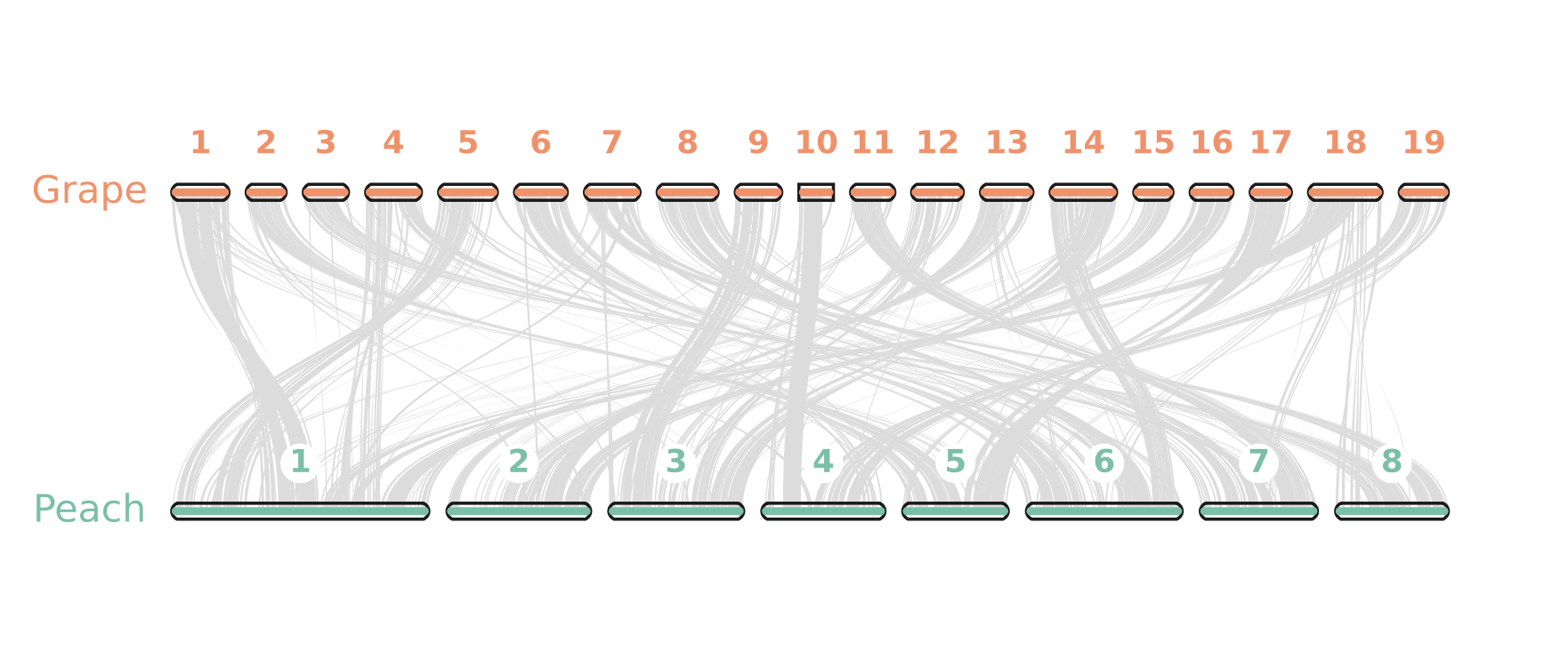
五、补充说明
#笔者遇到的主要问题是最后的pdf无法生成,主要是latex似乎无法正常工作,参考https://github.com/tanghaibao/jcvi/issues/90得以解决。
#若无法生成anchors文件,可能需要执行如下命令,处理相关font的报错。
sudo apt-get install texlive-latex-extra texlive-fonts-recommended dvipng cm-super#另外如果latex尝试各种方案过后仍无法解决,建议使用--notex执行需要作图的步骤,比如下面的过程,添加--notex可以解决由latex引起的相关错误,最后得到作图输出。
python -m jcvi.compara.catalog ortholog --no_strip_names --notex peach grape
python -m jcvi.graphics.karyotype --notex seqids layout


 浙公网安备 33010602011771号
浙公网安备 33010602011771号