使用BUSCO评估组装效果
BUSCO,全名为Benchmarking Universal Single-Copy Orthologs,依据单拷贝同源基因评估基因组组装注释的完整度。
一、安装
为避免环境冲突,建议单独配置busco的环境。
conda create -n busco busco
conda activate busco二、配置数据库文件
数据库文件地址https://busco-data.ezlab.org/v4/data/lineages/,选择相应种类的数据集下载即可,这里选用有胚植物的数据库。
mkdir BUSCO_database && cd BUSCO_database
wget -c https://busco-data.ezlab.org/v4/data/lineages/embryophyta_odb10.2020-09-10.tar.gz
tar -vxzf *.gz三、运行
conda activate busco
busco -i ~/output/20220930_banana/banana.combined.assembly.fa -l ~/BUSCO_database/embryophyta_odb10 -o hifiasm.busco -m genome -c 48四、参数
主要参数说明如下
-i 输入文件
-o 输出文件后缀
-m 分析类型:包括genome、transcriptome、proteins
-c 线程数五、结果
若正常运行结束,标准输出如下。输出目录中找到short_summary*.txt文件,查看测试基因覆盖度C=S+D,总的覆盖度为C/n。
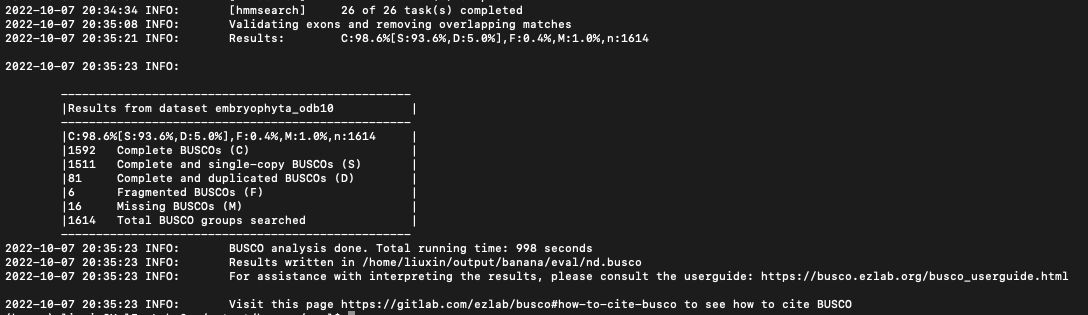
PS: 如果出现download connection problem,增加--offline参数关闭尝试下载最新的busco data,显式声明使用下载到本地的odb10。