TGS-GapCloser安装及使用
TGS代表Third Generation Sequence,TGS-GapCloser可以使用ONT reads、HiFi reads或者是初步组装的contigs进行基因组的gap filling。
此外,该工具可调用racon以及pilon处理原始reads,因此也可以输入三代的raw reads。
需要注意的是,所有的TGS reads输入文件,只能是fasta格式,fastq会导致程序崩溃。
GitHub官网:TGS-GapCloser
一、conda安装
#conda创建并激活新环境
conda create -n tgsgapcloser | conda acitvate tgsgapcloser #先安装依赖包minimap2
conda install -c bioconda minimap2#安装tgsgapcloser
conda install -c bioconda tgsgapcloser#测试,显示manual即为安装成功
tgsgapcloser -h二、查看使用说明
以下参数说明相当清楚,看Example时有不懂可以参考下表。需要注意的是,默认是使用ONT reads,若是使用PacBio reads,需要使用--tgstype pb显式声明。
INFO : Run tgsgapcloser from /home/liuxin/miniconda3/envs/tgsgapcloser/bin ;
Version : 1.1.1 ;
Release time : 2019-12-31 .
INFO : Parsing args starting ...
Usage:
tgsgapcloser --scaff SCAFF_FILE --reads TGS_READS_FILE --output OUT_PREFIX [options...]
required :
--scaff <scaffold_file> the input scaffold file.
--reads <tgs_reads_file> the input TGS reads file.
--output <output_prefix> the output prefix.
part required :
--ne do not error correct. error correct by default.
or
--racon <racon> the installed racon.
or
--ngs <ngs_reads> the ngs reads used for pilon
--pilon <pilon> the installed pilon.
--samtools <samtools> the installed samtools.
--java <java> the installed java.
optional:
--tgstype <pb/ont> TGS type . ont by default.
--min_idy <min_idy> min_idy for filter reads .
0.3 for ont by default.
0.2 for pb by default.
--min_match <min_idy> min match length for filter reads .
300bp for ont by default.
200bp for pb by default.
--thread <t_num> thread uesd . 16 by default.
--chunk <chunk_num> split candidate into chunks to error-correct separately.
--pilon_mem <t_num> memory used for pilon , 300G for default.
--p_round <pilon_round> pilon error-correction round num . 3 by default.
--r_round <racon_round> racon error-correction round num . 1 by default.
--g_check gapsize diff check , none by default.三、Example
#an example of pre-corrected reads without error correction
tgsgapcloser \
--scaff <scaffold.fasta> \
--reads <tgs.reads.fasta> \
--output <test_racon> \
--ne \
>pipe.log 2>pipe.err#an example of raw ONT reads with error correction using long reads only
tgsgapcloser \
--scaff <scaffold.fasta> \
--reads <tgs.reads.fasta> \
--output test_pilon \
--racon /home/liuxin/miniconda3/envs/tgsgapcloser/bin \
>pipe.log 2>pipe.err#an example of raw ONT reads with error correction using NGS reads
tgsgapcloser \
--scaff <scaffold.fasta> \
--reads <tgs.reads.fasta> \
--output test_pilon \
--pilon /home/liuxin/miniconda3/envs/tgsgapcloser/bin/pilon-1.23.jar \
--ngs <ngs.reads.fastq.gz> \
--samtools /usr/local/bin/samtools \
--java /usr/bin/java \
>pipe.log 2>pipe.err四、实战
#策略一:gap filling using hifi reads
tgsgapcloser \
--scaff Cbp.LG.fasta \
--reads cbp.ccs.fasta \
--output hifi_fill_outfile \
--minmap_arg '-x asm20' \
--racon /home/liuxin/miniconda3/envs/tgsgapcloser/bin/racon \
--tgstype pb \
>pipe.log 2>pipe.err#策略二:gap filling using raw ont reads
tgsgapcloser --scaff Cbp.LG.fasta --reads ont.pass.fasta --output test_ne --ne > ont_fill.log 2>ont_fill.err#运行成功查看log文件会显示如下内容
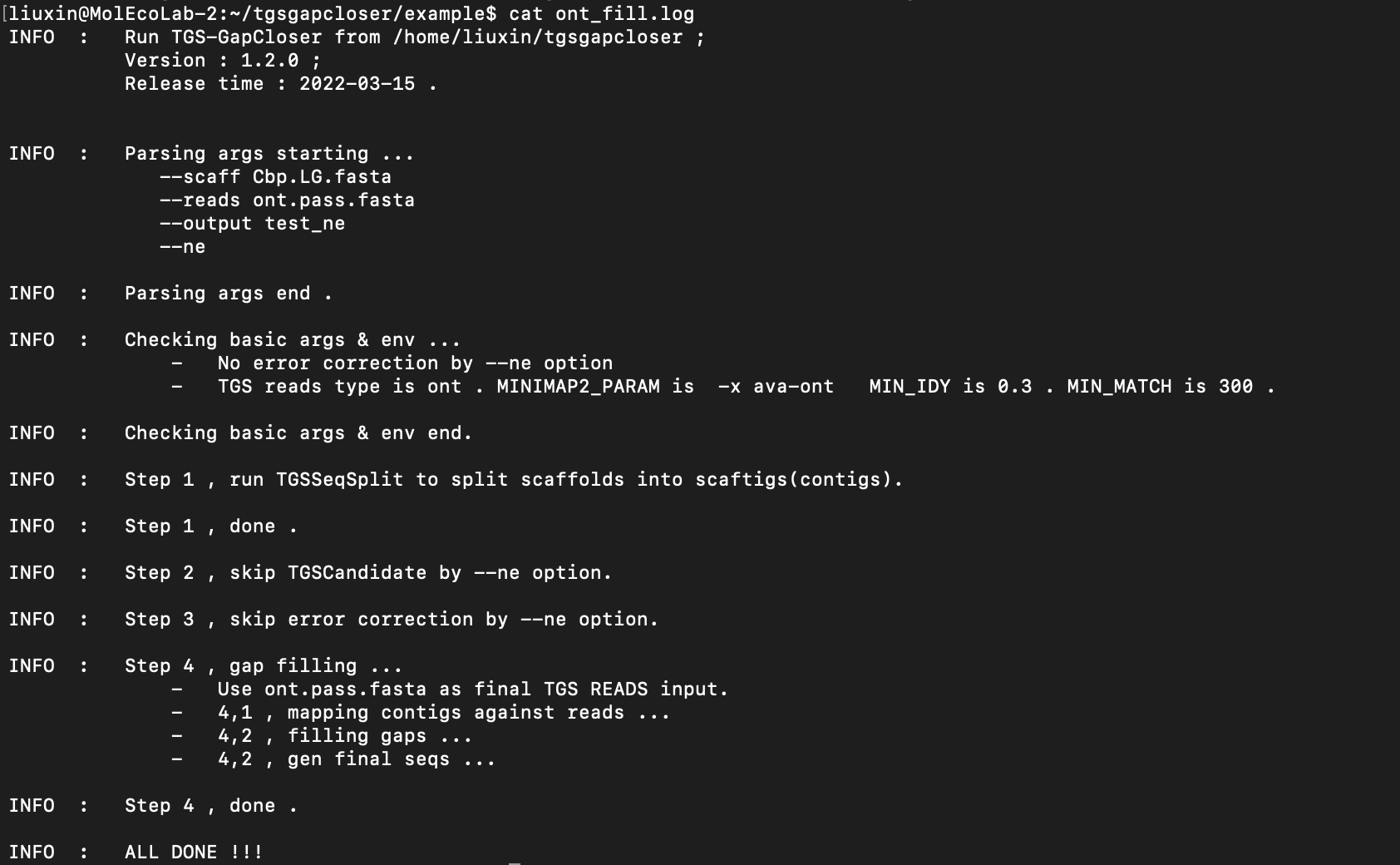
#主要关注两个结果文件
#统计信息
test_ne.gap_fill_detail
#gap_filled结果文件
test_ne.scaff_seqs五、总结优化
#输出结果与Github显示结果文件类型不一致,查看几个输出结果也没有找到gap fill后的fasta。因此,建议该换使用git clone下载及配置。
#克隆安装包
git clone https://github.com/BGI-Qingdao/TGS-GapCloser.git YOUR-INSTALL-DIR#minimap2的配置参照官网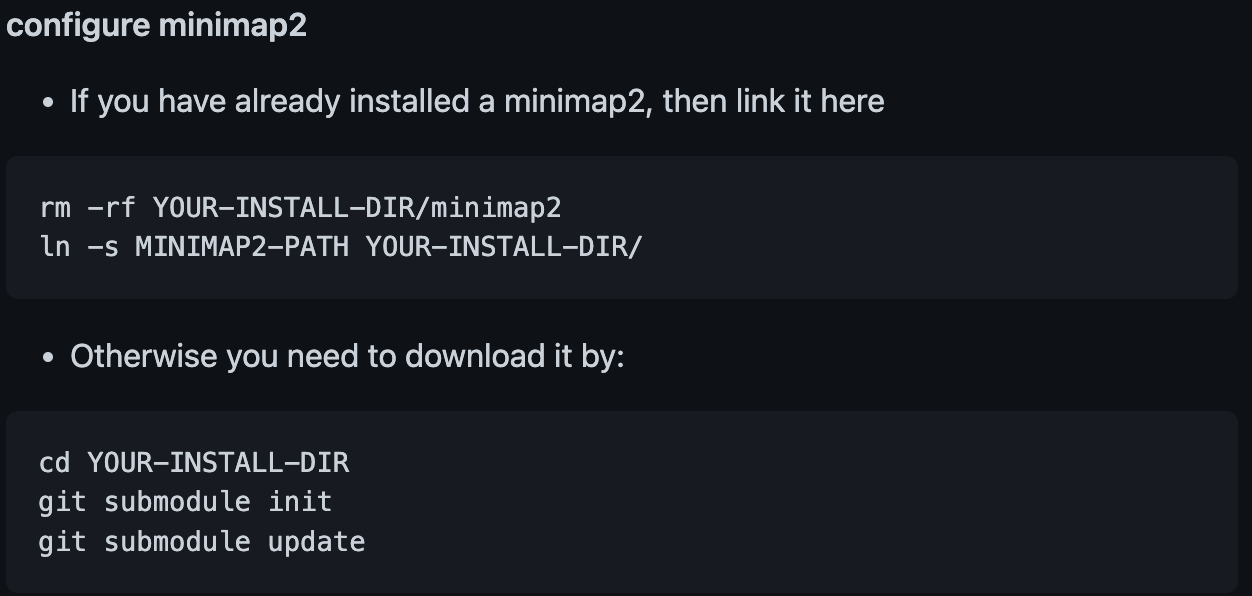
#建议移除包带的minimap2,为已有的minimap2创建软链接
rm -rf /home/liuxin/tgsgapcloser/minimap2
conda install -c bioconda minimap2
ln -s /home/liuxin/miniconda3/bin/minimap2 /home/liuxin/tgsgapcloser/minimap2/#进入安装目录,完成编译
cd YOUR-INSTALL-DIR
make#此处进入到准备好数据的工作目录下,使用绝对路径运行TGS-GapCloser.sh
nohup /home/liuxin/tgsgapcloser/TGS-GapCloser.sh --scaff Cbp.LG.fasta --reads ont.pass.fasta --output test_ne --ne --tgstype pb > pipe.log 2> pipe.err &#输出结果应该包含如下两个文件
test_ne.scaff_seq #this is the final assembly after gap filling
test_ne.gap_fill_details #details about how the final assembly was assemblied#.gap_fill_details文件格式如下
>scaffold_1
1 1000 S 1000 2000
1001 1010 N
1011 1100 S 2201 2290
1101 1110 F
1111 1200 S 2301 2390
>scaffold_2
......#关于.gap_fill_details的格式说明如下,其中第一列起始位置,第二列结束位置,第三列是type信息,F表示该条序列是filled后产生的;N表示存在Gap;S表示经过补洞后,某些序列位置信息存在改变,后序两列表明原始的位置。
参考文献
Mengyang Xu, Lidong Guo, Shengqiang Gu, Ou Wang, Rui Zhang, Brock A Peters, Guangyi Fan, Xin Liu, Xun Xu, Li Deng, Yongwei Zhang, TGS-GapCloser: A fast and accurate gap closer for large genomes with low coverage of error-prone long reads, GigaScience, Volume 9, Issue 9, September 2020, giaa094, https://doi.org/10.1093/gigascience/giaa094


