Linux 中 使用awk数组根据基因的PAV矩阵计算基因的存在频率
001、测试数据
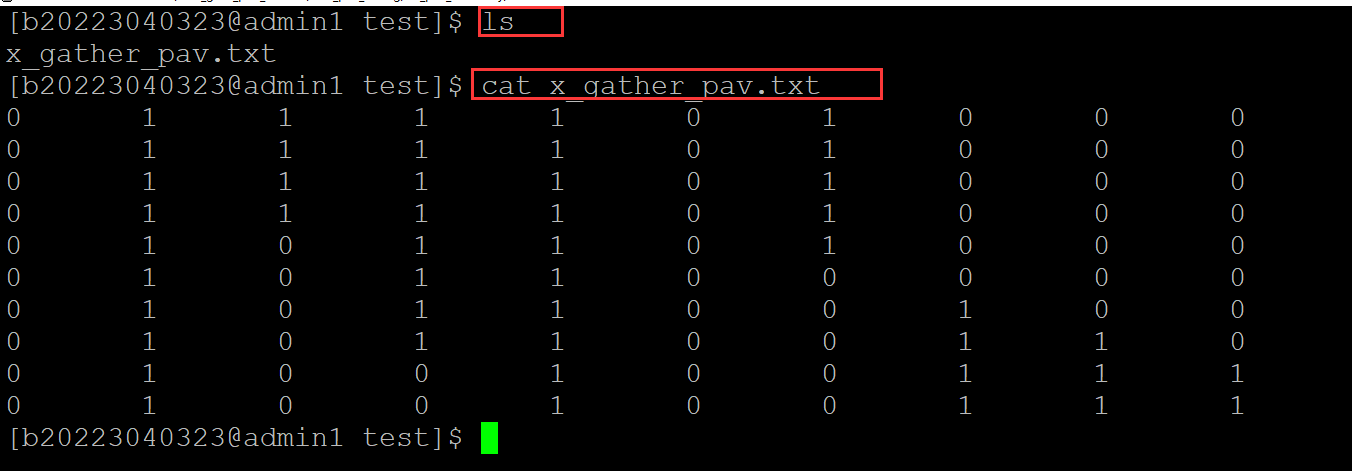
[b20223040323@admin1 test]$ ls x_gather_pav.txt [b20223040323@admin1 test]$ cat x_gather_pav.txt ## 测试数据;每一行是一个个体;每一列是一个基因;矩阵中的0表示基因在这个个体中缺失, 1表示基因在这个个体中存在 0 1 1 1 1 0 1 0 0 0 ## 计算基因存在频率的出发点就是基因在群体中存在的个体数除以样本总数,基于awk数组可以显著提高运算速度! 0 1 1 1 1 0 1 0 0 0 0 1 1 1 1 0 1 0 0 0 0 1 1 1 1 0 1 0 0 0 0 1 0 1 1 0 1 0 0 0 0 1 0 1 1 0 0 0 0 0 0 1 0 1 1 0 0 1 0 0 0 1 0 1 1 0 0 1 1 0 0 1 0 0 1 0 0 1 1 1 0 1 0 0 1 0 0 1 1 1
002、计算如下:
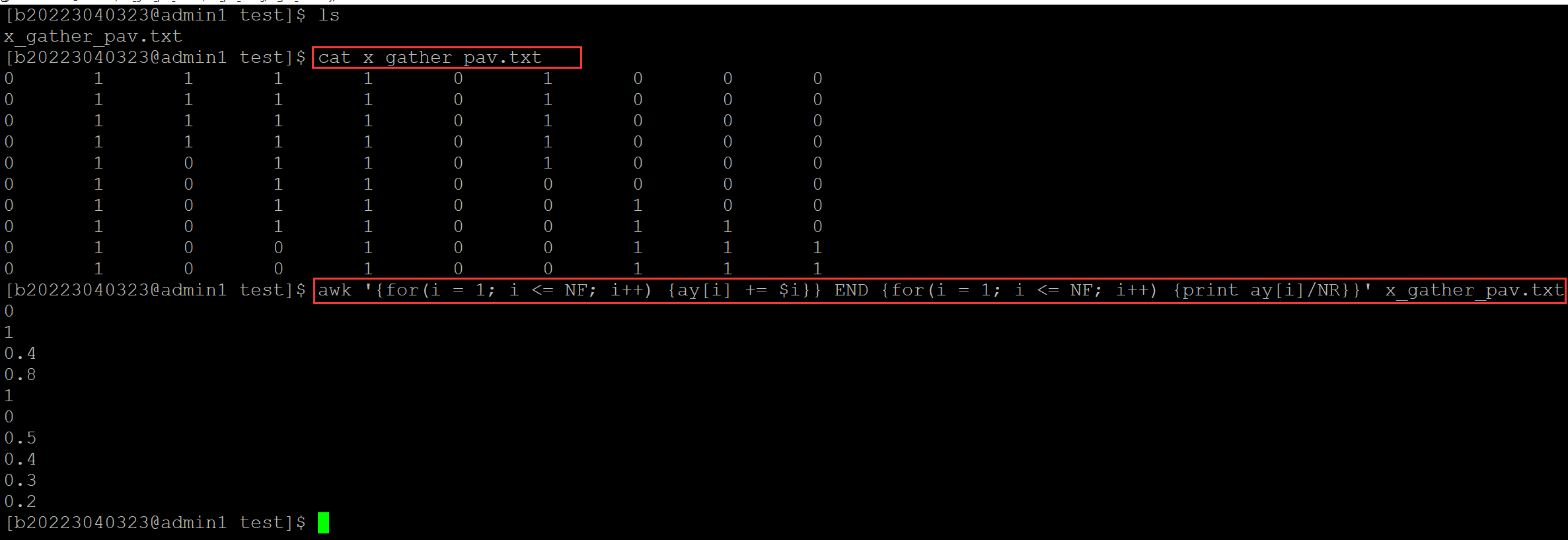
[b20223040323@admin1 test]$ ls x_gather_pav.txt [b20223040323@admin1 test]$ cat x_gather_pav.txt 0 1 1 1 1 0 1 0 0 0 0 1 1 1 1 0 1 0 0 0 0 1 1 1 1 0 1 0 0 0 0 1 1 1 1 0 1 0 0 0 0 1 0 1 1 0 1 0 0 0 0 1 0 1 1 0 0 0 0 0 0 1 0 1 1 0 0 1 0 0 0 1 0 1 1 0 0 1 1 0 0 1 0 0 1 0 0 1 1 1 0 1 0 0 1 0 0 1 1 1 ## awk的列迭代将基因计数累加与ay数组以列为索引的元素中,行迭代实现不同个体的累加;最终对数据元素进行迭代,并除以awk内置变量NR(样本数) [b20223040323@admin1 test]$ awk '{for(i = 1; i <= NF; i++) {ay[i] += $i}} END {for(i = 1; i <= NF; i++) {print ay[i]/NR}}' x_gather_pav.txt ## 统计结果如下 0 1 0.4 0.8 1 0 0.5 0.4 0.3 0.2
。






【推荐】国内首个AI IDE,深度理解中文开发场景,立即下载体验Trae
【推荐】编程新体验,更懂你的AI,立即体验豆包MarsCode编程助手
【推荐】抖音旗下AI助手豆包,你的智能百科全书,全免费不限次数
【推荐】轻量又高性能的 SSH 工具 IShell:AI 加持,快人一步
· 震惊!C++程序真的从main开始吗?99%的程序员都答错了
· 【硬核科普】Trae如何「偷看」你的代码?零基础破解AI编程运行原理
· 单元测试从入门到精通
· 上周热点回顾(3.3-3.9)
· winform 绘制太阳,地球,月球 运作规律