linux shell实现计算SNP在指定群体的ROH片段中出现的频率
a、shell实现
001、ROH 检测
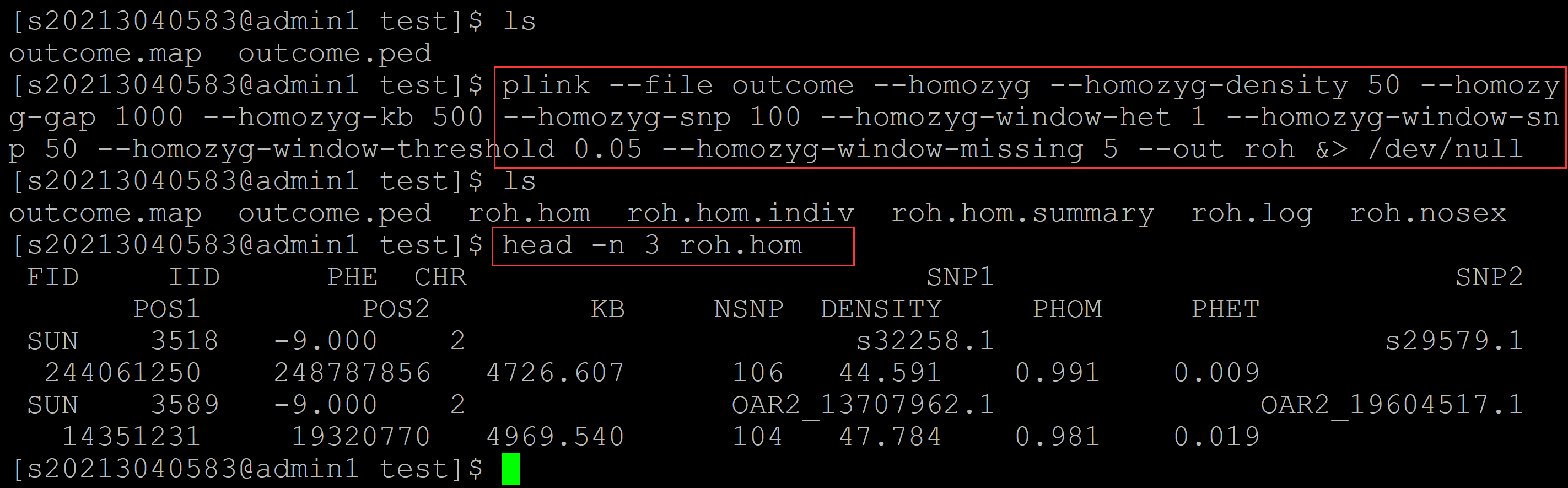
[s20213040583@admin1 test]$ ls ## 测试文件 outcome.map outcome.ped ## plink软件检测 [s20213040583@admin1 test]$ plink --file outcome --homozyg --homozyg-density 50 --homozyg-gap 1000 --homozyg-kb 500 --homozyg-snp 100 --homozyg-window-het 1 --homozyg-window-snp 50 --homozyg-window-threshold 0.05 --homozyg-window-missing 5 --out roh &> /dev/null [s20213040583@admin1 test]$ ls outcome.map outcome.ped roh.hom roh.hom.indiv roh.hom.summary roh.log roh.nosex [s20213040583@admin1 test]$ head -n 3 roh.hom ## 查看前几行 FID IID PHE CHR SNP1 SNP2 POS1 POS2 KB NSNP DENSITY PHOM PHET SUN 3518 -9.000 2 s32258.1 s29579.1 244061250 248787856 4726.607 106 44.591 0.991 0.009 SUN 3589 -9.000 2 OAR2_13707962.1 OAR2_19604517.1 14351231 19320770 4969.540 104 47.784 0.981 0.019
02、输出出现在ROH中的染色体和SNP的ID
## 统计出现在ROH的SNP
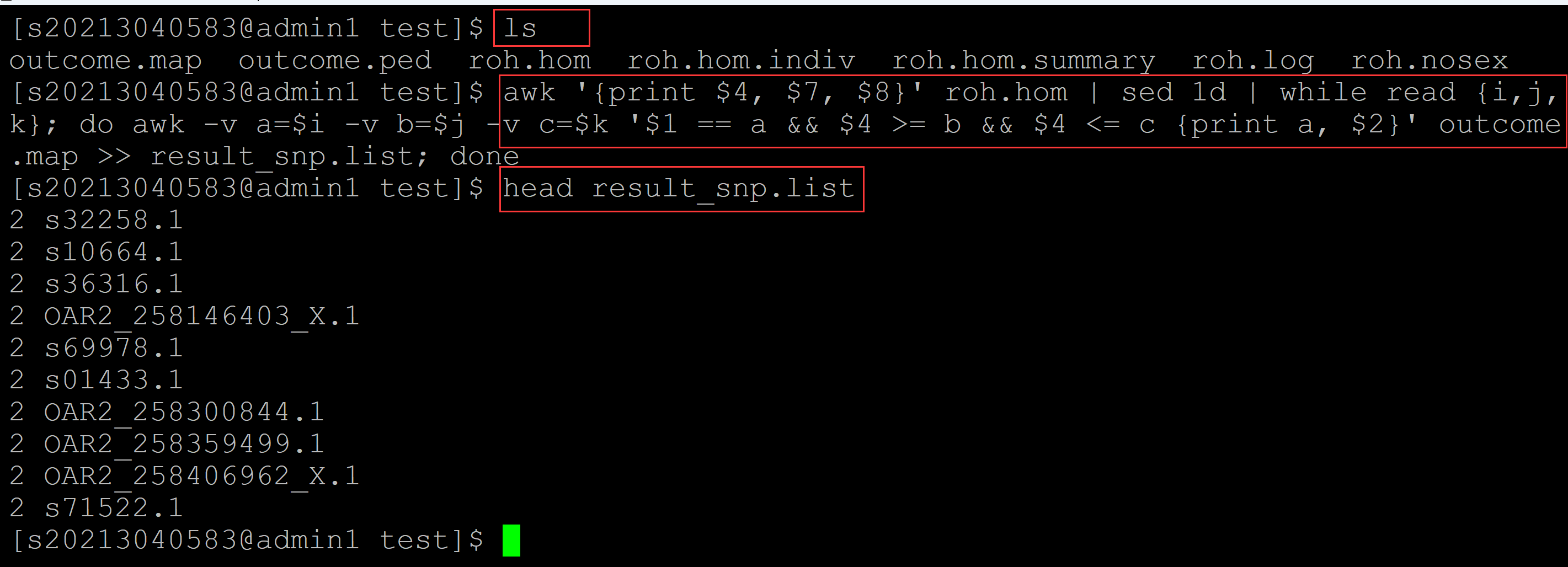
[s20213040583@admin1 test]$ awk '{print $4, $7, $8}' roh.hom | sed 1d | while read {i,j,k}; do awk -v a=$i -v b=$j -v c=$k '$1 == a && $4 >= b && $4 <= c {print a, $2}' outcome.map >> result_snp.list; done [s20213040583@admin1 test]$ head result_snp.list 2 s32258.1 2 s10664.1 2 s36316.1 2 OAR2_258146403_X.1 2 s69978.1 2 s01433.1 2 OAR2_258300844.1 2 OAR2_258359499.1 2 OAR2_258406962_X.1 2 s71522.1
03、统计出现的频率
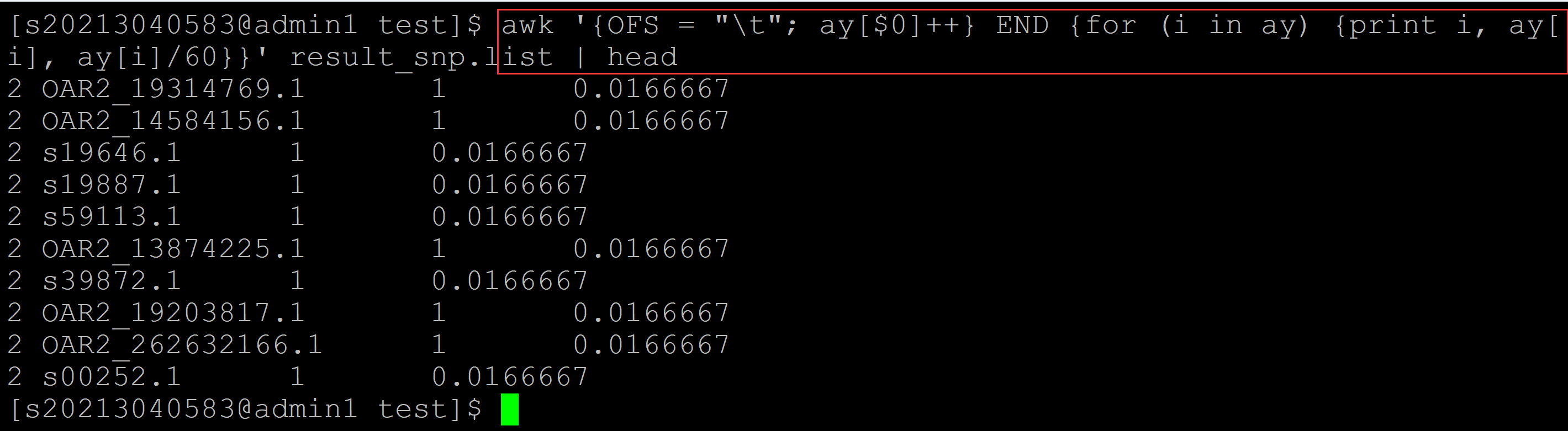
[s20213040583@admin1 test]$ awk '{OFS = "\t"; ay[$0]++} END {for (i in ay) {print i, ay[i], ay[i]/60}}' result_snp.list | head 2 OAR2_19314769.1 1 0.0166667 ## 第一列染色体; 第二列SNP; 第三列次数; 第四列概率; 60表示的样本数目 2 OAR2_14584156.1 1 0.0166667 2 s19646.1 1 0.0166667 2 s19887.1 1 0.0166667 2 s59113.1 1 0.0166667 2 OAR2_13874225.1 1 0.0166667 2 s39872.1 1 0.0166667 2 OAR2_19203817.1 1 0.0166667 2 OAR2_262632166.1 1 0.0166667 2 s00252.1 1 0.0166667
。
b、python实现
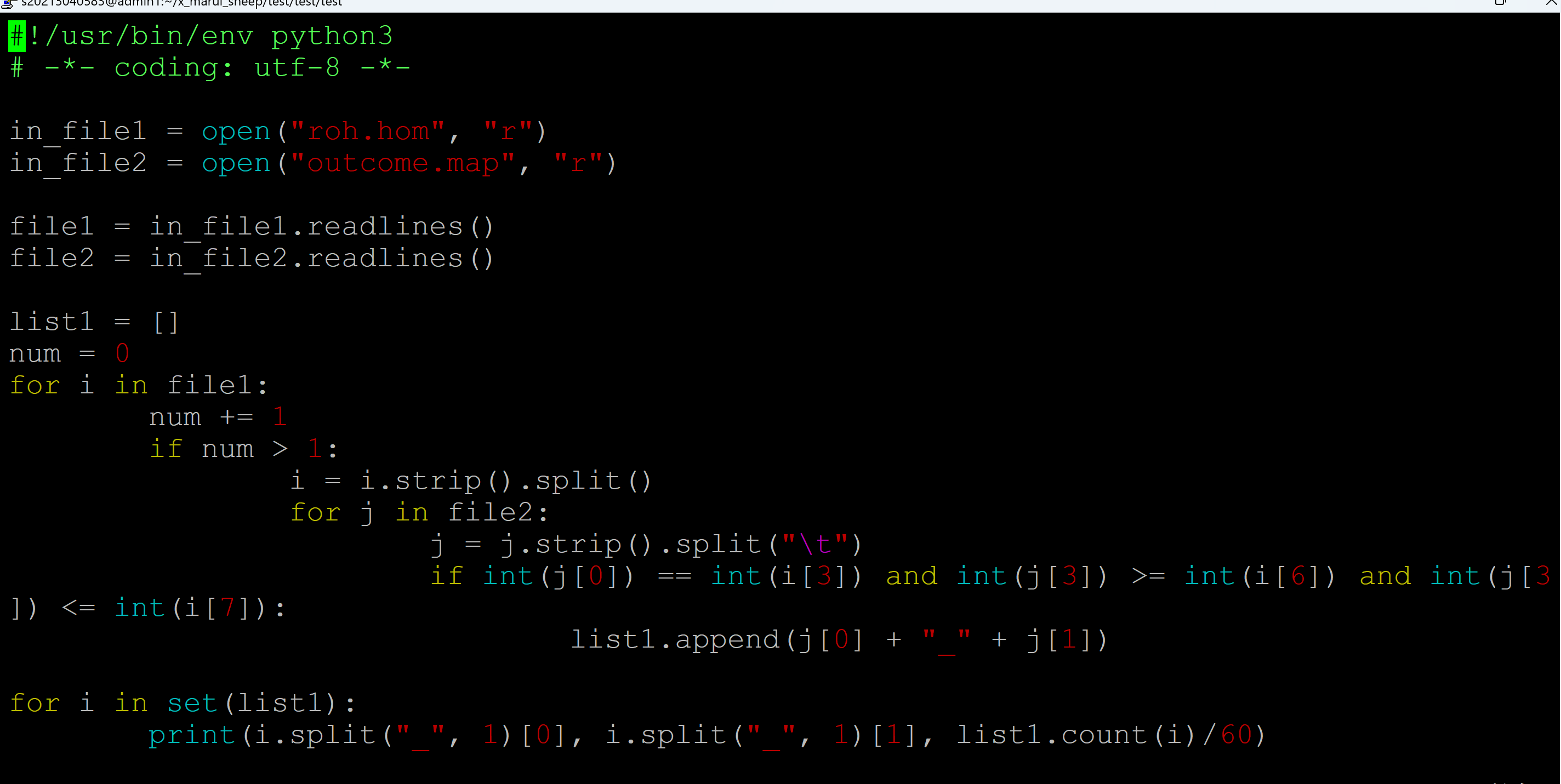
(base) [s20213040583@admin1 test]$ cat test.py ## 计算程序 #!/usr/bin/env python3 # -*- coding: utf-8 -*- in_file1 = open("roh.hom", "r") in_file2 = open("outcome.map", "r") file1 = in_file1.readlines() file2 = in_file2.readlines() list1 = [] num = 0 for i in file1: num += 1 if num > 1: i = i.strip().split() for j in file2: j = j.strip().split("\t") if int(j[0]) == int(i[3]) and int(j[3]) >= int(i[6]) and int(j[3]) <= int(i[7]): list1.append(j[0] + "_" + j[1]) for i in set(list1): print(i.split("_", 1)[0], i.split("_", 1)[1], list1.count(i)/60) ## 60为样本数目 in_file1.close() in_file2.close()
(base) [s20213040583@admin1 test]$ python3 test.py | head ## 测试效果, 查看前几行 2 OAR2_15721601.1 0.016666666666666666 ## 第一列为染色体; 第二列为SNP;第三列为频率 2 OAR2_15830990.1 0.016666666666666666 2 s10664.1 0.016666666666666666 2 OAR2_19100124.1 0.016666666666666666 2 OAR2_17890079.1 0.016666666666666666 2 OAR2_260323417.1 0.016666666666666666 2 OAR2_19203817.1 0.016666666666666666 2 s31977.1 0.016666666666666666 2 s04563.1 0.016666666666666666 2 s29579.1 0.016666666666666666
。
分类:
生信






【推荐】国内首个AI IDE,深度理解中文开发场景,立即下载体验Trae
【推荐】编程新体验,更懂你的AI,立即体验豆包MarsCode编程助手
【推荐】抖音旗下AI助手豆包,你的智能百科全书,全免费不限次数
【推荐】轻量又高性能的 SSH 工具 IShell:AI 加持,快人一步
· 震惊!C++程序真的从main开始吗?99%的程序员都答错了
· 【硬核科普】Trae如何「偷看」你的代码?零基础破解AI编程运行原理
· 单元测试从入门到精通
· 上周热点回顾(3.3-3.9)
· winform 绘制太阳,地球,月球 运作规律