二代测序分析流程
001、
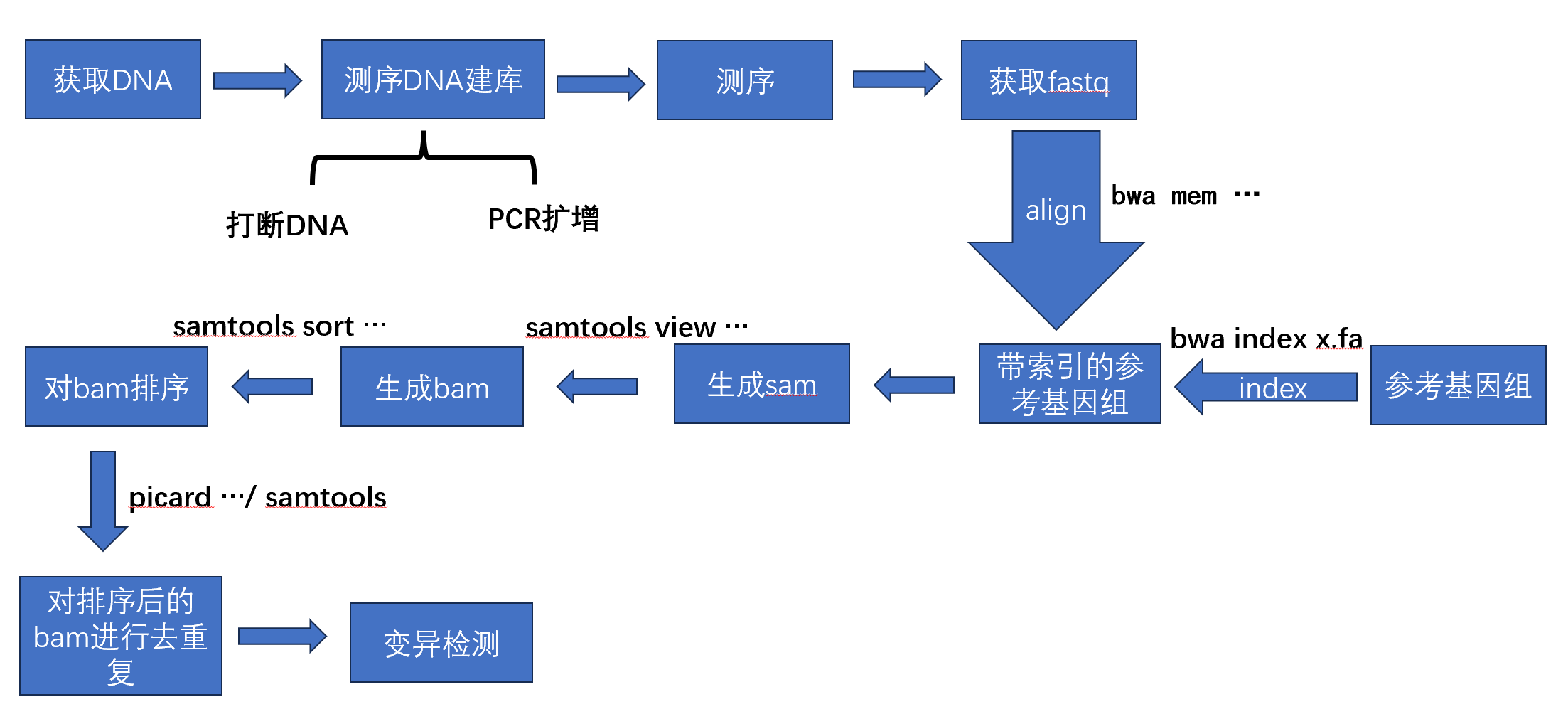
01、bwa index x.fa:
对参考基因组构建索引,这其实是在为参考序列进行Burrows Wheeler变换(wiki: 块排序压缩),以便能够在序列比对的时候进行快速的搜索和定位。
02、samtools sort:
因为测序的reads是无序的,比对生成的sam/bam文件也是无序的。
03、去重复
重复DNA片段的来源:建库过程中PCR的扩增
重复DNA片段的影响:对结果造成偏差,造成假阳性和假阴性。
1、DNA片段打断的过程中,会发生转换或者颠换,PCR反应会放大这种错误(假阳性)
2、PCR反应过程自身也会产生错误(假阳性)
3、PCR bias:某些DNA片段PCR反应强烈,如果含有reference序列的片段反应强烈,则会掩盖掉真实的变异(假阴性)
去重复的原理:PCR扩增的片段都是一样的,那么比对到参考基因组的位置也是一样的(原始的DNA片段,长度完全一样概率低),可以根据这个特点找到这些重复序列了。
04、
参考:
01、https://www.jianshu.com/p/8cdbb89530c6?utm_campaign=maleskine&utm_content=note&utm_medium=seo_notes





【推荐】国内首个AI IDE,深度理解中文开发场景,立即下载体验Trae
【推荐】编程新体验,更懂你的AI,立即体验豆包MarsCode编程助手
【推荐】抖音旗下AI助手豆包,你的智能百科全书,全免费不限次数
【推荐】轻量又高性能的 SSH 工具 IShell:AI 加持,快人一步
· 震惊!C++程序真的从main开始吗?99%的程序员都答错了
· 【硬核科普】Trae如何「偷看」你的代码?零基础破解AI编程运行原理
· 单元测试从入门到精通
· 上周热点回顾(3.3-3.9)
· winform 绘制太阳,地球,月球 运作规律
2022-09-22 linux 中统计相同序列出现的次数
2022-09-22 sbatch命令在集群递交任务模板
2022-09-22 linux 中 利用命令向文件的末尾添加空行
2022-09-22 linux中basename命令
2022-09-22 linux 中 date +%s 获取1970年以来的秒数