blast软件比对库nr、nt库的下载、构建索引、序列比对
001、

002、
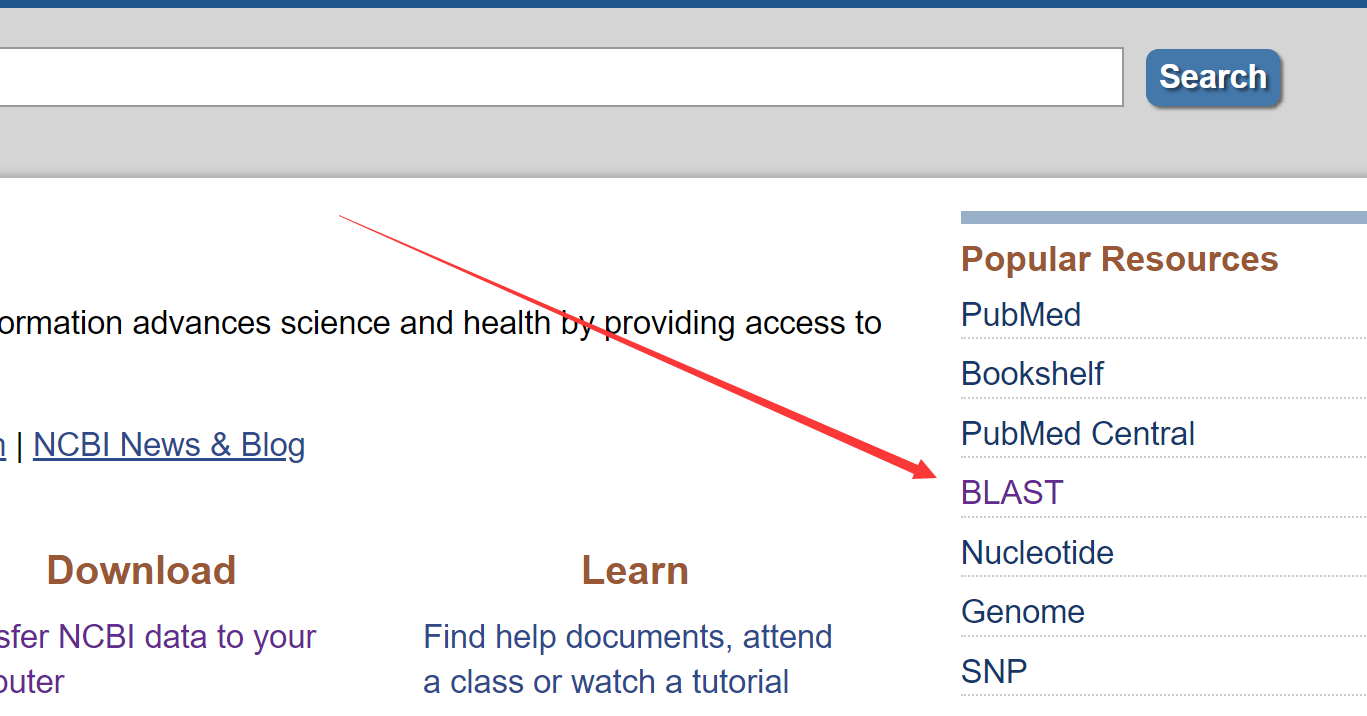
003、
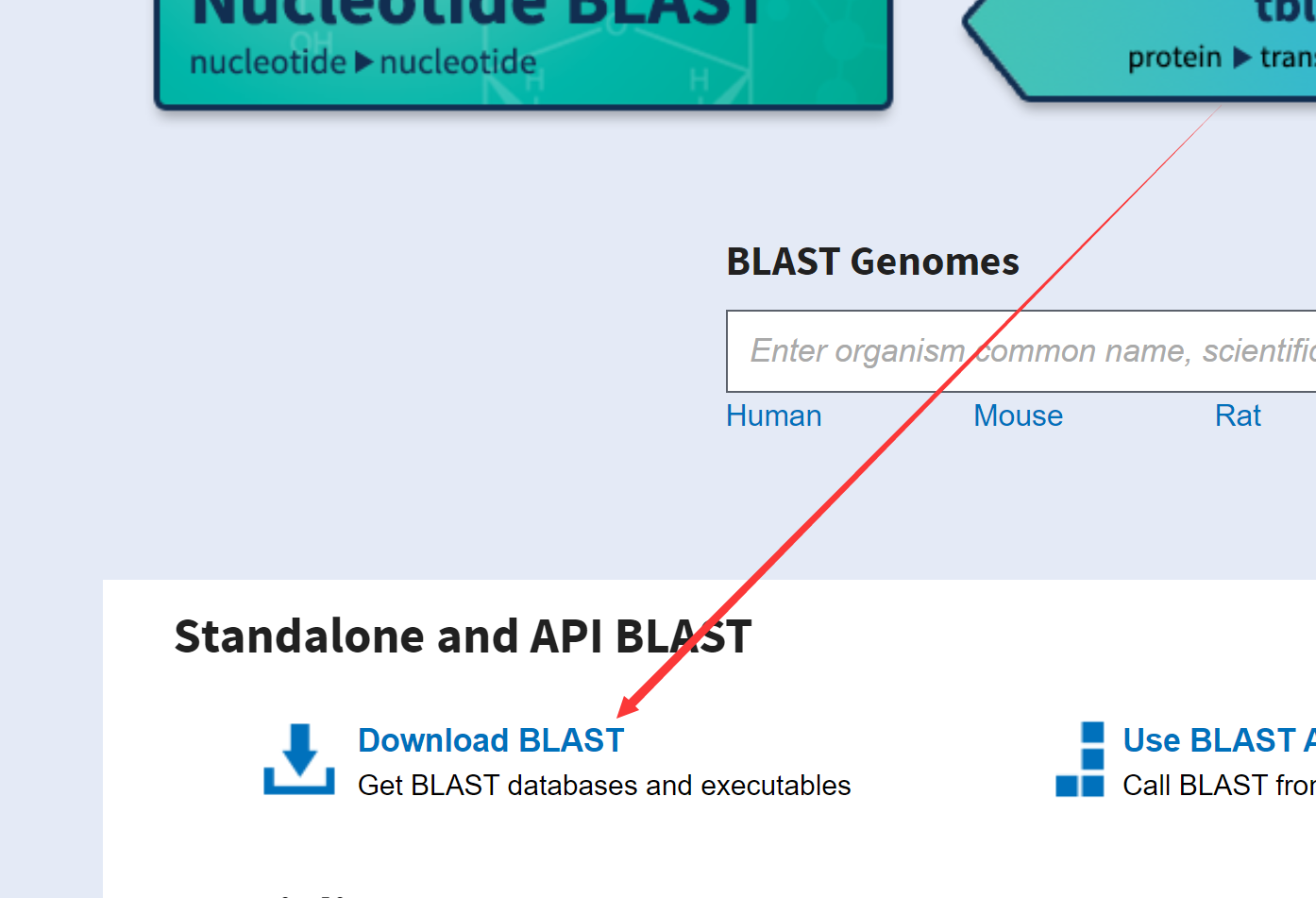
004、

005、

006、
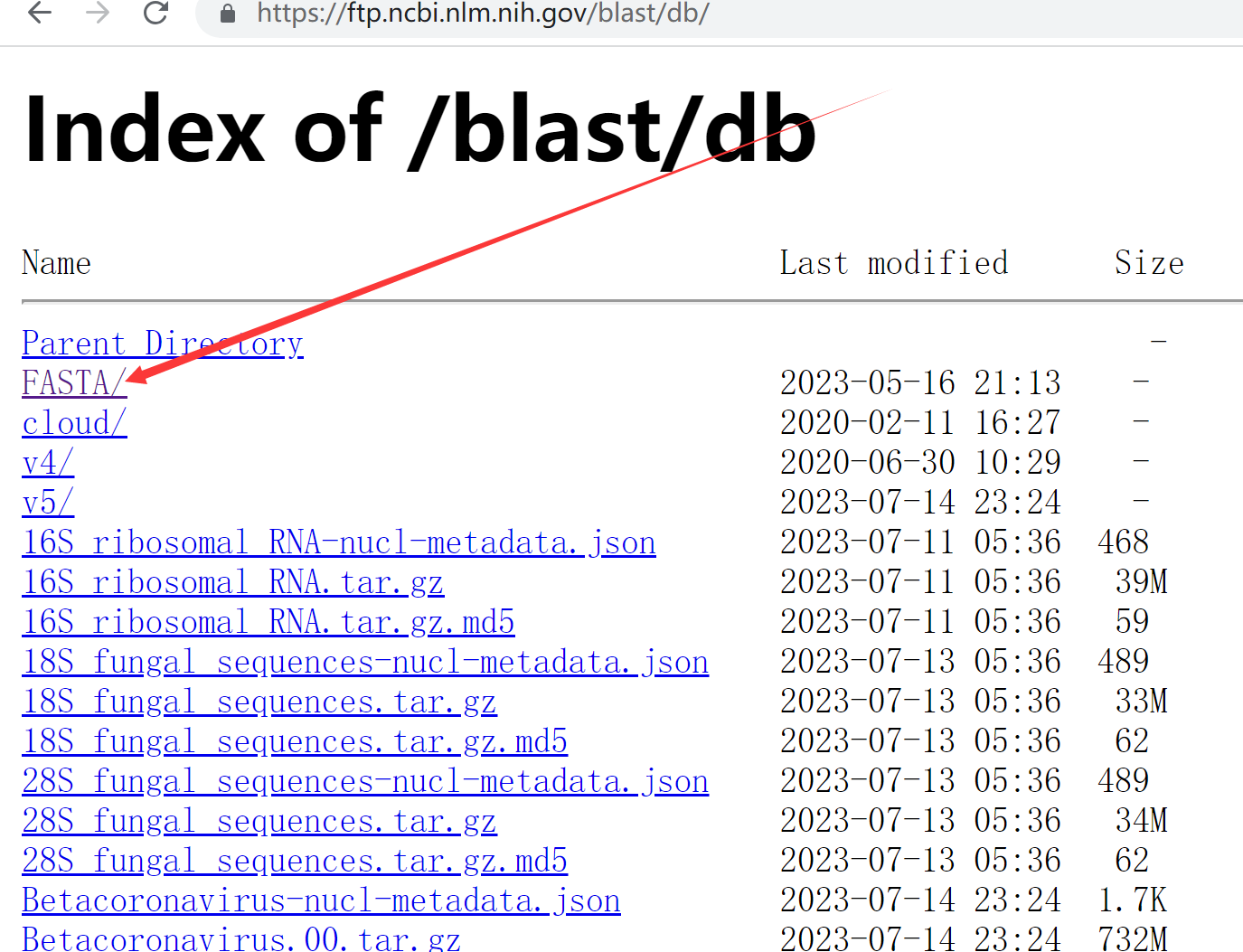
007、
链接:https://ftp.ncbi.nlm.nih.gov/blast/db/FASTA/

。
nt为核酸数据库,nr为蛋白质数据库
008、使用命令行下载
a、
[root@PC1 test02]# wget -c https://ftp.ncbi.nlm.nih.gov/blast/db/FASTA/nr.gz [root@PC1 test02]# wget -c https://ftp.ncbi.nlm.nih.gov/blast/db/FASTA/nt.gz
b、解压
[root@PC1 test02]# gunzip nr.gz
[root@PC1 test02]# gunzip nt.gz
c、创建nr/nt数据库目录
[root@PC1 test02]# mkdir nr_db nt_db
d、构建nr/nt数据库
[root@PC1 test02]# makeblastdb -in nr -dbtype prot -title my_nr -parse_seqids -out ./nr_db/nr -logfile make_nr.log [root@PC1 test02]# makeblastdb -in nt -dbtype nucl -title my_nt -parse_seqids -out ./nt_db/nt -logfile make_nt.log
008、测试fastq文件的下载(用于生成进行比对的fasta格式数据)
wget -c https://sra-downloadb.be-md.ncbi.nlm.nih.gov/sos2/sra-pub-zq-18/ERR007/7959/ERR7959948/ERR7959948.lite.1
fastq-dump --split-files ERR7959948.lite.1
009、生成进行比对的测试的fasta格式数据
cat ERR7959948.lite.1_1.fastq | head -n 20000 | awk '{if(NR%4==1){print ">"$1}else if(NR%4==2){print $0}}'|sed 's/@//g' > myfile.fa
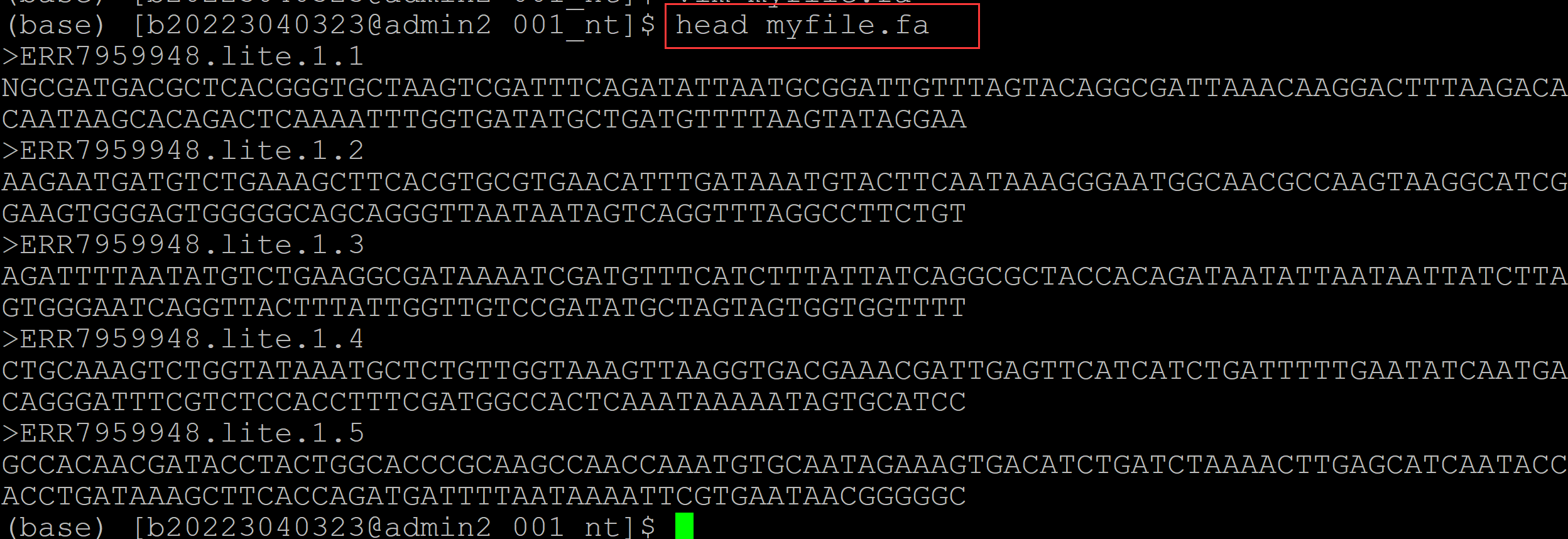
010、比对命令
blastn -num_threads 32 -max_target_seqs 10 -evalue 1e-05 -db /xxx/nt -outfmt "7 qseqid sseqid evalue pident ppos length mismatch gapopen qstart qend sstart send bitscore staxid sscinames stitle" -query ./xxx.fa -out ./xxx.blastn.out
参考:
001、https://www.jianshu.com/p/c4b6723fd786
002、https://blog.csdn.net/qq_42962326/article/details/105081327
003、





【推荐】国内首个AI IDE,深度理解中文开发场景,立即下载体验Trae
【推荐】编程新体验,更懂你的AI,立即体验豆包MarsCode编程助手
【推荐】抖音旗下AI助手豆包,你的智能百科全书,全免费不限次数
【推荐】轻量又高性能的 SSH 工具 IShell:AI 加持,快人一步
· 震惊!C++程序真的从main开始吗?99%的程序员都答错了
· 【硬核科普】Trae如何「偷看」你的代码?零基础破解AI编程运行原理
· 单元测试从入门到精通
· 上周热点回顾(3.3-3.9)
· winform 绘制太阳,地球,月球 运作规律
2022-07-15 gemma 软件对数量性状 混合线性模型T值及P值的计算
2022-07-15 gemma 对 数量性状 一般线性模型关联分析计算 T值
2022-07-15 plink软件 --linear 对数量性状进行关联分析 SE计算
2022-07-15 plink 软件 --assoc 数量性状关联分析 T值的来源
2021-07-15 4-4
2021-07-15 4-2-4
2021-07-15 4-1-3