linux 中blast序列比对
001、对数据库构建索引
[root@PC1 001_protein_database]# ls protein.faa ## 构建索引 [root@PC1 001_protein_database]# makeblastdb -in protein.faa -dbtype prot -title xxx -parse_seqids -hash_index -out index -logfile log.txt [root@PC1 001_protein_database]# ls index.pdb index.phi index.pin index.pog index.pot index.ptf log.txt index.phd index.phr index.pjs index.pos index.psq index.pto protein.faa [root@PC1 001_protein_database]# cat log.txt Building a new DB, current time: 07/15/2023 20:05:09 New DB name: /home/blast_test/001_protein_database/index New DB title: xxx Sequence type: Protein Keep MBits: T Maximum file size: 3000000000B Adding sequences from FASTA; added 48265 sequences in 0.836226 seconds.
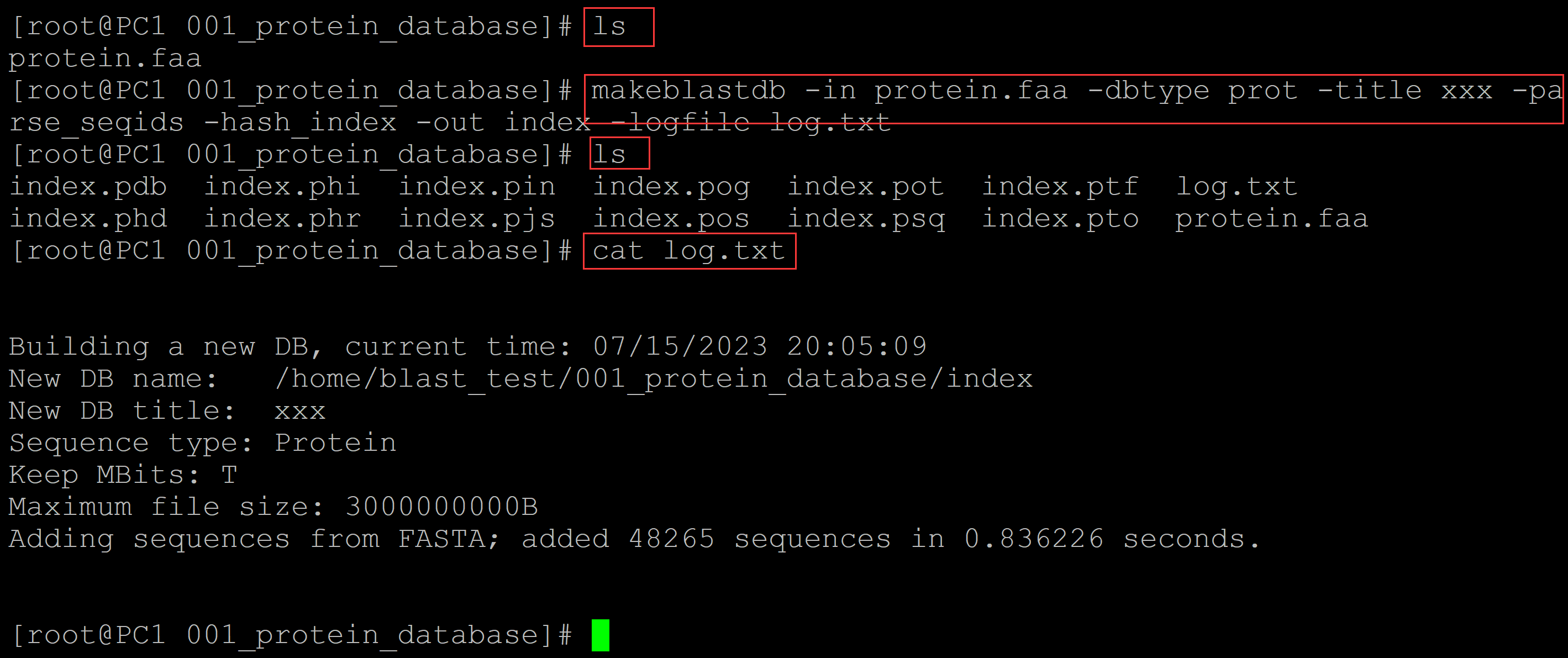
makeblastdb:构建索引的软件
-in protein.faa: 要构建索引的数据库(这里是拟南芥的蛋白质序列)
-dbtype : 指定数据类型,prot为蛋白质、nucl为核酸; 这里是蛋白质,因此选prot
-parse_seqids: 帮助我们解析fa文件中,“>”后面的id信息
-parse_seqids, -hash_index: 两个参数一般都带上,主要是为blastdbcmd取子序列时使用;
-title:给数据库起个名(不能用在后面搜索时-db的参数)
-out: 后接数据库名
-logfile:输出日志文件。
02、可以直接使用简洁命令:
makeblastdb -in input_file -dbtype prot/nucl -out index
002、balst软件的主要程序
01、blastn:核酸比对到核酸
02、blastp:蛋白质比对到蛋白质
03、blastx:核酸比对到蛋白质
04、tbalstn:蛋白质比对到核酸
05、tblastx: 核酸比对到核酸;(核酸首先翻译成蛋白质,然后进行比对)。
003、NCBI中的几个主要数据库
01、nt:核苷酸库
02、nr:蛋白质库
03、swissprot:蛋白质库。
。
分类:
生信






【推荐】国内首个AI IDE,深度理解中文开发场景,立即下载体验Trae
【推荐】编程新体验,更懂你的AI,立即体验豆包MarsCode编程助手
【推荐】抖音旗下AI助手豆包,你的智能百科全书,全免费不限次数
【推荐】轻量又高性能的 SSH 工具 IShell:AI 加持,快人一步
· 震惊!C++程序真的从main开始吗?99%的程序员都答错了
· 【硬核科普】Trae如何「偷看」你的代码?零基础破解AI编程运行原理
· 单元测试从入门到精通
· 上周热点回顾(3.3-3.9)
· winform 绘制太阳,地球,月球 运作规律
2022-07-15 gemma 软件对数量性状 混合线性模型T值及P值的计算
2022-07-15 gemma 对 数量性状 一般线性模型关联分析计算 T值
2022-07-15 plink软件 --linear 对数量性状进行关联分析 SE计算
2022-07-15 plink 软件 --assoc 数量性状关联分析 T值的来源
2021-07-15 4-4
2021-07-15 4-2-4
2021-07-15 4-1-3