linux 中shell脚本实现统计每一个read的长度
001、
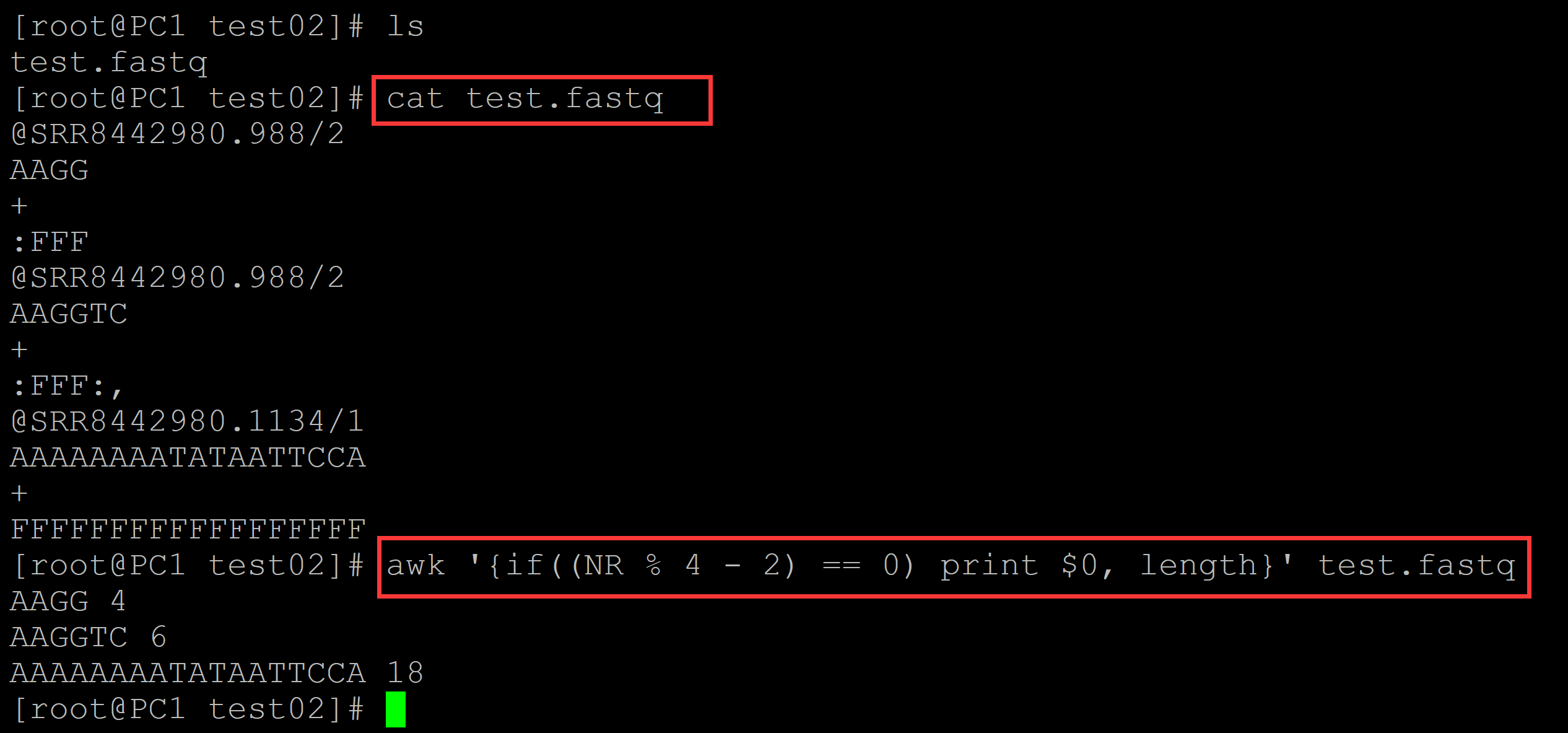
[root@PC1 test02]# ls test.fastq [root@PC1 test02]# cat test.fastq ## 测试fastq数据 @SRR8442980.988/2 AAGG + :FFF @SRR8442980.988/2 AAGGTC + :FFF:, @SRR8442980.1134/1 AAAAAAAATATAATTCCA + FFFFFFFFFFFFFFFFFF [root@PC1 test02]# awk '{if((NR % 4 - 2) == 0) print $0, length}' test.fastq ## 统计每一个read的长度 AAGG 4 AAGGTC 6 AAAAAAAATATAATTCCA 18
。
002、借助与Bio 中的 SeqIO模块统计
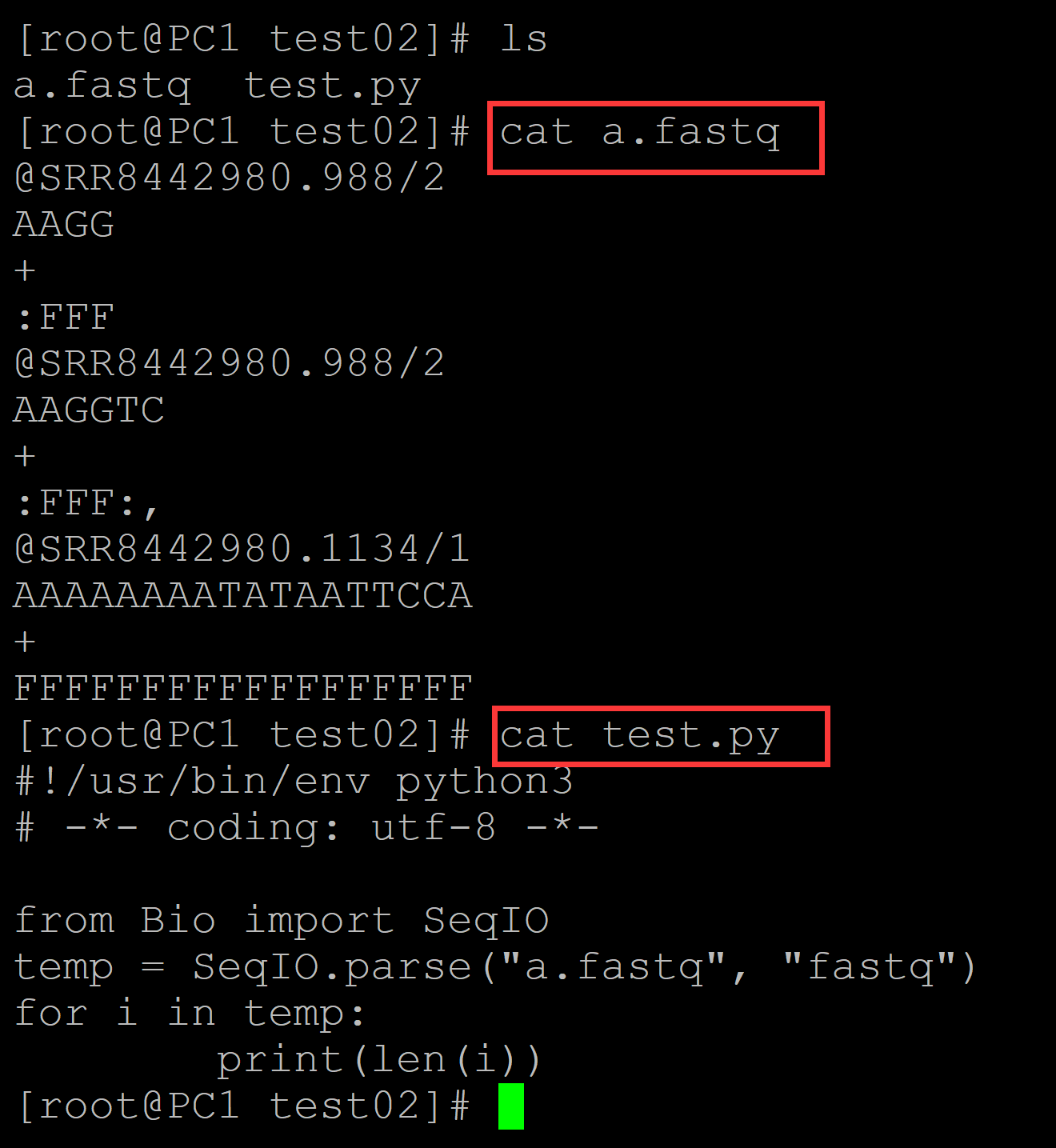
[root@PC1 test02]# ls a.fastq test.py [root@PC1 test02]# cat a.fastq ## 测试fastq文件 @SRR8442980.988/2 AAGG + :FFF @SRR8442980.988/2 AAGGTC + :FFF:, @SRR8442980.1134/1 AAAAAAAATATAATTCCA + FFFFFFFFFFFFFFFFFF [root@PC1 test02]# cat test.py ## 统计程序 #!/usr/bin/env python3 # -*- coding: utf-8 -*- from Bio import SeqIO temp = SeqIO.parse("a.fastq", "fastq") for i in temp: print(len(i))
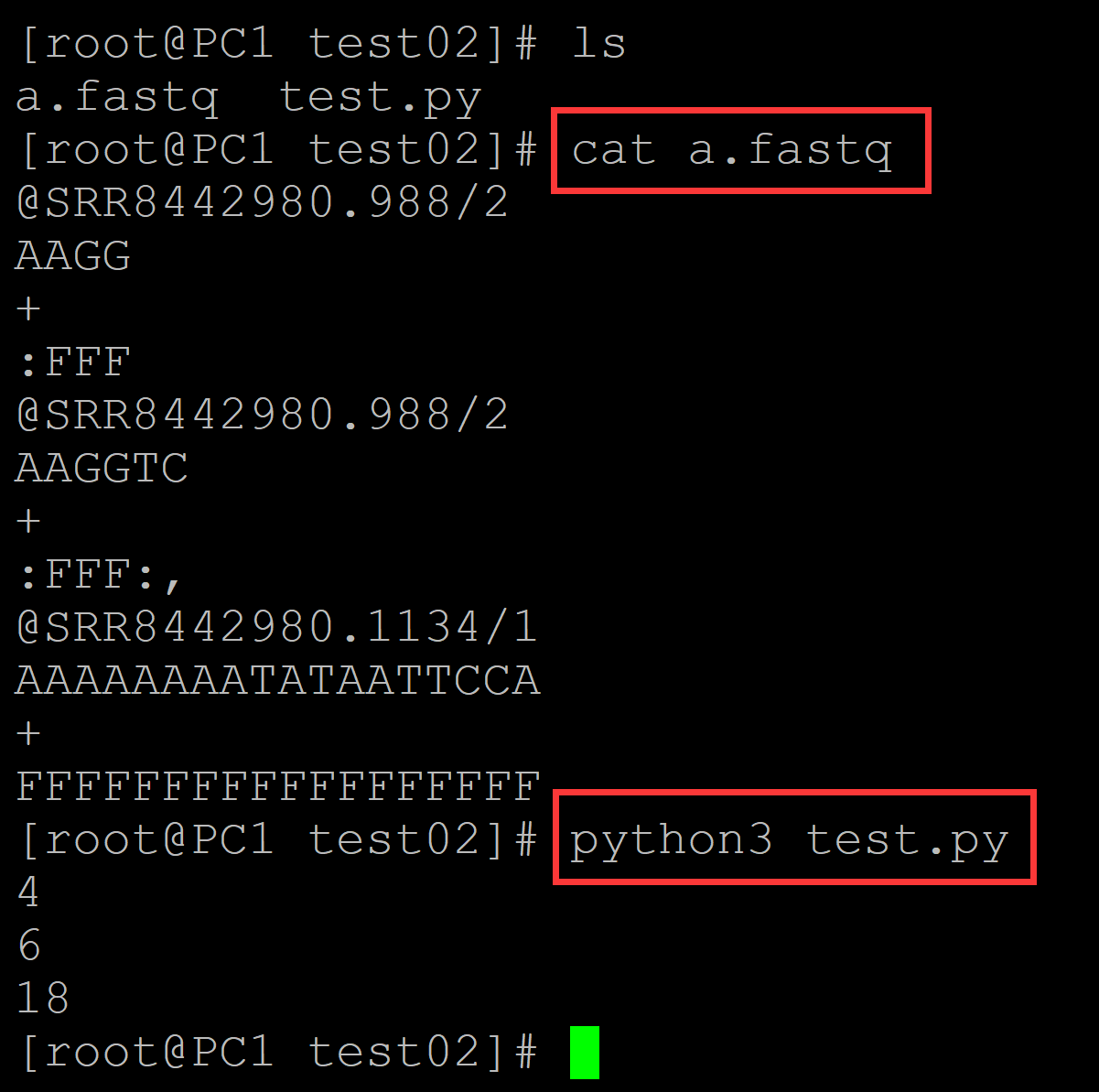
执行程序:
[root@PC1 test02]# ls a.fastq test.py [root@PC1 test02]# cat a.fastq @SRR8442980.988/2 AAGG + :FFF @SRR8442980.988/2 AAGGTC + :FFF:, @SRR8442980.1134/1 AAAAAAAATATAATTCCA + FFFFFFFFFFFFFFFFFF [root@PC1 test02]# python3 test.py 4 6 18
分类:
生信






【推荐】国内首个AI IDE,深度理解中文开发场景,立即下载体验Trae
【推荐】编程新体验,更懂你的AI,立即体验豆包MarsCode编程助手
【推荐】抖音旗下AI助手豆包,你的智能百科全书,全免费不限次数
【推荐】轻量又高性能的 SSH 工具 IShell:AI 加持,快人一步
· 震惊!C++程序真的从main开始吗?99%的程序员都答错了
· 【硬核科普】Trae如何「偷看」你的代码?零基础破解AI编程运行原理
· 单元测试从入门到精通
· 上周热点回顾(3.3-3.9)
· winform 绘制太阳,地球,月球 运作规律
2021-06-23 c语言中float关键字和double关键字的区别
2021-06-23 linux 系统如何给软件设置环境变量
2021-06-23 使用detectRUNS包进行ROH检测,计算近交系数实践
2021-06-23 gcta 计算 fst 实践
2021-06-23 PCA 主成分分析实践 gcta软件
2021-06-23 使用admixture软件分析群体组成
2021-06-23 PCA 主成分分析实践 plink软件