python 中 pyfaidx 模块统计fasta文件每一条染色体的长度
001、python版本 和 pip版本
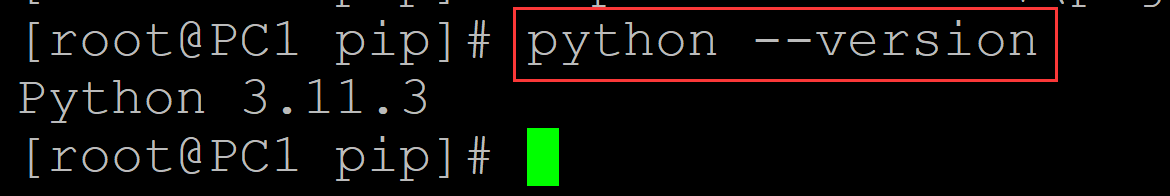
a、python版本
[root@PC1 pip]# python --version Python 3.11.3
b、pip版本
[root@PC1 Python-3.11.3]# pip --version pip 23.1.2 from /usr/local/lib/python3.11/site-packages/pip (python 3.11)

002、利用 pip安装 pyfaidx 模块
[root@PC1 Python-3.11.3]# pip install pyfaidx

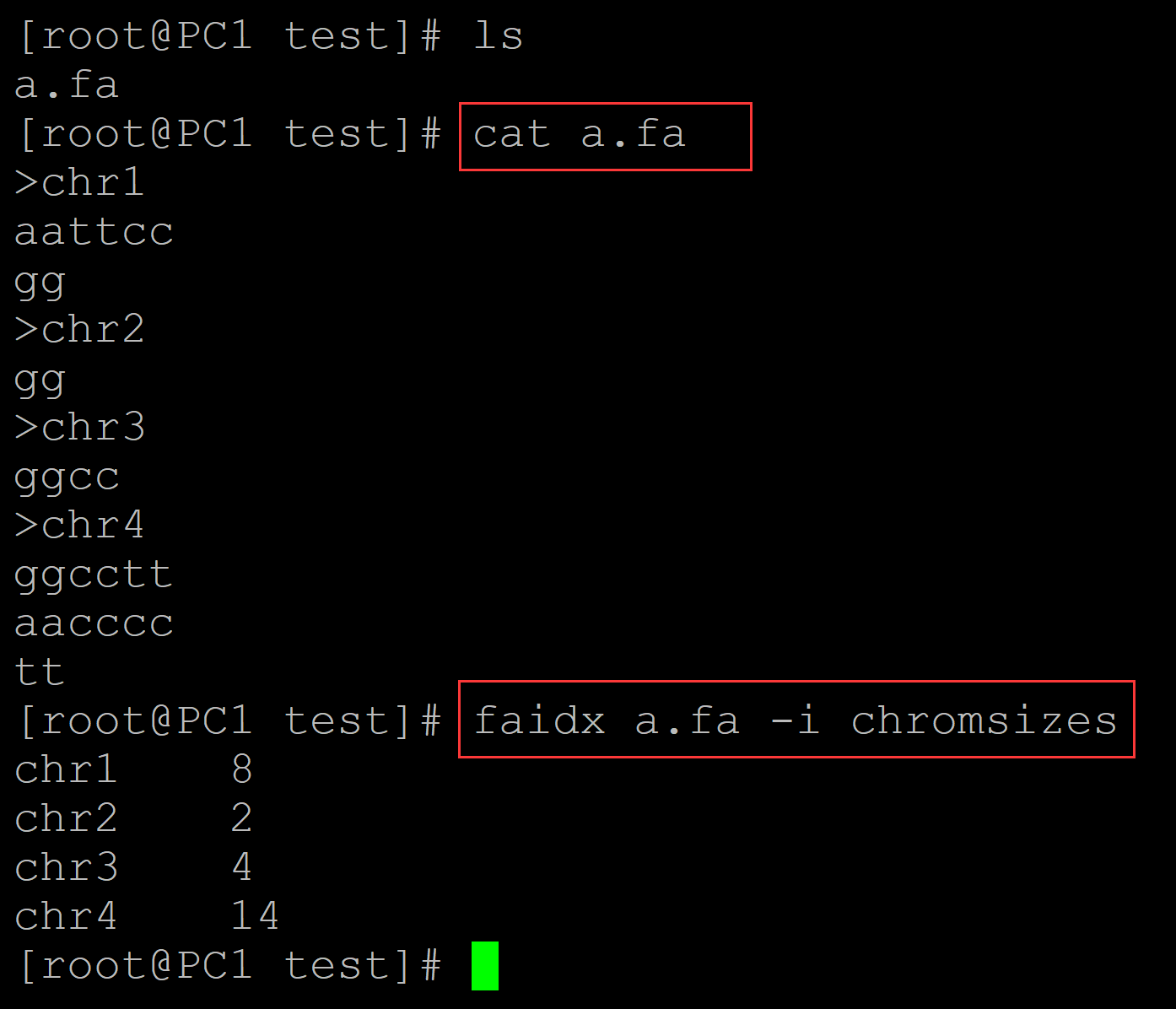
003、利用pyfaidx统计染色体的长度
[root@PC1 test]# ls a.fa [root@PC1 test]# cat a.fa ## 测试fasta文件 >chr1 aattcc gg >chr2 gg >chr3 ggcc >chr4 ggcctt aacccc tt [root@PC1 test]# faidx a.fa -i chromsizes ## 统计每一条scaffold的长度 chr1 8 chr2 2 chr3 4 chr4 14






【推荐】国内首个AI IDE,深度理解中文开发场景,立即下载体验Trae
【推荐】编程新体验,更懂你的AI,立即体验豆包MarsCode编程助手
【推荐】抖音旗下AI助手豆包,你的智能百科全书,全免费不限次数
【推荐】轻量又高性能的 SSH 工具 IShell:AI 加持,快人一步
· 震惊!C++程序真的从main开始吗?99%的程序员都答错了
· 【硬核科普】Trae如何「偷看」你的代码?零基础破解AI编程运行原理
· 单元测试从入门到精通
· 上周热点回顾(3.3-3.9)
· winform 绘制太阳,地球,月球 运作规律
2022-05-15 org.Mm.eg.db包的安装
2022-05-15 R语言中assign函数
2022-05-15 R语言中mode和class的区别
2022-05-15 R语言中attr()函数