linux 中 awk命令实现将fasta文件中每个scaffold中的所有碱基转换为一行
001、
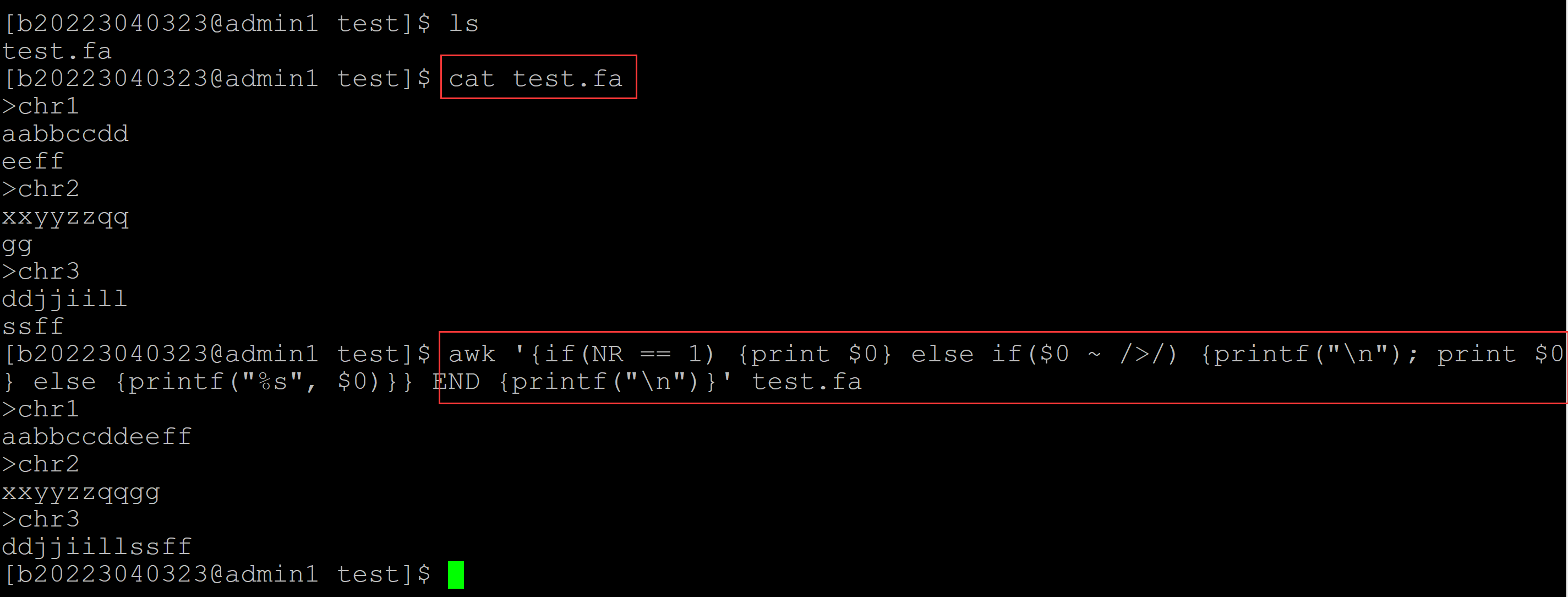
[b20223040323@admin1 test]$ ls test.fa [b20223040323@admin1 test]$ cat test.fa ## 测试数据 >chr1 aabbccdd eeff >chr2 xxyyzzqq gg >chr3 ddjjiill ssff ## 转换程序 [b20223040323@admin1 test]$ awk '{if(NR == 1) {print $0} else if($0 ~ />/) {printf("\n"); print $0} else {printf("%s", $0)}} END {printf("\n")}' test.fa >chr1 aabbccddeeff >chr2 xxyyzzqqgg >chr3 ddjjiillssff
分类:
linux shell






【推荐】国内首个AI IDE,深度理解中文开发场景,立即下载体验Trae
【推荐】编程新体验,更懂你的AI,立即体验豆包MarsCode编程助手
【推荐】抖音旗下AI助手豆包,你的智能百科全书,全免费不限次数
【推荐】轻量又高性能的 SSH 工具 IShell:AI 加持,快人一步
· 震惊!C++程序真的从main开始吗?99%的程序员都答错了
· 【硬核科普】Trae如何「偷看」你的代码?零基础破解AI编程运行原理
· 单元测试从入门到精通
· 上周热点回顾(3.3-3.9)
· winform 绘制太阳,地球,月球 运作规律
2021-02-22 python中实现列表的倒序排列