R语言中利用prcomp函数 对基因型数据进行PCA分析
001、plink软件生成raw文件
root@PC1:/home/test# ls outcome.map outcome.ped ## maf过滤,并将基因型数据转换为0、1、2的形式 root@PC1:/home/test# plink --file outcome --sheep --maf 0.01 --recode A 1> /dev/null; rm *.log *.nosex root@PC1:/home/test# ls outcome.map outcome.ped plink.raw root@PC1:/home/test# sed 1d plink.raw | cut -d " " -f 7- > use.raw ## 修改数据格式 root@PC1:/home/test# ls outcome.map outcome.ped plink.raw use.raw
002、R读入,并使用prcomp函数计算, 然后绘图
dat <- read.table("use.raw") ## 读入数据 pca <- prcomp(dat,center = TRUE,scale. = TRUE) ## prcomp函数进行pca分析 plot(pca$x[,1], pca$x[,2], pch = 19) ## 绘图
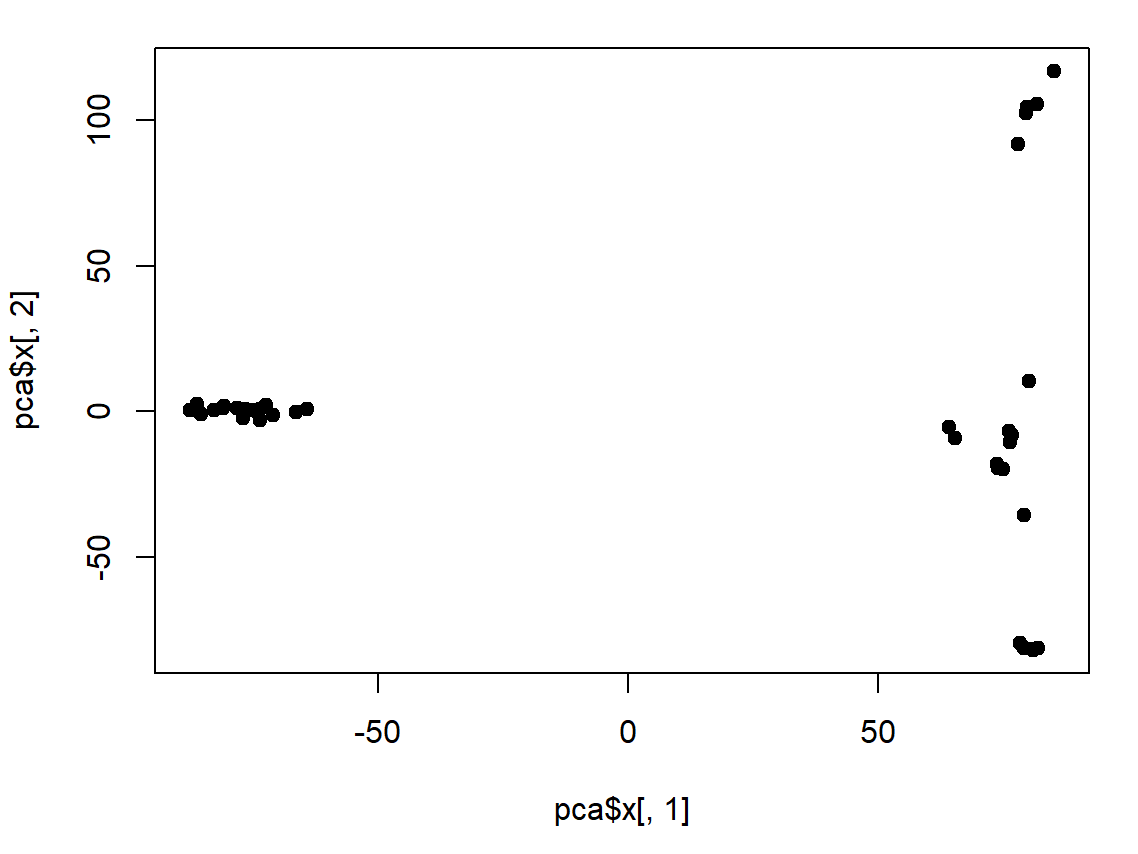
002、plink软件pca验证
root@PC1:/home/test# ls outcome.map outcome.ped ## plink软件计算pca root@PC1:/home/test# plink --file outcome --sheep --pca 3 header tabs --out pca 1> /dev/null; rm *.log *.nosex root@PC1:/home/test# ls ## 结果文件 outcome.map outcome.ped pca.eigenval pca.eigenvec
R绘图:
par(mfrow = c(1,2)) ##分割画布 dat <- read.table("use.raw") pca <- prcomp(dat,center = TRUE,scale. = TRUE) plot(pca$x[,1], pca$x[,2], pch = 19, main = "R") ## R中生成的结果 dat2 <- read.table("pca.eigenvec", header = T) plot(dat2$PC1, dat2$PC2, pch = 19, main = "plink") ## plink中生成的结果
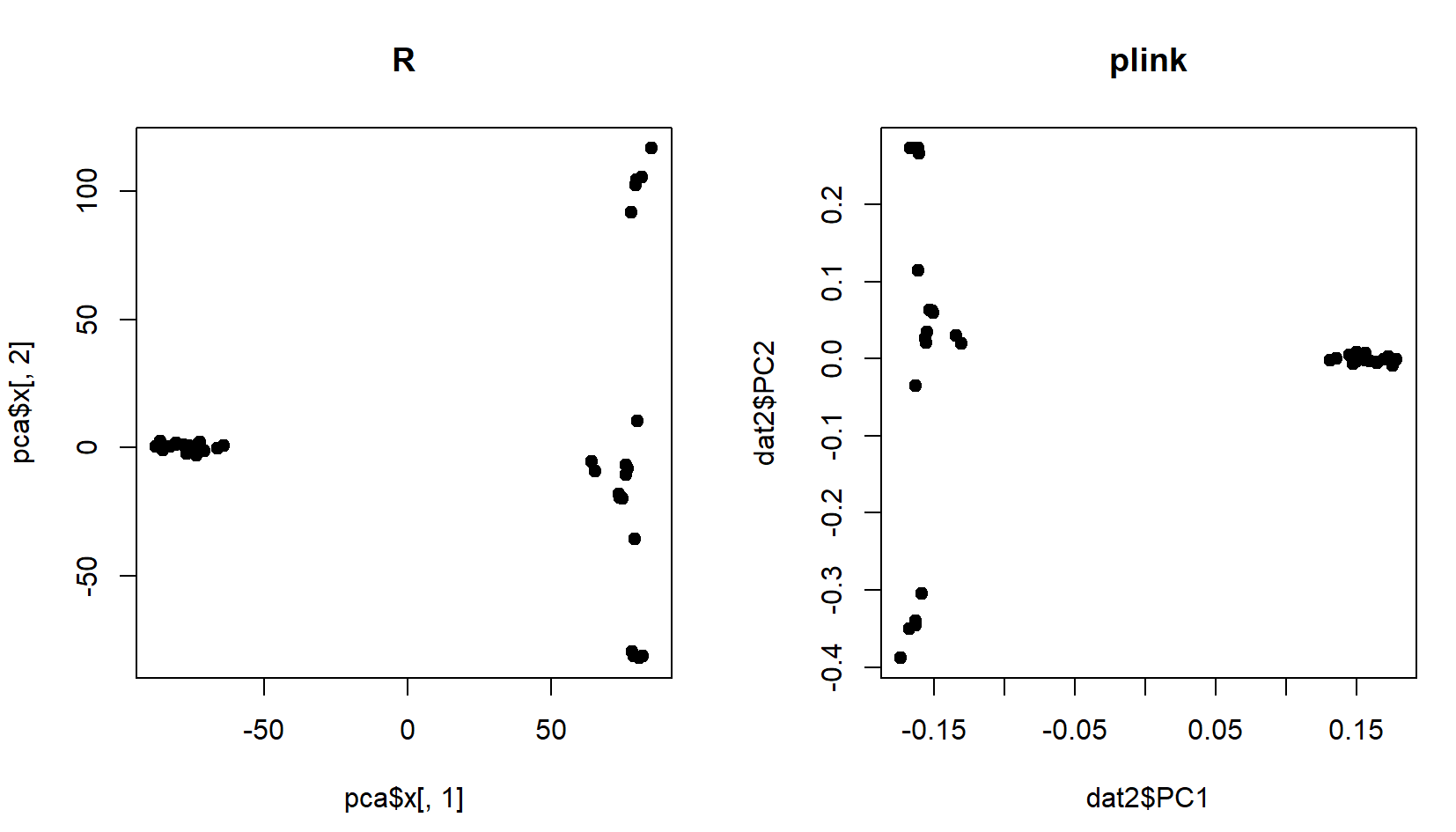
为什么结果时反着呢?





【推荐】国内首个AI IDE,深度理解中文开发场景,立即下载体验Trae
【推荐】编程新体验,更懂你的AI,立即体验豆包MarsCode编程助手
【推荐】抖音旗下AI助手豆包,你的智能百科全书,全免费不限次数
【推荐】轻量又高性能的 SSH 工具 IShell:AI 加持,快人一步
· 震惊!C++程序真的从main开始吗?99%的程序员都答错了
· 【硬核科普】Trae如何「偷看」你的代码?零基础破解AI编程运行原理
· 单元测试从入门到精通
· 上周热点回顾(3.3-3.9)
· winform 绘制太阳,地球,月球 运作规律
2020-09-08 linux 系统如何给软件设置环境变量