GWAS分析中一般线性模型GML + 协变量中snp的回归系数r2的计算
001、plink + R
root@PC1:/home/test# ls gwas_test.map gwas_test.ped root@PC1:/home/test# plink --file gwas_test --recode A 1> /dev/null root@PC1:/home/test# ls gwas_test.map gwas_test.ped plink.log plink.raw root@PC1:/home/test# plink --file gwas_test --pca 3 header 1> /dev/null root@PC1:/home/test# ls gwas_test.map gwas_test.ped plink.eigenval plink.eigenvec plink.log plink.raw root@PC1:/home/test# awk '{print $2,$6}' plink.raw | paste -d " " - <(cut -d " " -f 3- plink.eigenvec ) | paste -d " " - <(cut -d " " -f 7- plink.raw ) > dat.txt root@PC1:/home/test# head -n 3 dat.txt | cut -d " " -f 1-10 | column -t IID PHENOTYPE PC1 PC2 PC3 snp1_G snp2_T snp3_G snp4_T snp5_G id1 580 -0.0102664 0.0442708 -0.0658835 0 1 1 0 1 id2 690 0.0421124 -0.0313769 -0.0705913 0 0 0 0 0
读入R,计算r2:
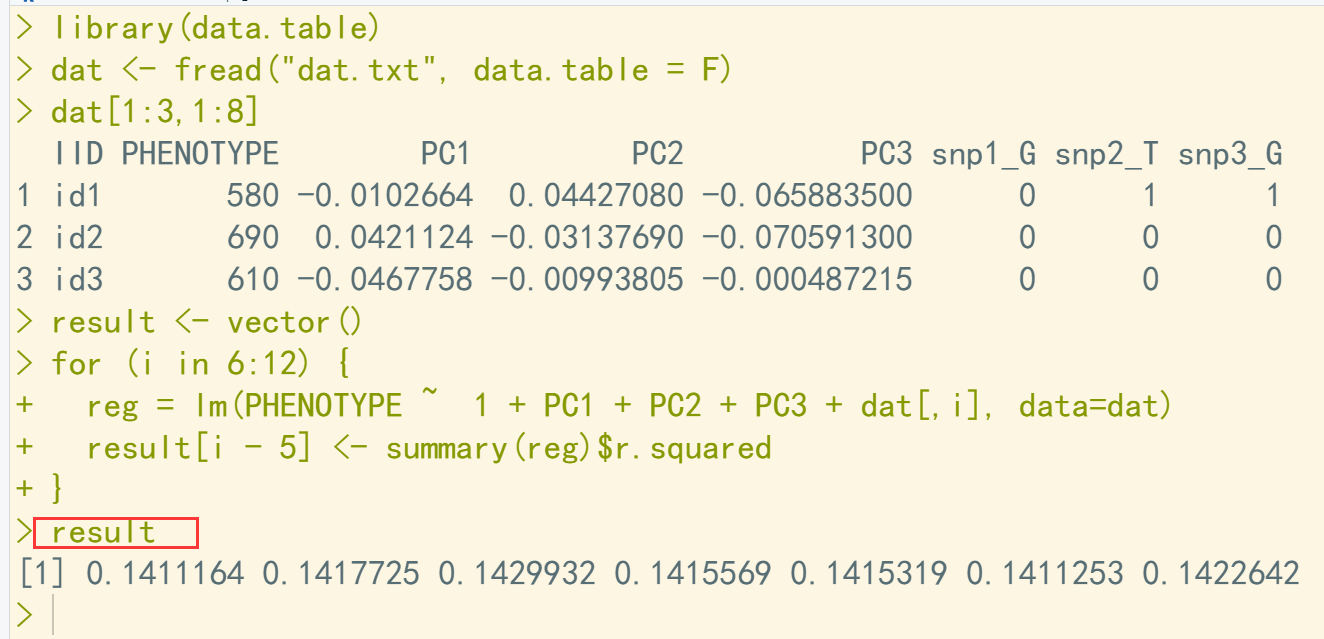
library(data.table) dat <- fread("dat.txt", data.table = F) dat[1:3,1:8] result <- vector() for (i in 6:12) { reg = lm(PHENOTYPE ~ 1 + PC1 + PC2 + PC3 + dat[,i], data=dat) result[i - 5] <- summary(reg)$r.squared } result
分类:
生信






【推荐】国内首个AI IDE,深度理解中文开发场景,立即下载体验Trae
【推荐】编程新体验,更懂你的AI,立即体验豆包MarsCode编程助手
【推荐】抖音旗下AI助手豆包,你的智能百科全书,全免费不限次数
【推荐】轻量又高性能的 SSH 工具 IShell:AI 加持,快人一步
· 震惊!C++程序真的从main开始吗?99%的程序员都答错了
· 【硬核科普】Trae如何「偷看」你的代码?零基础破解AI编程运行原理
· 单元测试从入门到精通
· 上周热点回顾(3.3-3.9)
· winform 绘制太阳,地球,月球 运作规律
2021-07-27 c primer plus 3.11-2
2021-07-27 c primer plus 3.11
2021-07-27 c语言中如何处理整数值的上溢和下溢