单细胞数据分析-数据的标准化 NormalizeData函数
001、
pbmc <- NormalizeData(pbmc)
002、实现过程
dat <- pbmc[["RNA"]]@counts dat <- as.data.frame(dat) for (i in 1:ncol(dat)) { dat[,i] <- log1p(dat[,i]/sum(dat[,i]) * 10000) } tail(dat)[, 1:5]
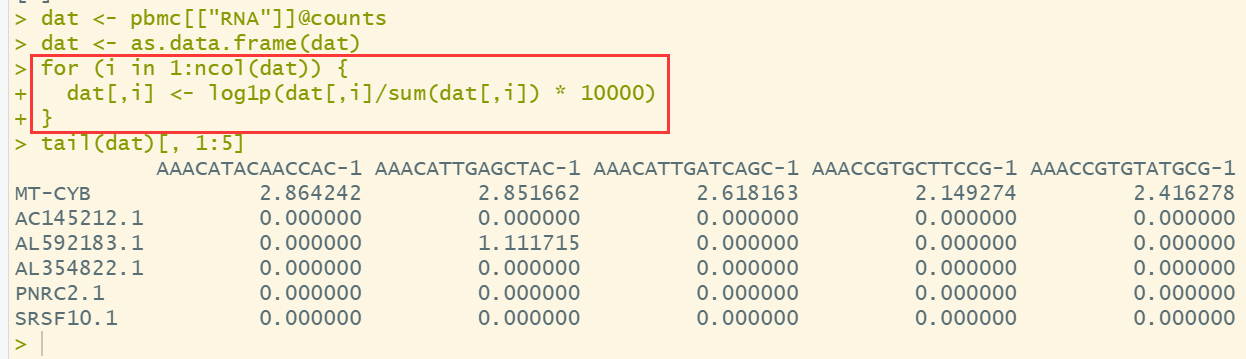
001、
pbmc <- NormalizeData(pbmc)
002、实现过程
dat <- pbmc[["RNA"]]@counts dat <- as.data.frame(dat) for (i in 1:ncol(dat)) { dat[,i] <- log1p(dat[,i]/sum(dat[,i]) * 10000) } tail(dat)[, 1:5]

