单细胞数据处理中数据标准化函数NormalizeData()实现细节
数据来源:https://www.jianshu.com/p/4f7aeae81ef1
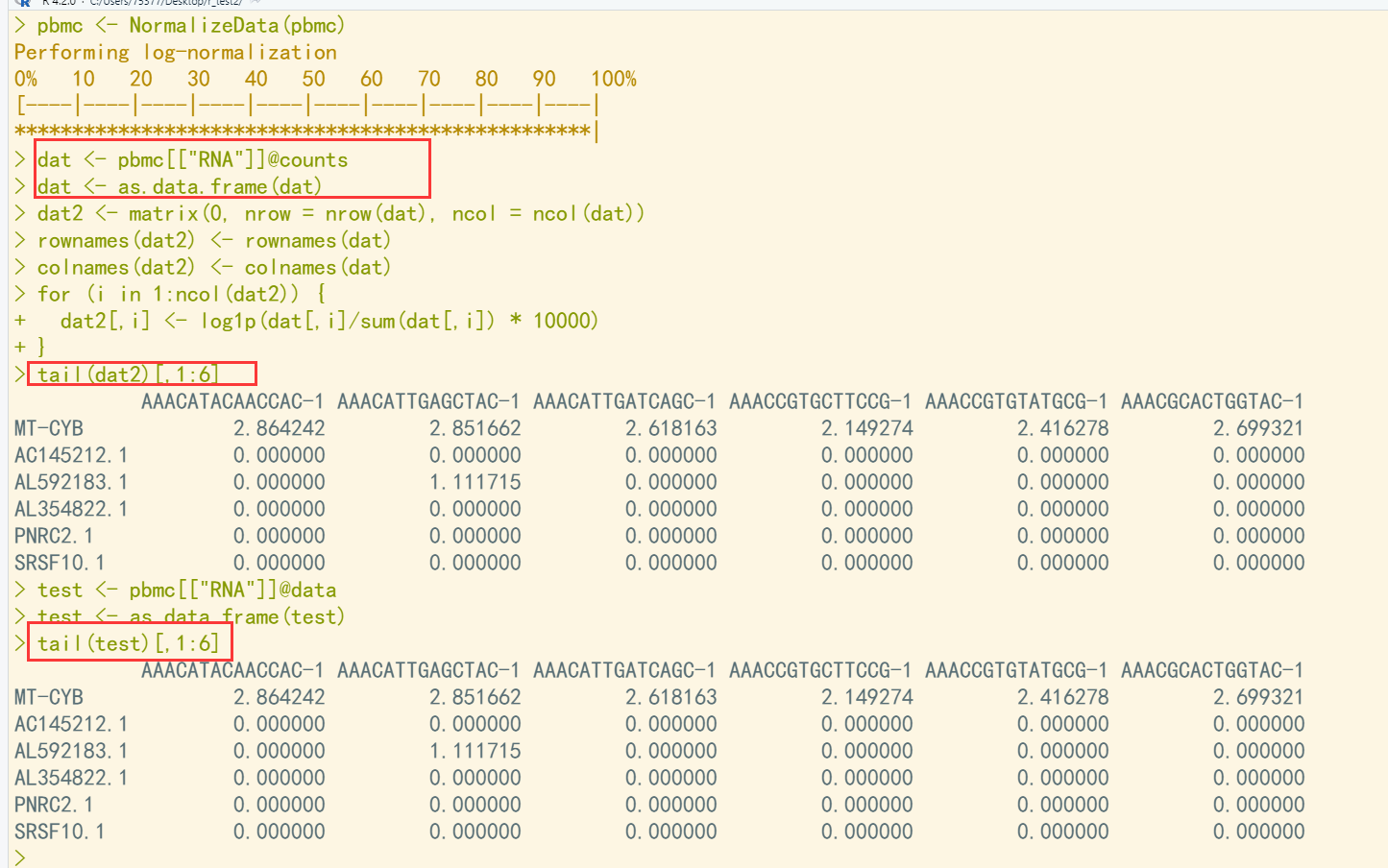
1、
pbmc <- NormalizeData(pbmc) dat <- pbmc[["RNA"]]@counts ## 原始数据 dat <- as.data.frame(dat) dat2 <- matrix(0, nrow = nrow(dat), ncol = ncol(dat)) ## 手动标准化变量 rownames(dat2) <- rownames(dat) colnames(dat2) <- colnames(dat) for (i in 1:ncol(dat2)) { dat2[,i] <- log1p(dat[,i]/sum(dat[,i]) * 10000) ## NormalizeData()实现过程 } tail(dat2)[,1:6] test <- pbmc[["RNA"]]@data ## NormalizeData()函数标准化结果
test <- as.data.frame(test) tail(test)[,1:6]
分类:
R语言






【推荐】国内首个AI IDE,深度理解中文开发场景,立即下载体验Trae
【推荐】编程新体验,更懂你的AI,立即体验豆包MarsCode编程助手
【推荐】抖音旗下AI助手豆包,你的智能百科全书,全免费不限次数
【推荐】轻量又高性能的 SSH 工具 IShell:AI 加持,快人一步
· 震惊!C++程序真的从main开始吗?99%的程序员都答错了
· 【硬核科普】Trae如何「偷看」你的代码?零基础破解AI编程运行原理
· 单元测试从入门到精通
· 上周热点回顾(3.3-3.9)
· winform 绘制太阳,地球,月球 运作规律
2021-06-02 c语言中strncp函数,函数原型、头文件
2021-06-02 c语言中strcpy函数,函数原型、头文件
2021-06-02 c语言 11-6
2021-06-02 c语言 11-5
2021-06-02 c语言 11-4
2021-06-02 c语言中求字符串的长度
2021-06-02 c语言 11-3