plink软件统计缺失率
1、准备测试数据
[root@linuxprobe test3]# cat test.map
1 snp1 0 55910
1 snp2 0 85204
1 snp3 0 122948
1 snp4 0 203750
1 snp5 0 312707
1 snp6 0 356863
1 snp7 0 400518
1 snp8 0 487423
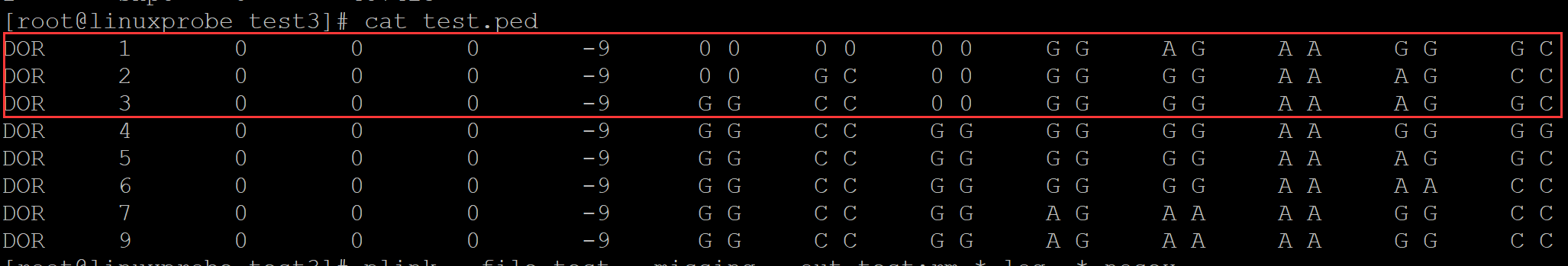
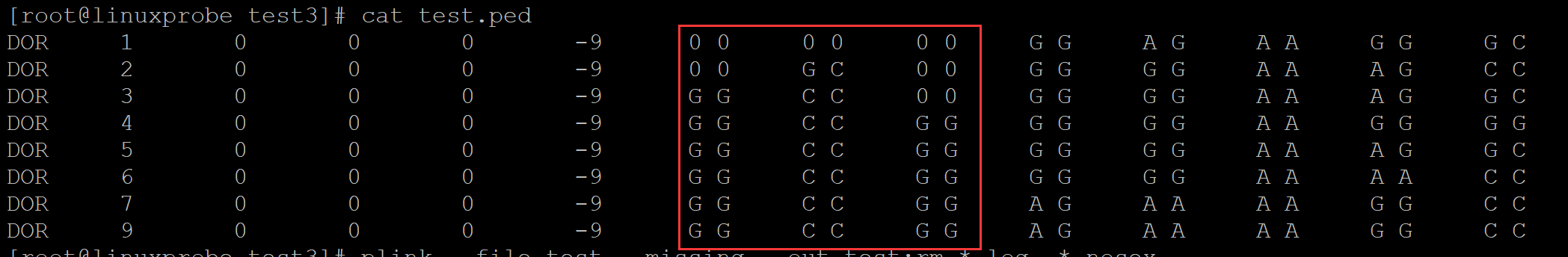
[root@linuxprobe test3]# cat test.ped
DOR 1 0 0 0 -9 0 0 0 0 0 0 G G A G A A G G G C
DOR 2 0 0 0 -9 0 0 G C 0 0 G G G G A A A G C C
DOR 3 0 0 0 -9 G G C C 0 0 G G G G A A A G G C
DOR 4 0 0 0 -9 G G C C G G G G G G A A G G G G
DOR 5 0 0 0 -9 G G C C G G G G G G A A A G G C
DOR 6 0 0 0 -9 G G C C G G G G G G A A A A C C
DOR 7 0 0 0 -9 G G C C G G A G A A A A G G C C
DOR 9 0 0 0 -9 G G C C G G A G A A A A G G C C
2、统计缺失率 加 --missing参数即可
plink --file test --missing --out test;rm *.log *.nosex ##加 --missing 参数即可
[root@linuxprobe test3]# ls
test.imiss test.lmiss test.map test.ped
[root@linuxprobe test3]# cat test.imiss ## 个体缺失率
FID IID MISS_PHENO N_MISS N_GENO F_MISS
DOR 1 Y 3 8 0.375
DOR 2 Y 2 8 0.25
DOR 3 Y 1 8 0.125
DOR 4 Y 0 8 0
DOR 5 Y 0 8 0
DOR 6 Y 0 8 0
DOR 7 Y 0 8 0
DOR 9 Y 0 8 0
[root@linuxprobe test3]# cat test.lmiss ##位点缺失率
CHR SNP N_MISS N_GENO F_MISS
1 snp1 2 8 0.25
1 snp2 1 8 0.125
1 snp3 3 8 0.375
1 snp4 0 8 0
1 snp5 0 8 0
1 snp6 0 8 0
1 snp7 0 8 0
1 snp8 0 8 0






【推荐】国内首个AI IDE,深度理解中文开发场景,立即下载体验Trae
【推荐】编程新体验,更懂你的AI,立即体验豆包MarsCode编程助手
【推荐】抖音旗下AI助手豆包,你的智能百科全书,全免费不限次数
【推荐】轻量又高性能的 SSH 工具 IShell:AI 加持,快人一步
· 基于Microsoft.Extensions.AI核心库实现RAG应用
· Linux系列:如何用heaptrack跟踪.NET程序的非托管内存泄露
· 开发者必知的日志记录最佳实践
· SQL Server 2025 AI相关能力初探
· Linux系列:如何用 C#调用 C方法造成内存泄露
· 震惊!C++程序真的从main开始吗?99%的程序员都答错了
· 【硬核科普】Trae如何「偷看」你的代码?零基础破解AI编程运行原理
· 单元测试从入门到精通
· 上周热点回顾(3.3-3.9)
· winform 绘制太阳,地球,月球 运作规律