文献分析 FEAST 单细胞变量选择
原文pdf连接
https://www.ncbi.nlm.nih.gov/pmc/articles/PMC8644062/
摘要
好的特征区分不同细胞类型
FEAST的特征选择结果可以提升聚类效果
|
|
Progress
|
Challenge
|
Demand
|
|
Background
|
单细胞技术 细胞数据的聚类中有一步很关键:特征选择 |
没有专门研究评估变量选择带来的影响 |
|
|
Solve
|
What
|
How
|
Effect
|
|
广泛评估和比较变量选择在单细胞数据上的影响
主要是对聚类的影响
|
FEAST 是一个为单细胞聚类选择代表性基因的方法
|
优于现有选择方法
提升聚类精确度
|
|
|
Result
|
Feature selection in scRNA-seq cell clustering大多数基因在不同细胞类型之间没有差异表达 特征选择无监督 Seurat中的方法:将基因按平均表达分为20个bins,再寻找每个bins中的方差最大基因 F-test基于组间方差和组内方差的比值评估辨别效率 |
||
Feature evaluation in scRNA-seq cell clustering直接评估:评估聚类后与真实标签的一致性 没有标签数据时:使用“伪监督”的方法:根据变量选择的结果检查cluster的分离:如果一组特征导致具有较大组间距离和较小组内距离的集群,则认为该组特征更好。 |
|||
特征选择确实对聚类结果有很大影响FEAST 是生成具有代表性的特征集以提高聚类精度。 FEAST 首先进行共识聚类得到初始细胞聚类结果,基于初始聚类进行特征的排序和选择。 最佳特征数量由来自不同数量top特征的聚类结果的fitness决定。 我们证明了 FEAST 可以识别更具代表性的特征并显着提高聚类精度。 |
|||
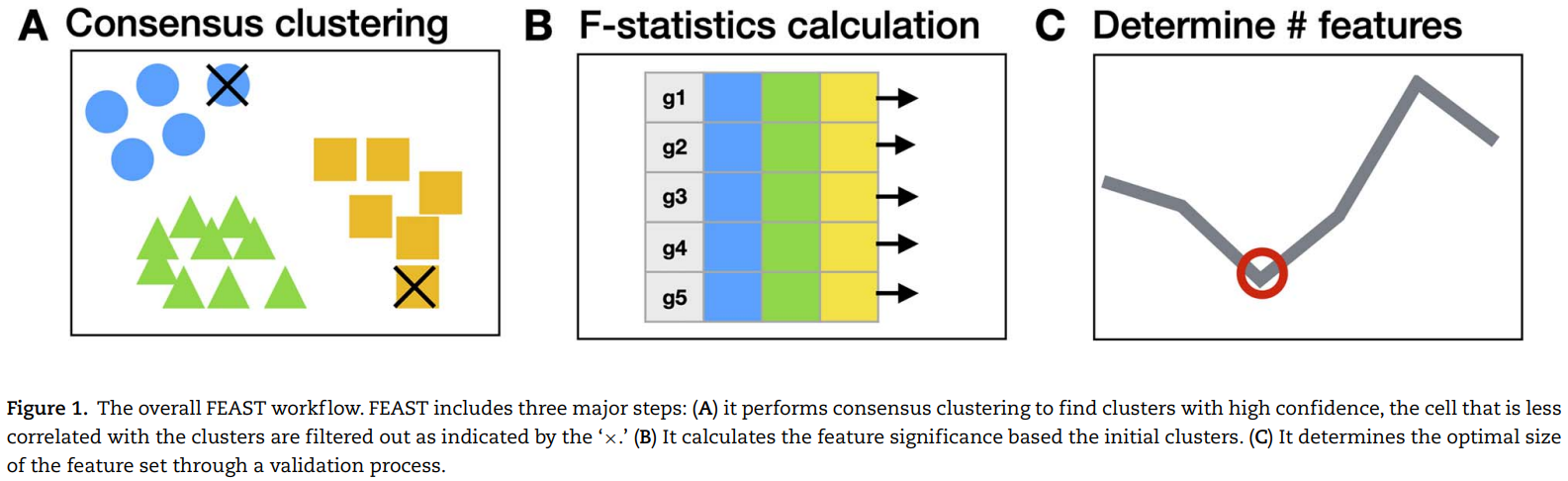
Overview of FEAST
1.通过高效的方法得到共识聚类结果,主要是为了得到可信的聚类结果,为了在下一步特征选择中使用 2.基于聚类结果,通过F-test计算每个特征的显著性,根据F-检验统计量排序特征 3.通过特征评估算法选择最佳的特征集合 |
|||
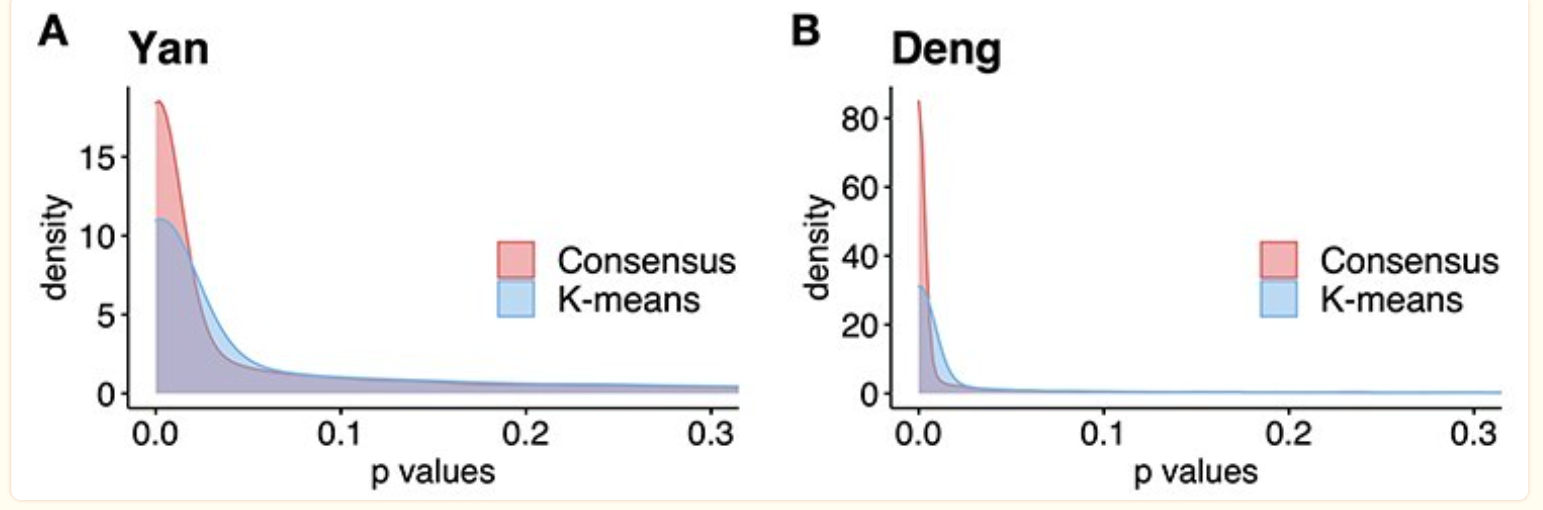
Consensus clustering improves the signal
通过共识聚类得到的p值更显著 |
|||
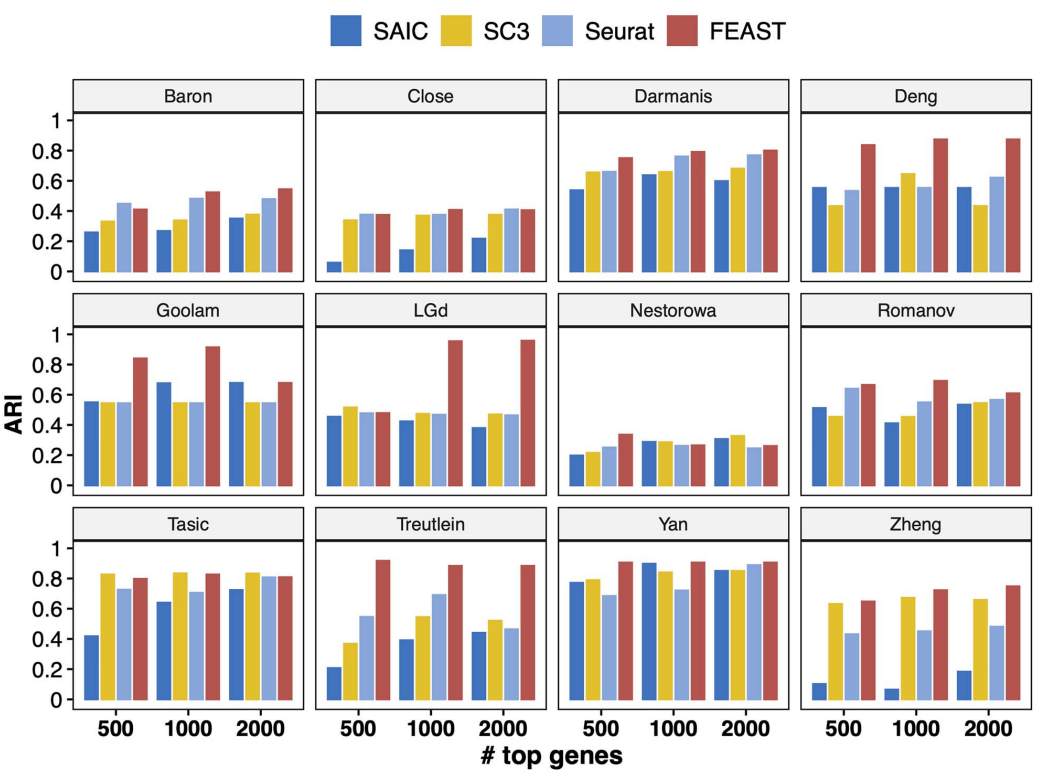
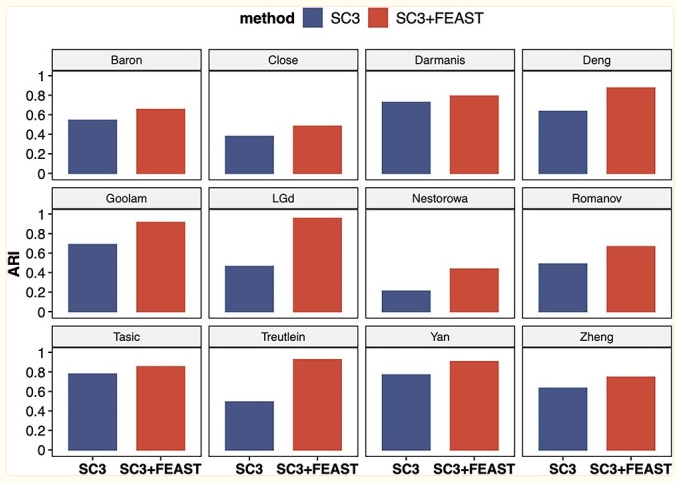
FEAST selects features better than other unsupervised approaches对比FEAST ,SAIC, SC3 and Seurat三种方法 通过不同的top数量评估不同方法的聚类效果
研究发现,有些top的基因仅在少数细胞中显示出极高的表达,而在其余细胞中保持相同(通常为 0)。这些是表达分布高度偏斜的基因,显然不是很好的聚类特征。 (确实需要与已知的marker求交集,仅保留有生物学意义的特征) 而且有时包含了过多的特征起始并没有带来本质的提升 |
|||
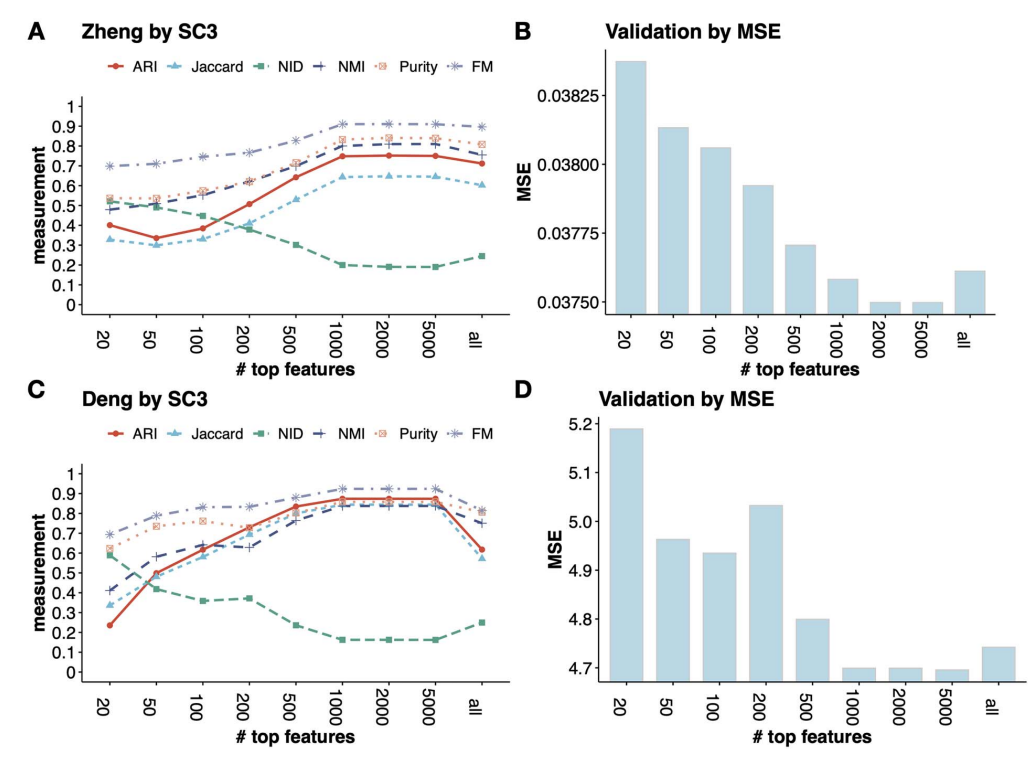
FEAST optimize the feature set through validation
基于该方法评估最佳的top数量 对于没有标签的数据可以设计基于MSE的交叉验证方法确定最佳feature数量 |
|||
FEAST improves the clustering accuracy
Test FEAST on larger datasets |
|||
|
Conclusion& Discussion
|
|
||
|
Method
|
基于初始聚类结果进行F检验
F 统计量本质上是计算组间方差 (varb) 和组内方差 (varw) 之间的比例
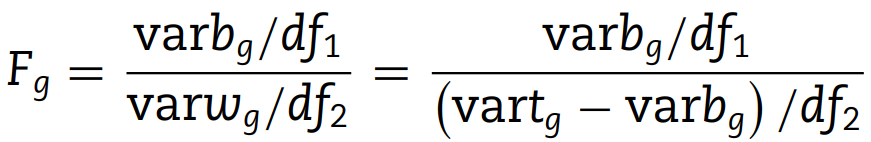 对每个基因计算一下这个式子,
其中,df1 = K-1,df2 = N' - K 为自由度
N'是共识聚类clusters的细胞数总数
K是找到的类别数
FEAST 使用总方差 (vartg) 和组间方差之间的差异来表示组内方差
varbg=
vartg=
|
||
"The world is a fine place and worth fighting for." I agree with the second part.