文献分析 scNT-seq
原文pdf连接
https://www.nature.com/articles/s41592-020-0935-4.pdf
摘要
区分单个细胞中新生的mRNA和之前就存在的mRNA
有效地标记新转录的具有 T 到 C 替换的 mRNA。
|
|
Progress
|
Challenge
|
Demand
|
|
Background
|
要研究不同细胞基因表达的动态情况 |
普通scRNA技术捕获的是新旧mRNA混合的结果,无法解析RNA动力学 |
|
|
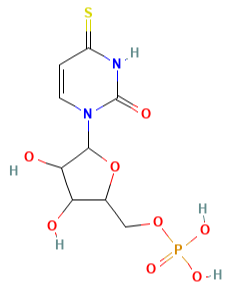
传统区分新旧RNA的方式为,利用外源性核苷类似物进行RNA代谢标记(4sU),然后再对标记的RNA进行生化富集; 4su = 4-thiouridine = 4-硫尿苷
|
问题:需要样本量大?富集的效果? |
|
|
|
又有一些方法是将4sU换成胞苷类似物,替换的结果是尿嘧啶U-胞嘧啶C的替换,这样的方法不需要生化富集 |
|
||
| 已有研究开发出将单细胞技术(smartseq)与上述标记技术相结合,也就是RNA代谢测序 |
通量低,成本高;没有UMI,无法定量新生转录本水平 |
|
|
|
Solve
|
What
|
How
|
Effect
|
|
scNT-seq
a
high-throughput and UMI-based scRNA-seq method |
结合RNA代谢标记,微流控,将 4sU 重新编码为胞嘧啶类似物,以同时测量来自同一细胞的新旧转录组
|
对细胞 RNA 动力学进行时间分辨分析
|
|
|
Result
|
Development and validation of scNT-seq.
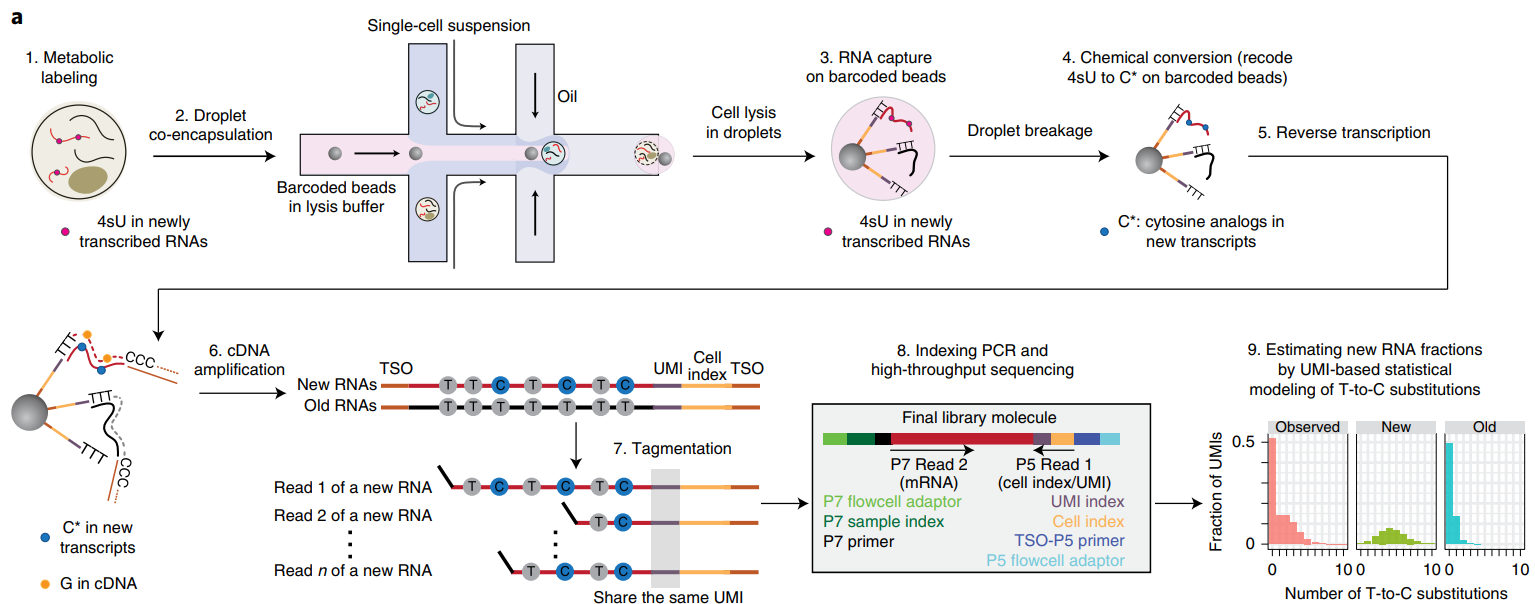 1.通过4sU代谢标记,4sU会在转录过程中替换U的角色,但凡在标记后新生成了RNA,都会标记上4sU
2.在逆转录之间将4sU处理为C*类似物
最终的结果是测序结果中出现了T-to-C的转换
|
||
|
1.Development and validation of scNT-seq.
2.Evaluating scNT-seq performance for detecting activity-induced
new RNAs. 3.Identification of neuronal activity-induced, time-resolved regulon activity.
新旧转录本转录因子的调控活性 SCENIC
4.Metabolic labeling-based time-resolved RNA velocity analysis.
基于代谢标记的RNA速率 dynamo
5.scNT-seq reveals distinct RNA regulatory strategies during stem cell-state transition.
为研究罕见瞬时细胞群的RNA调控策略
多能 - 中间 - 全能C2样(调控机制不清楚)
|
|||
|
Conclusion& Discussion
|
|
||
|
|
|
||
|
|
Read alignment and quantification of metabolically labeled transcripts.
每个细胞中高质量的reads取出来,通过UMI分组
仅保留质量值大于27的TC转换
背景的TC转换需要根据对照组的数据得到再减去
但凡发现TC转换的UMI就是新生的了
|
||
|
Method
|
Estimation of the fraction of newly synthesized transcripts.
基于二项分布混合模型
二项分布模拟的是每个基因转录本T-to-C发生的数量
其中θ是新生转录本的比例,pq是新旧转录本TC替换发生在每个核苷酸的概率
ni是U碱基在转录本i中的观察值
|
||
"The world is a fine place and worth fighting for." I agree with the second part.
分类:
文献分析
« 上一篇: 文献分析 Single-cell transcriptomic analysis in a mouse model deciphers cell transition states in the multistep development of esophageal cancer 小鼠模型中的单细胞转录组学分析破译食管癌多步发展中的细胞过渡状态
» 下一篇: 文献分析 基于监督学习的细胞类型注释策略 Evaluation of some aspects in supervised cell type identification for single-cell RNAseq: classifier, feature selection, and reference construction
» 下一篇: 文献分析 基于监督学习的细胞类型注释策略 Evaluation of some aspects in supervised cell type identification for single-cell RNAseq: classifier, feature selection, and reference construction




【推荐】国内首个AI IDE,深度理解中文开发场景,立即下载体验Trae
【推荐】编程新体验,更懂你的AI,立即体验豆包MarsCode编程助手
【推荐】抖音旗下AI助手豆包,你的智能百科全书,全免费不限次数
【推荐】轻量又高性能的 SSH 工具 IShell:AI 加持,快人一步
· winform 绘制太阳,地球,月球 运作规律
· AI与.NET技术实操系列(五):向量存储与相似性搜索在 .NET 中的实现
· 超详细:普通电脑也行Windows部署deepseek R1训练数据并当服务器共享给他人
· 【硬核科普】Trae如何「偷看」你的代码?零基础破解AI编程运行原理
· 上周热点回顾(3.3-3.9)