文献分析 PseudotimeDE: inference of differential gene expression along cell pseudotime with well-calibrated p-values from single-cell RNA sequencing data
原文pdf连接: https://genomebiology.biomedcentral.com/track/pdf/10.1186/s13059-021-02341-y.pdf
摘要:
为了研究细胞状态变化的分子机制,一个关键的分析是沿着从单细胞 RNA 测序数据推断的拟时间识别差异表达 (DE) 基因。然而,现有方法没有考虑拟时间推理的不确定性,并且它们具有无效 p 值或限制性模型。在这里,我们提出了 PseudotimeDE,这是一种适用于各种拟时间推理方法的 DE 基因识别方法,考虑了拟时间推理的不确定性,并输出了经过良好校准的 p 值。综合模拟和真实数据应用验证 PseudotimeDE 在错误发现率控制和功率方面优于现有方法。
|
|
Progress
|
Challenge
|
demand
|
|
background
|
scRNA-seq |
|
|
|
Pseudotime inference,differentially expressed (DE)
|
|
|
|
|
已有的方法:TSCAN [10], Slingshot [11], Monocle [8], and Monocle2
都是先提供了轨迹分析,再提供差异检验
都是通过generalized additive model拟合基因变化
|
不支持外部导入的拟时间数据
|
|
|
|
目前有两个方法支持导入的数据:
tradeSeq (NB-GAM)其 p 值计算基于卡方分布,这是对零分布的不准确近似。因此,其 p 值缺乏正确的概率解释。
Monocle3 generalized linear model (GLM) GLM 比 GAM 更具限制性,因为 GLM 假设细胞中基因counts计数的对数变换是细胞伪时间的严格线性函数,这通常是不对的。
|
以上方法都没有考虑拟时间的不确定性;
要知道,细胞的拟时间没有置信区间;
不确定性导致无效的p-value,无法FDR矫正
|
|
|
|
除了以上方法,还有一些是处理连续时间点bulk数据的;
NBAMSeq: NB-GAM + Bayesian shrinkage method in DESeq2 类似 tradeSeq
ImpulseDE2:“impulse” model,单细胞的性能未被充分评估
|
|
||
|
solve
|
what
|
how
|
effect
|
|
PseudotimeDE:
适用于用户输入的拟时间数据,
考虑拟时间的随机性,
输出经过良好校准的 p 值
|
通过抽样估计拟时间的不确定性,
将不确定性纳入DE基因分析
|
确保了FDR矫正的可靠,以及其他下游分析,避免由于过于保守的 p 值而导致的不必要的性能损失。
|
|
|
result
|
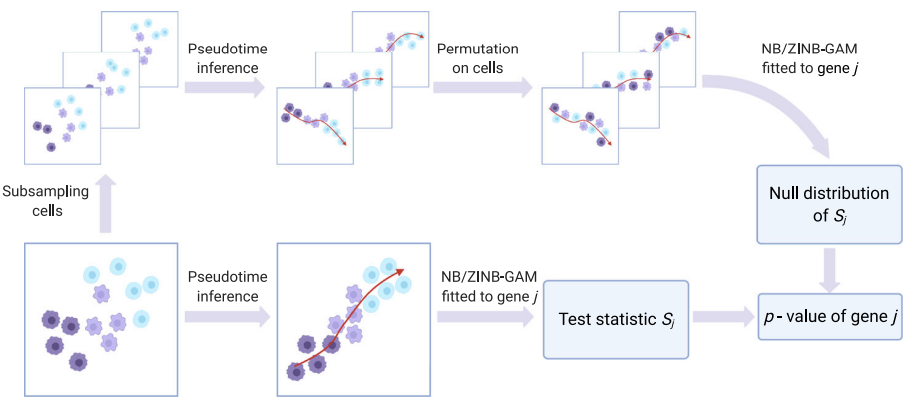
Overview of the PseudotimeDE methodfour major steps: subsampling, pseudotime inference, model fitting, and hypothesis testing
1.subsampling,每次抽样80%细胞
2.pseudotime inference, 原始数据也推断,抽样也推断;通过每个抽样的细胞重排得到0假设
3.model fitting,原始数据中的每个基因拟合NB-GAM or zero-inflated negative binomial GAM (ZINB-GAM)。
4.hypothesis testing,对于每个基因,拟合重排的抽样,得到检验统计量的0值;用每个基因的检验统计量和0值比较得到p-value
所谓检验统计两就是推断的拟时间对基因表达的影响大小,通过拟合自然得到。
 |
||
Simulations verify that PseudotimeDE outperforms existing methods in the validity of p-values and the identification powerp值的有效性
dyntoy模拟不同离散程度和分支的单细胞数据
构建拟时间的方法为Slingshot and Monocle3-PI
拟时间推断具有“线性不确定性”:在一个分支内的不确定性,不同方法线性不确定性不同
拟时间推断具有“拓扑结构不确定性”:不同抽样的结构不同,不同方法识别结构的性能不同
比较方法:PseudotimeDE,tradeSeq, Monocle3-DE, NBAMSeq, and ImpulseDE2
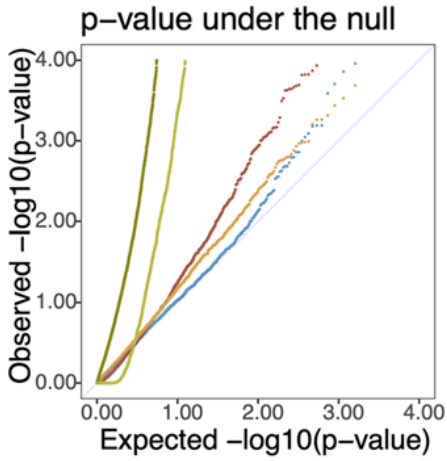
比较p值的有效性:0假设下p值应均匀分布在0-1
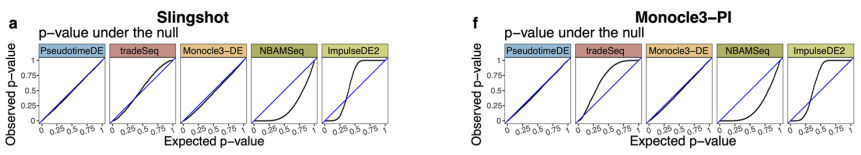 特别是在小p值情况下表现也比较好:
 比较鉴定差异基因的能力
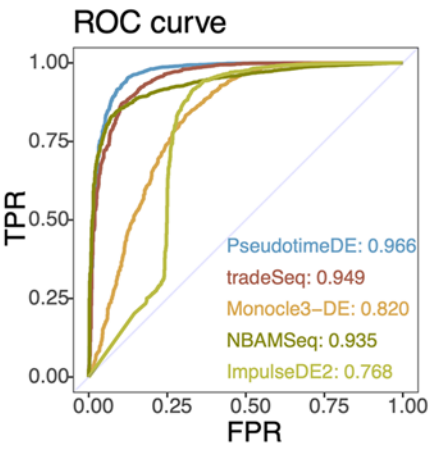 比较了所有五种方法在实际5%的错误发现比例下的能力
以上比较 PseudotimeDE 均最佳
|
|||
Real data example 1: dendritic cells stimulated with lipopolysaccharidecompare PseudotimeDE with tradeSeq and Monocle3-DE
LPS刺激后的小鼠树突状细胞 (DC)数据
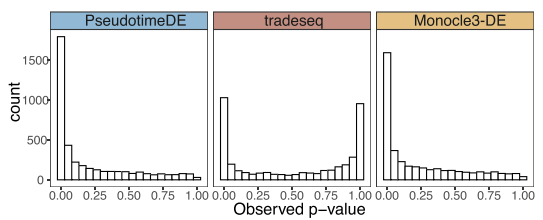 p值双峰分布使得鉴定差异的能力降低
PseudotimeDE 特异性的差异基因的GO分析发现,它找到的基因与实际更相符
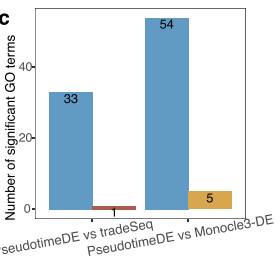 总之,我们的功能分析验证了PseudotimeDE识别出tradeSeq和Monocle3-DE遗漏的具有生物学意义的DE基因,证实了PseudotimeDE除了具有良好校准的p值外还具有高效率。
|
|||
Real data example 2: pancreatic beta cell maturationPseudotimeDE识别具与实际意义相符合的差异基因
bulk RNA DE 基因在 PseudotimeDE 发现的排名靠前的 DE 基因中最为丰富。
Slc39a10 为已知的变异基因,Sst为已知的不变基因,比较实际效果:
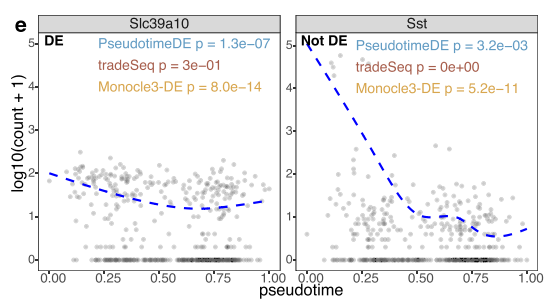 |
|||
Real data example 3: bone marrow differentiation除了p值的排序,p值的标称值对于GSEA等下游分析也很关键。因此,经过良好校准的 p 值使 PseudotimeDE 优于现有的 DE 基因鉴定和下游分析方法。 |
|||
Real data example 4: natural killer T cell subtypes多分支的情况,逐个分支检验, PseudotimeDE 最优
|
|||
Real data example 5: cell cycle phases提前确定数据的真实差异基因和非差异基因
用各种方法与实际基因比较
我们得出结论,PseudotimeDE 在从这个 iPSC scRNA-seq 数据集中识别细胞周期相关基因方面优于 tradeSeq 和 Monocle3-DE。
|
|||
Computational time1.计算前过滤低质量基因
2.减少抽样次数:>=100
|
|||
|
Conclusion& Discussion
|
-
|
||
|
method
|
-
|
||
"The world is a fine place and worth fighting for." I agree with the second part.


