3-基因家族的鉴定-基于Windows系统上的HMMER

下面讲如何在windows系统上用HMMER做基因家族的鉴定:
1, 下载
我们电脑首先要有个迅雷,然后复制这个链接,用迅雷打开(http://hmmer.janelia.org/static/binaries/hmmer3.0_windows.zip),下载到本地,解压。

2, 案例分析
HMMER是根据蛋白的结构域进行搜寻的,例如我们现在要找毛果杨的bZIP转录因子家族。在以往的文献中,我们知道拟南芥的bZIP蛋白序列,我们将拟南芥的bZIP蛋白序列提交到Pfam网站(http://pfam.xfam.org/)看看它的结构域。
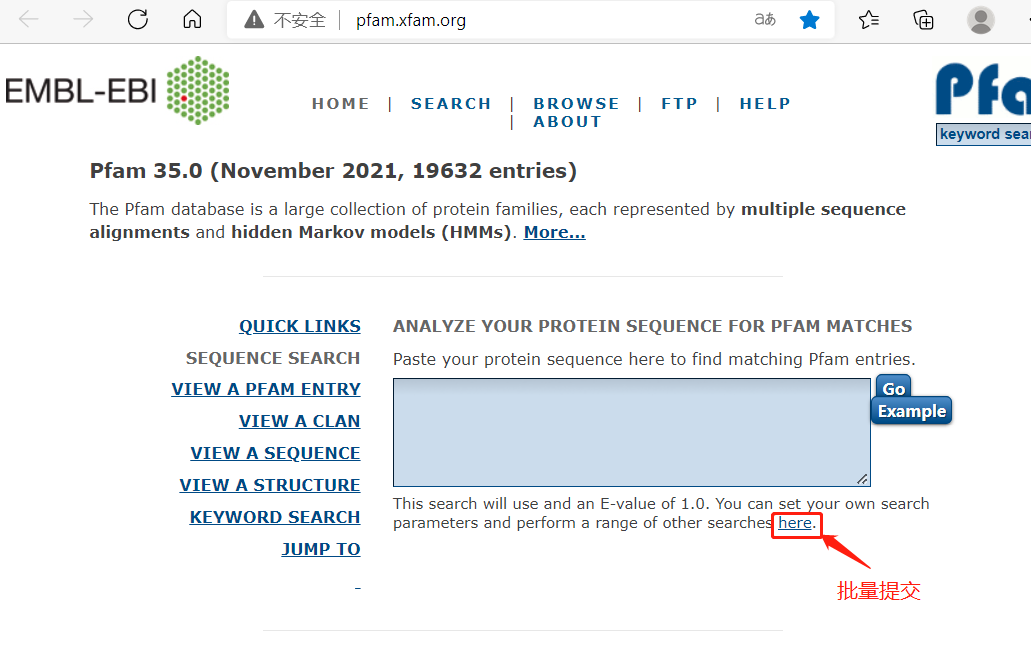
我们批量提交并填写邮件,其它默认参数,可能要等半天甚至更长,结果会发至邮箱。
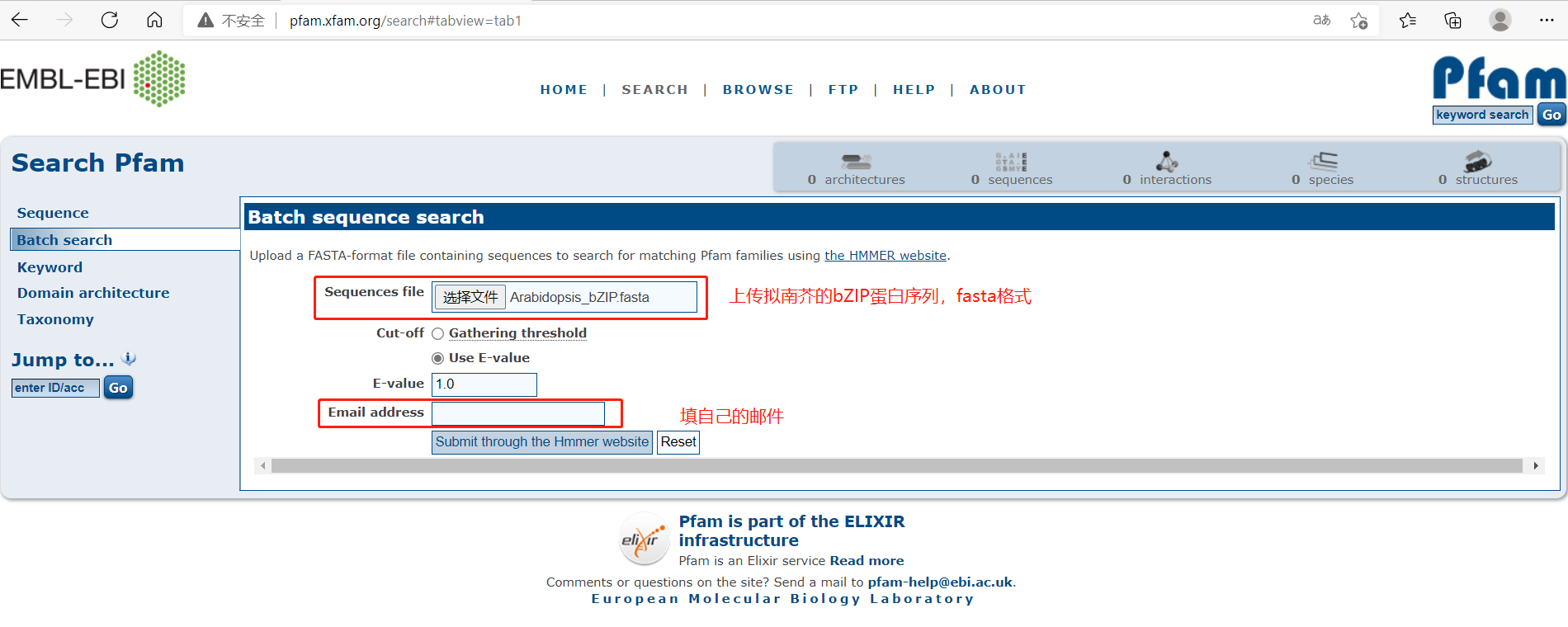
得到结果后,发现邮件里面有结果和结果的链接,点击链接可以去到网站上,结果可以也复制粘贴到excel中处理,在结果中,发现它们几乎都含有bZIP_1和bZIP_2结构域,结构域的编号分别是PF00170,PF07716。
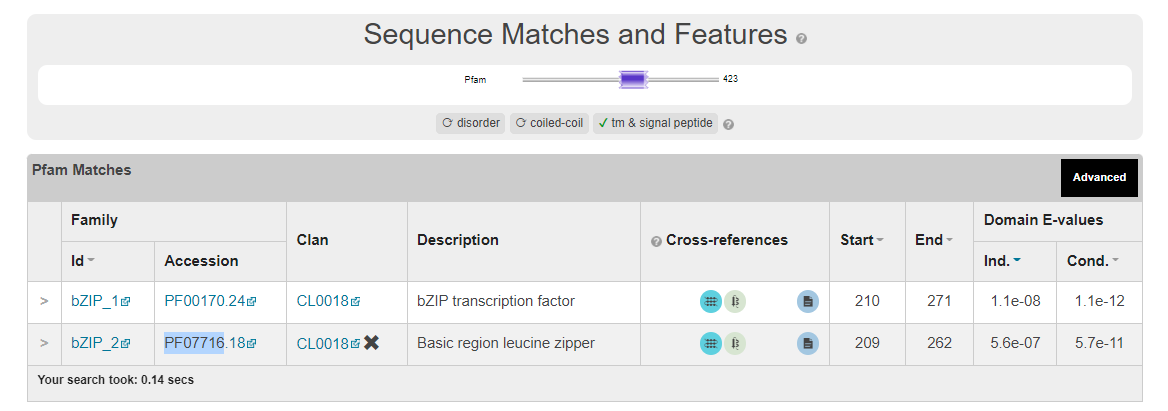
点进去,下载stockholm格式的文件。
并将这两个结构域的文件,和毛果杨的全部蛋白文件都拷贝一份到HMMER的文件夹里面
在电脑的左下角打“cmd”,进入命令提示符
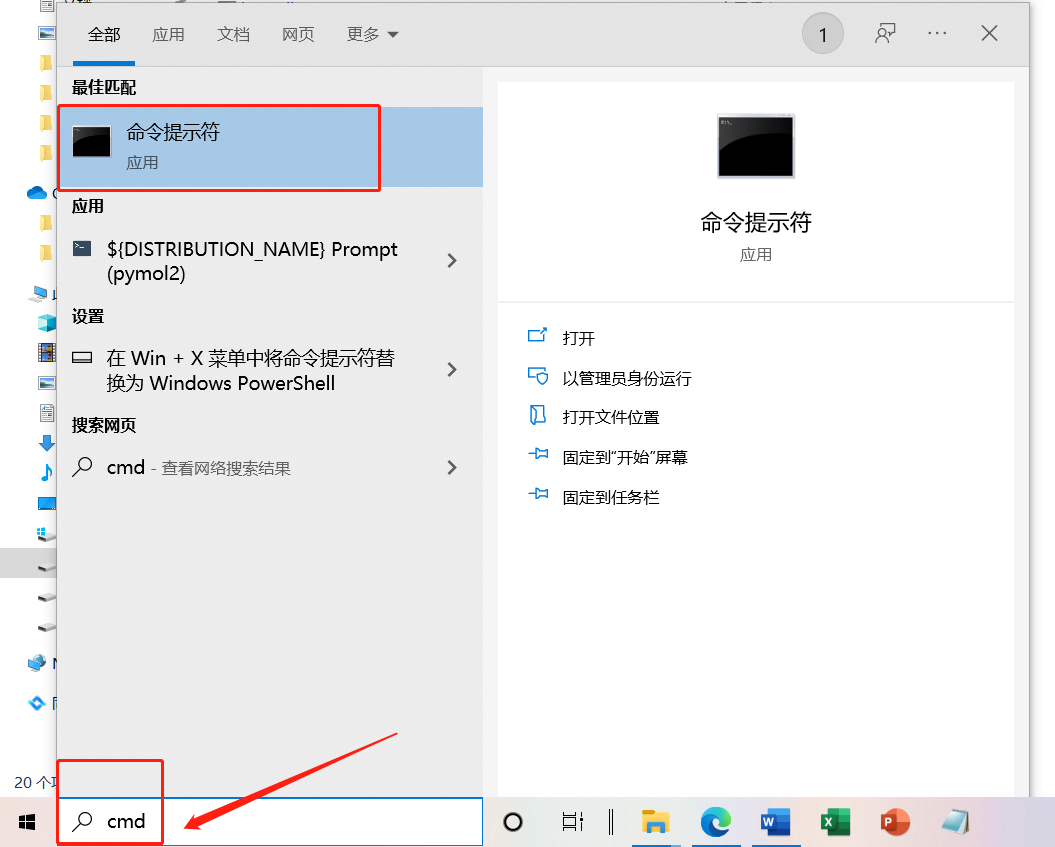
因为我的HMMER放在D盘中,路径是D:\HMMER,所以先进入D盘,输入命令
d:
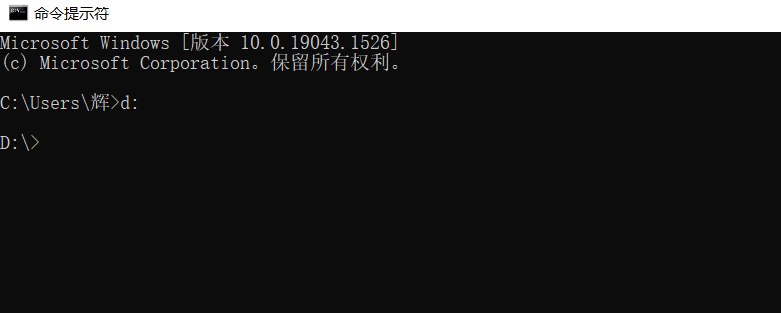
再进入HMMER文件夹中,输入
cd HMMER
先对PF00170(bZIP_1)处理,输入命令
hmmbuild PF00170.hmm PF00170_seed.txt

再输入命令,搜索毛果杨中含有该结构域的蛋白序列
hmmsearch PF00170.hmm Ptrichocarpa_protein.fasta > PF00170.out
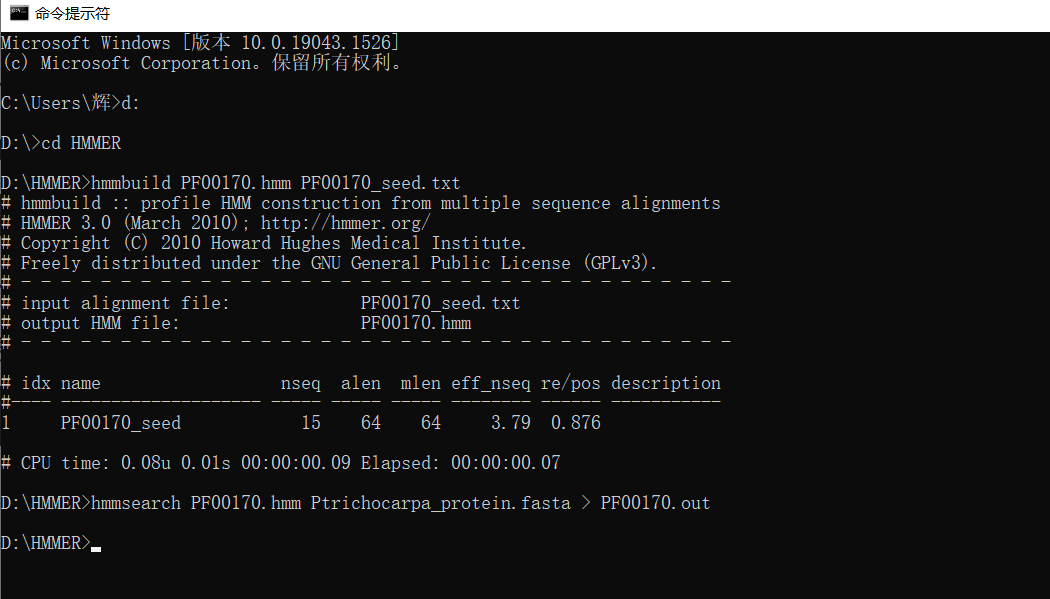
2秒算完,回到HMMER的文件夹中,发现了一些中间文件和PF00170.out的结果文件,可用excel打开处理。
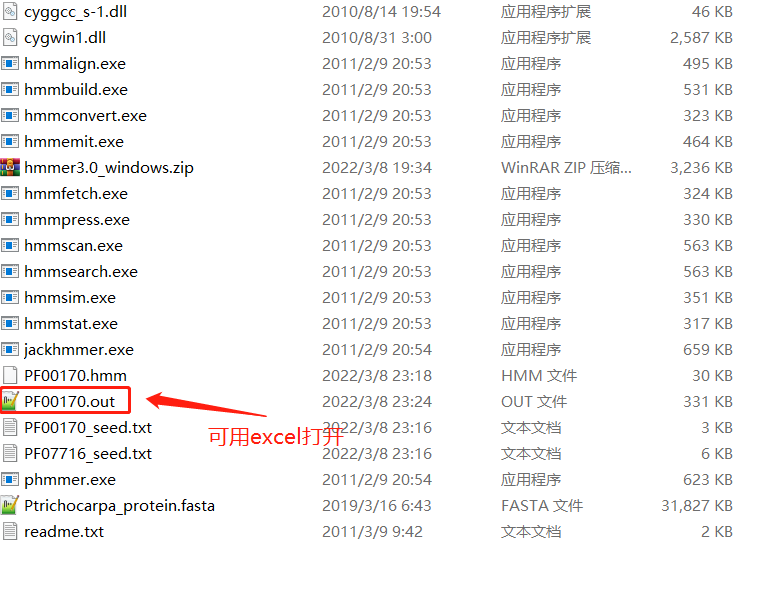
PF07716(bZIP_2)也是同样的操作。
具体的结果分析,自行百度即可,网上的教程很多。


 下面讲如何在windows系统上用HMMER做基因家族的鉴定
下面讲如何在windows系统上用HMMER做基因家族的鉴定


【推荐】国内首个AI IDE,深度理解中文开发场景,立即下载体验Trae
【推荐】编程新体验,更懂你的AI,立即体验豆包MarsCode编程助手
【推荐】抖音旗下AI助手豆包,你的智能百科全书,全免费不限次数
【推荐】轻量又高性能的 SSH 工具 IShell:AI 加持,快人一步
· 震惊!C++程序真的从main开始吗?99%的程序员都答错了
· winform 绘制太阳,地球,月球 运作规律
· 【硬核科普】Trae如何「偷看」你的代码?零基础破解AI编程运行原理
· 上周热点回顾(3.3-3.9)
· 超详细:普通电脑也行Windows部署deepseek R1训练数据并当服务器共享给他人