R笔记1
gsub format
> measurements<-c('3.95*3.99*2.43mm','3*3*5mm','2*2*2mm')
> measurements
[1] "3.95*3.99*2.43mm" "3*3*5mm" "2*2*2mm"
> # 去掉mm后缀
> a<-gsub('mm','',measurements)
> a
[1] "3.95*3.99*2.43" "3*3*5" "2*2*2"
> library(plyr)
> ?strsplit
> # 用 * 将数据分隔开,并将列表型数据转换为数据框格式
> strsplit(a,'[*]')
[[1]]
[1] "3.95" "3.99" "2.43"
[[2]]
[1] "3" "3" "5"
[[3]]
[1] "2" "2" "2"
> res <-ldply(strsplit(a,'[*]'))
> # 重命名数据框
> names(res)<-c('L','W','H')
> # 更改各个变量的数据类型为数值型
> res
L W H
1 3.95 3.99 2.43
2 3 3 5
3 2 2 2
> str(res)
'data.frame': 3 obs. of 3 variables:
$ L: chr "3.95" "3" "2"
$ W: chr "3.99" "3" "2"
$ H: chr "2.43" "5" "2"
> res<- as.data.frame(sapply(res,FUN=as.numeric))
> head(res)
L W H
1 3.95 3.99 2.43
2 3.00 3.00 5.00
3 2.00 2.00 2.00
> str(res)
'data.frame': 3 obs. of 3 variables:
$ L: num 3.95 3 2
$ W: num 3.99 3 2
$ H: num 2.43 5 2
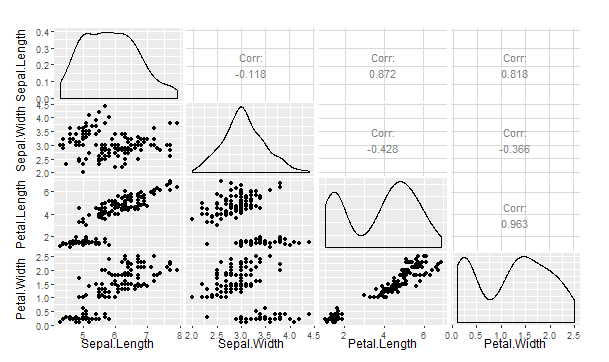
> #从定性角度,通过可视化来进行数据的探索性分析,强烈推荐使用GGally包中的ggpairs()函数,
> #该函数将绘制两辆变量的相关系数、散点图,同时也绘制出单变量的密度分布图
> library(ggplot2)
> library(GGally)
> head(iris)
Sepal.Length Sepal.Width Petal.Length Petal.Width Species
1 5.1 3.5 1.4 0.2 setosa
2 4.9 3.0 1.4 0.2 setosa
3 4.7 3.2 1.3 0.2 setosa
4 4.6 3.1 1.5 0.2 setosa
5 5.0 3.6 1.4 0.2 setosa
6 5.4 3.9 1.7 0.4 setosa
> ggpairs(iris[,1:4])
> ggsave("aaaaa.png")
Saving 8.46 x 5.03 in image
> memory.limit() #查看系统规定的内存使用上限
[1] 4077
> memory.limit(newlimit)#更改到新的上限
Error in memory.limit(newlimit) : object 'newlimit' not found
> #养成清理内存的习惯
> rm(object) #删除变量
Warning message:
In rm(object) : object 'object' not found
> gc() #在rm()后,记得使用gc()做garbage collection ,否则内存是不会释放的,相当于没有做rm()
used (Mb) gc trigger (Mb) max used (Mb)
Ncells 1330209 71.1 2164898 115.7 2164898 115.7
Vcells 6475711 49.5 10202170 77.9 8435142 64.4
> td=data.frame(c('2016-01','2016-02'),stringsAsFactors =F)
> td
c..2016.01....2016.02..
1 2016-01
2 2016-02
> td2<- sapply(td,paste,"-01",sep="")
> td2
c..2016.01....2016.02..
[1,] "2016-01-01"
[2,] "2016-02-01"
> as.Date(td2[,1]) #画图用
[1] "2016-01-01" "2016-02-01"
> format(as.Date(td2[,1]),"%Y-%m")
[1] "2016-01" "2016-02"
```
专注数据分析
欢迎转载并注明出处
```



