PCR Genotyping | 利用PCR来确定基因型 | Sanger sequencing | T7E1 | TIDE | NGS
2023年07月18日
用primer跑完PCR后,发现agarose gel有2-3个非特异性条带,于是切胶,用另一个protocol来提取gel里面的DNA。
最后一步失误地加入了PCR那一步的mixed primers(F+R),导致样本全废,只能从过滤膜里继续提取DNA了,5min提取出了3-5 mg/ul的DNA,提交了Sanger测序,结果还不错,NTC非常clean。
TIDE分析结果显示,gRNA附近有清晰的insertion和deletion,到此为止,实验成功。
后面就是功能验证,以及NGS测序,这个月我将有一大批data。
2023年07月14日
参考:A Survey of Validation Strategies for CRISPR-Cas9 Editing
常见的四种CRISPR效率验证方式:
- T7 endonuclease 1 (T7E1) mismatch detection assay - 不太靠谱,只能检测NHEJ
- Tracking of Indels by Decomposition (TIDE) assay - 性价比最高的Sanger测序
- targeted next-generation sequencing (NGS) - 金标准最贵
- Indel Detection by Amplicon Analysis (IDAA) assay
2023年07月05日
结果解读:
1. agarose条带,大部分是正常的,达到了送样标准;
2. Sanger结果,测序本身前50bp、后20bp很多N base,测序是不准的,所以你得primer不能太靠近gRNA,必须预留最少150-200bp;
结果就是要为gRNA重新设计primer,确保其有足够的距离(200bp);
如何设计primer,参见:引物设计 | Primer design
然后结果可以用一些工具来处理:
- https://ice.synthego.com/#/ - PB推荐
- https://tide.nki.nl SANGER - Paul
- http://crispresso2.pinellolab.org/submission - NGS - Paul
PCR目标区域DNA扩增流程:
- Trypsinizing Cells,取50000-100000 cells;
- 离心,500-1000rcf for 5min,丢掉培养液,注意不要丢掉细胞沉淀cell pellet;
- 加入试剂盒里的dilution buff和DNA release additive,室温2min,98度5分钟,spin,取上清液supernatant 1ul;
- 加入2.5 ul primer混合液,再加入25ul PCR master mix;
- 加入dw至50 ul;
- 上机,选Paul - Conoly PCR vector-specific TD65 1kb;
- 配置Agarose gel,看目标长度250-450是否有band,有就可以送样测序了;
- 取出2ul PCR product + 13ul dilute printer (5ul F primer + 250ul dw), 送去Dropbox盒子,ddl 7PM;
所需试剂:
- Gibco™ Trypsin-EDTA (0.25%), phenol red
- Thermo Scientific™ Phire Tissue Direct PCR Master Mix
- genewiz
primer配方:
- 在IDT oligos管里直接溶解,Double distilled water NF,管子上标记多少,就加入x10倍的dw溶解;
- 然后配置混合液,10ul F + 10ul R + 80ul dw = 100ul,等于稀释了10倍;【可以直接用于qRT-PCR】
- 然后取2.5ul mix,准备PCR,计算如下C1xV1 = C2xV2,C1=10,C2=0.5,V2=50,所以V1=2.5;
Quick Agarose protocol:
- Agarose LE 1.5g
- TAE buffer 150 ml
- Wait 1-2min
- Add SYBR safe dye for gel (1:10000) 15ul
- Plug in comb, cool down for 10 min
- Add 5ul ladder (purple one), 5ul PCR product
- 120-150V for 20-35min
- Check bottom Yellow band (should reach the bottom)
前面已经利用了PCR做了一些核心的实验:
- 目标区域扩增
- 实时定量mRNA
这里的目的还是目标区域扩增,不过material不再是细菌DNA,而是human cancer cell line的DNA(只需要几千细胞即可)。
Kits和protocol:
- 2X Phire Tissue Direct PCR Master Mix 【中等级别DNA提纯方案,仅适用于genotyping单个基因】
- Direct PCR on Hair: A New Animal-Friendly Genotyping Method
- 740951.50 NucleoSpin® Blood 【最高纯度的DNA提取方案,可直接用于WGS测序,Broad在用】
uM = micromole
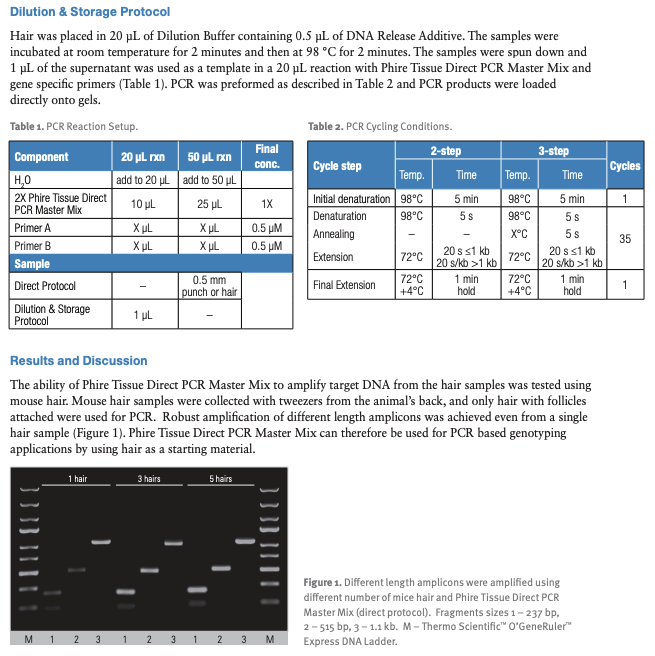
注意:
- Primer的保存浓度是一定的,10nmol + 100ul dw = 0.1 nM/uL,0.5 uM / 0.1 = 5 uL;
- 不要直接放入dirty cells,最好使用Dilution & Storage Protocol,98 °C for 5 minutes;
然后run一下agar gel,看一下目标区域是否有条带;
然后就可以送样,做sanger sequencing,第二天就能得到结果了。

 浙公网安备 33010602011771号
浙公网安备 33010602011771号