genome browser | ggplot | 基因组可视化 | R | transcript | isoform
2021年08月24日 更新
给每个exon编排序号,方便管理
1 | cat gencode.v37.annotation.gtf | grep ENSG00000011304 | cut -f1-5 | grep exon | sort -k 4 | uniq | less -S |
更新:又用了一段时间,发现是真的好用,可惜就是服务器上装不上,只能在Mac上用了。
核心代码很少:
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 | library(ggbio)# hg19library(EnsDb.Hsapiens.v75)ensdb <- EnsDb.Hsapiens.v75options(repr.plot.width=15, repr.plot.height=9)autoplot(ensdb, GeneNameFilter("PKM")) + # ME of exon 9 and 10 geom_vline(xintercept = c(72492996,72494795), linetype="dashed", color = "red", size=0.5) + theme_bw()# hg38library(EnsDb.Hsapiens.v86)ensdb <- EnsDb.Hsapiens.v86options(repr.plot.width=15, repr.plot.height=9)autoplot(ensdb, GeneNameFilter("PKM")) + # ME of exon 9 and 10 geom_vline(xintercept = c(72200655,72202454), linetype="dashed", color = "red", size=0.5) + theme_bw() |
版本问题
ensembl版本列表:https://asia.ensembl.org/info/website/archives/assembly.html
NCBI上的基因都有hg19和hg38两个版本的坐标,可以辅助使用。
只搞基因表达还没啥,现在搞到了isoform,即exon级别,想要随时查看底层的数据就很困难了。
比如下面就是AS分析的结果,想要手动去检查数据,完全不知所措。

一个AS event的命名:
1 | isoform1=junction:chr11:237145-244160:+|isoform2=junction:chr11:237145-238997:+@exon:chr11:238998-239076:+@junction:chr11:239077-244160:+ |
里面都是具体的基因组位置了,UCSC Genome Browser和IGV都无法胜任,因为无法标明单个碱基的位置。
这时候就发现了一个神器,ggbio: An R implementation for extending the Grammar of Graphics for Genomic Data
http://bioconductor.org/packages/release/bioc/html/ggbio.html
https://github.com/tengfei/ggbio
另一个发现:spliceclust
下面描述其具体用法:
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 | library(ggbio)library(Homo.sapiens)class(Homo.sapiens)data(genesymbol, package = "biovizBase")wh <- genesymbol[c("PTBP1")]wh <- range(wh, ignore.strand = TRUE)p.txdb <- autoplot(Homo.sapiens, which = wh)p.txdboptions(repr.plot.width=8, repr.plot.height=4)p.txdbautoplot(Homo.sapiens, which = wh, label.color = "black", color = "brown", fill = "brown")autoplot(Homo.sapiens, which = wh, gap.geom = "chevron")autoplot(Homo.sapiens, which = wh, stat = "reduce")--------------------------------------------------library(EnsDb.Hsapiens.v75)ensdb <- EnsDb.Hsapiens.v75autoplot(ensdb, GeneNameFilter("PTBP1"))options(repr.plot.width=10, repr.plot.height=6)autoplot(ensdb, GeneNameFilter("PTBP1"))autoplot(ensdb, ~ symbol == "PTBP1", names.expr="gene_name")autoplot(ensdb, ~ symbol == "PSMD13", names.expr="gene_name")--------------------------------------------------## We specify "*" as strand, thus we query for genes encoded on both strandsgr <- GRanges(seqnames = 11, IRanges(238998, 239076), strand = "+")autoplot(ensdb, GRangesFilter(gr), names.expr = "gene_name")options(repr.plot.width=15, repr.plot.height=9)autoplot(ensdb, GRangesFilter(gr), names.expr = "gene_name") + #geom_vline(xintercept = c(237145,244160), linetype="dashed", color = "blue", size=0.5) + #geom_vline(xintercept = c(237145,238997), linetype="dashed", color = "red", size=0.5) + geom_vline(xintercept = c(238998,239076), linetype="dashed", color = "green", size=0.5) + #geom_vline(xintercept = c(239077,244160), linetype="dashed", color = "black", size=0.5) + theme_bw() |
最后的图片:
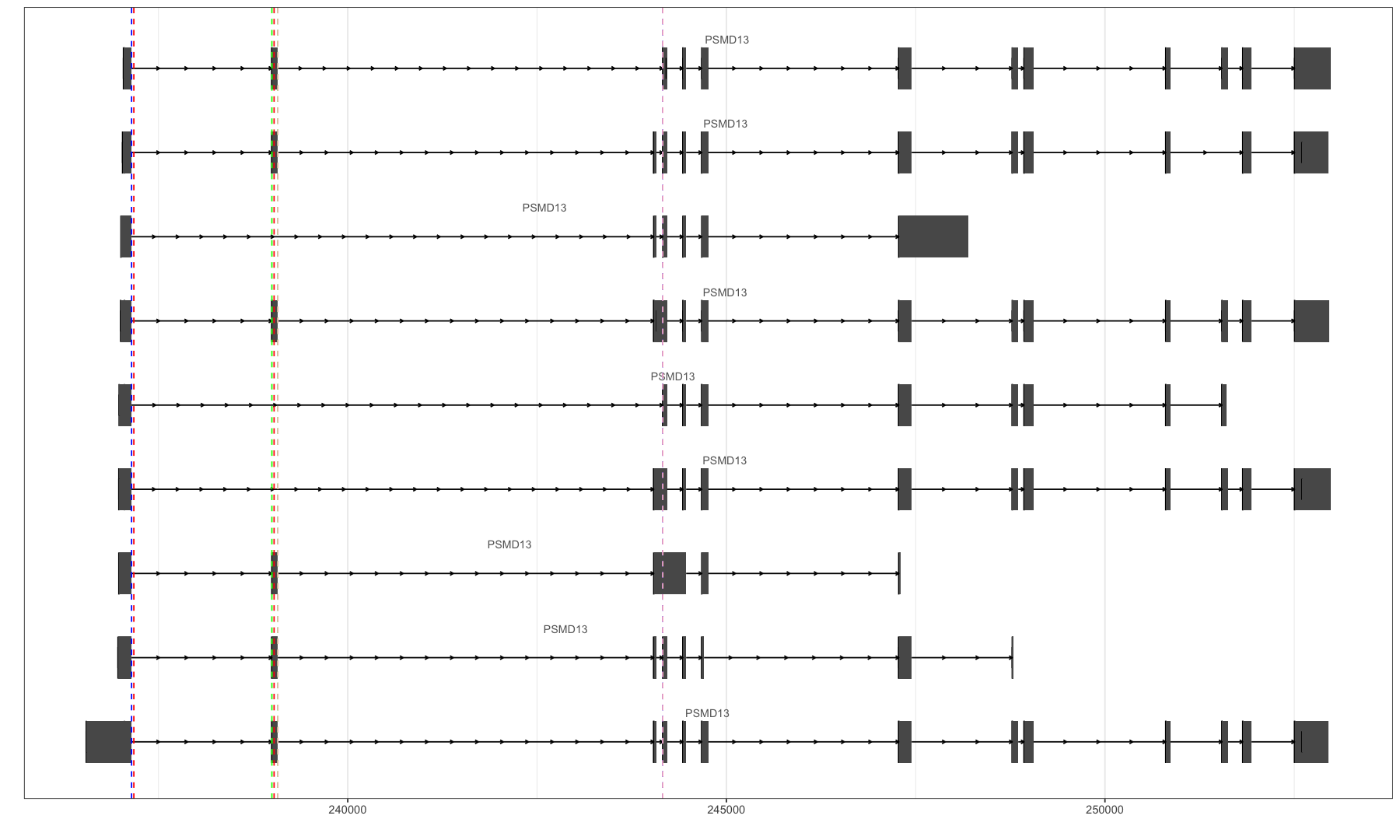
稍微搞清楚了的概念:
junction:从一个exon的结束到另一个exon的起点,这就是一个junction,俗称跳跃点、接合点,因为这两个点注定要连在一起。
AS event:一个可变剪切的事件,如何定义呢?一个exon,以及三个junction即可定义,要满足等式相等原则【一个完整的junction = 一个sub junction + exon + 一个sub junction】,即一个exon的inclusion/exclusion。
可视化的威力真是无穷,直观明了的揭示了数据之间的关系,这是肉眼直接看数据所做不到的!!!


【推荐】国内首个AI IDE,深度理解中文开发场景,立即下载体验Trae
【推荐】编程新体验,更懂你的AI,立即体验豆包MarsCode编程助手
【推荐】抖音旗下AI助手豆包,你的智能百科全书,全免费不限次数
【推荐】轻量又高性能的 SSH 工具 IShell:AI 加持,快人一步
· 基于Microsoft.Extensions.AI核心库实现RAG应用
· Linux系列:如何用heaptrack跟踪.NET程序的非托管内存泄露
· 开发者必知的日志记录最佳实践
· SQL Server 2025 AI相关能力初探
· Linux系列:如何用 C#调用 C方法造成内存泄露
· 震惊!C++程序真的从main开始吗?99%的程序员都答错了
· 【硬核科普】Trae如何「偷看」你的代码?零基础破解AI编程运行原理
· 单元测试从入门到精通
· 上周热点回顾(3.3-3.9)
· winform 绘制太阳,地球,月球 运作规律