3DUNet的Pytorch实现
编辑日期: 2021-04-24 16:57:48
本文主要介绍3DUNet网络,及其在LiTS2017肝脏肿瘤数据集上训练的Pytorch实现代码。
GitHub地址:
https://github.com/lee-zq/3DUNet-Pytorch
LiTS2017数据集 链接:
https://pan.baidu.com/s/1WgP2Ttxn_CV-yRT4UyqHWw
提取码:hfl8 (+*+||...==''。。。*_)
------------------------
2020.04.24更新:
- 删除了train_faster.py方法;
- 增加了只分割肝脏(不分割肿瘤)的设置方法;
- 参考其他文献,修改了训练和测试集的分配方式;
- 改进了预处理过程中的数据降采样方式
------------------------
一.3DUNet简介
最近重新整理了一下关于3DUNet网络原理及代码,这个网络其实和2DUNet区别不大,简单说可以理解为2d卷积换为了3d卷积。整体上没有什么创新,但可以基于一套完整的3DUNet代码(包括预处理、训练、可视化、测试等等)可以简化很多工作,在此基础上实现更多的细节改进,比如设计替换最新模块等等。对比下图中的2dUNet和3dUNet网络结构:
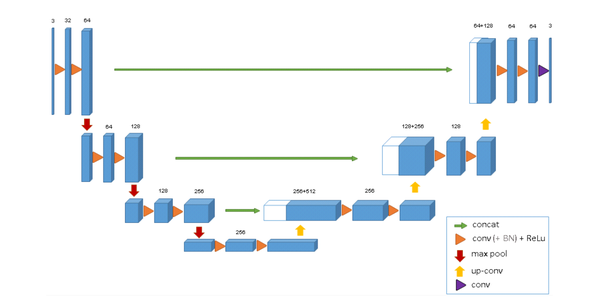
3DUNet网络结构
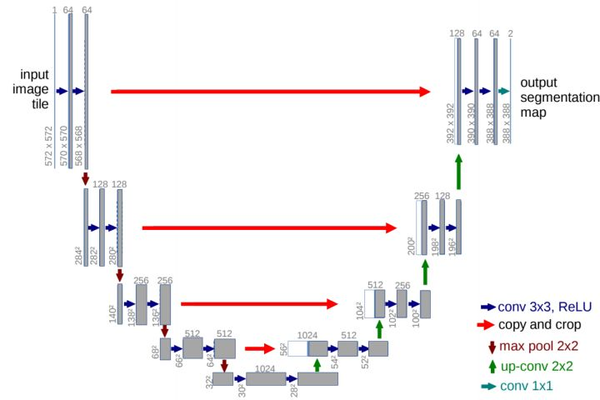
2DUNet网络结构
除了一些超参数设置不同,以及2d和3d卷积的区别,两者设计思路几乎完全一样。所以在网络结构上没啥要说的。
二. 3DUNet的Pytorch实现
本文的3DUNet代码主要参考了这个项目(here),修改了一些bug并进行了代码重构和梳理。可以直接访问下面的github仓库链接download并按照readme步骤使用:
https://github.com/lee-zq/3DUNet-Pytorch
在这里我也再梳理一下代码结构和设计思路,以及使用方法。
- 准备工作
首先下载代码:
git clone https://github.com/lee-zq/3DUNet-Pytorch.git
下载的代码结构和对应的功能如下:
│ .gitignore
│ config.py # 超参数配置
│ README.md # 使用方法介绍
│ train.py # 模型训练与验证函数 (主函数)
│ test.py # 针对每个测试样本分patch进行推理并拼接为分割结果
│
├─dataset
│ │ dataset_lits_train.py # 对LiTS数据集设计的dataset类,训练时调用
│ │ dataset_lits_test.py # 对LiTS数据集设计的dataset类,测试时调用
├─models
│ │ Unet.py # 2D、3DUNet网络模型
│ ├─nn # 网络公用模块存放位置,目前还未更新
│ ├── module.py # 一些CNN中的常用模块
│
├─output # trained models 都保存在这里面
│ readme.md
│
│ preprocess_LiTS.py # 针对LiTS2017数据集设计的预处理方案,主要对原始数据集进行初步处理,和项目内其他代码完全隔离。如格式转换、切片提取等等
│
├─utils # 其他模块
│ common.py # 常用函数包
│ weights_init.py # 初始化相关包
│ logger.py # 日志生成函数
│ metrics.py # 评价指标
└ loss.py # 损失函数
然后从文章开头的百度云链接下载LiTS2017数据集。
此外还有python环境配置要求:
pytorch >= 1.1.0
torchvision
SimpleITK
Tensorboard
Scipy
由上面的项目结构基本就可以知道代码运行的步骤了,如下:
2. 预处理步骤
将下载的LiTS数据集解压至任一目录(例如./raw_dataset/),并将batch1和batch2中的数据进行分配,这里建议将(2746)共20个样本作为测试集,将(026和47~131)共111个样本作为训练集。然后分别将训练集和测试集里面的volum数据和segmentation数据分别放入该目录下的data文件夹和label文件夹(若计算资源有限或测试代码,可先提取一部分数据)。如下:
raw_dataset:
├── train # (27~46)共20个样本
│ ├── data
│ │ ├── volume-27.nii
│ │ ├── volume-28.nii ...
│ └── label
│ ├── segmentation-27.nii
│ ├── segmentation-28.nii ...
│
├── test # 0~26和47~131)共111个样本
│ ├── data
│ │ ├── volume-0.nii
│ │ ├── volume-1.nii ...
│ └── label
│ ├── segmentation-0.nii
│ ├── segmentation-1.nii ...
然后在 ./preprocess_LiTS.py文件中更改预处理输入、输出目录:
raw_dataset_path = './raw_dataset/train/' # 输入数据集路径
fixed_dataset_path = './fixed_data/' # 预处理后的数据集的输出路径
执行 python ./preprocess_LiTS.py ,运行完后在 fixed_dataset_path='./fixed_data/' 目录下可生成如下内容:
│ train_name_list.txt # 训练集文件名 list
│ val_name_list.txt # 验证集文件名 list
│
├─data # 预处理后的data数据
│ volume-0.nii
│ volume-1.nii
│ volume-2.nii
│ ...
└─label # 预处理后的标签数据
segmentation-0.nii
segmentation-1.nii
segmentation-2.nii
...
到这里,数据预处理完成。预处理的内容可通过源代码了解,我也加了中文注释。
3. 模型训练
首先在 config.py中修改超参数--dataset_path为我们预处理后的数据根目录 :./fixed_data ,其他参数也可以根据注释自行修改。当然你也可以在下面运行训练命令的时候指定参数去覆盖默认参数。
然后运行 python train.py --save model_name
这样训练即开始,到指定迭代次数,就会在output/model_name下生成保存的模型文件和日志文件,你可以通过 tensorboard --logdir ./output/model_name在浏览器查看训练过程中dice和loss。
训练模型时,大概率会出现GPU利用率不满/跳变的情况,这时建议使用 train_faster.py替换 train.py进行训练,可以实现从一个输入样本提取一个batch进行训练(通过调用dataset_lits_faster.py 实现),这样速度会快很多,但可能导致收敛较慢。 2020.04.24更新,已将train_faster.py方法删除,因为会降低精度。而且数据加载速度可以用dataloader类的num_workers提高,从而提高GPU利用率,进而提高训练速度。 PS:设置非0的num_workers参数有时会在Windows系统下报错,而linux下不会
PS:一些问题(To Do List)
- 采用的数据集包含三类标签:背景、肝脏、肝肿瘤。肿瘤附着在肝脏上,体积很小。所以,直接按照三类来进行训练,会导致肿瘤分割效果较差,这个可以通过实验结果验证。所以对此类问题我们一般都是先分割或检测肝脏得到肝脏ROI,然后在此ROI内完成肿瘤分割。
- 模型分割结果还可以通过连通域分析等后处理策略,进一步提高分割结果的准确性
- 保存模型的机制还需要改进,后续会增加保存验证指标最高和最新epoch得到的模型。
参考:
- https://github.com/panxiaobai/lits_pytorch
- https://zhuanlan.zhihu.com/p/104854615
- https://github.com/jeya-maria-jose/KiU-Net-pytorch
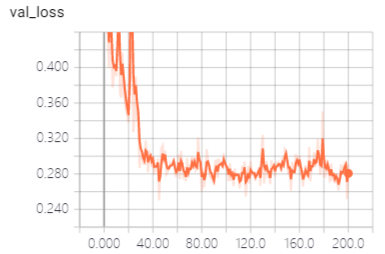
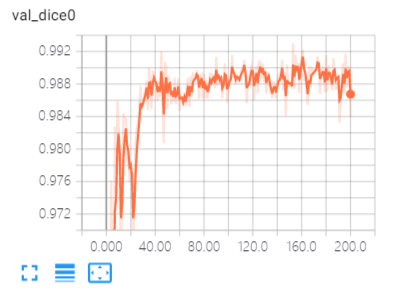
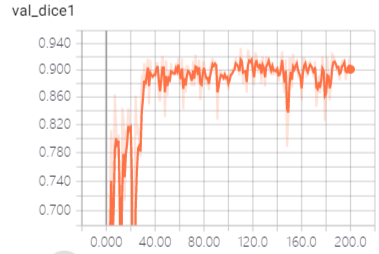
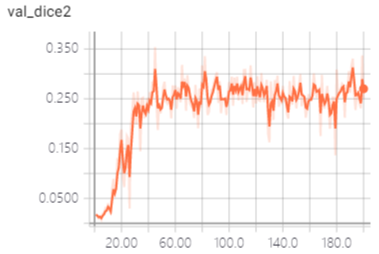
一些相关截图分享,验证结果可视化:





【推荐】国内首个AI IDE,深度理解中文开发场景,立即下载体验Trae
【推荐】编程新体验,更懂你的AI,立即体验豆包MarsCode编程助手
【推荐】抖音旗下AI助手豆包,你的智能百科全书,全免费不限次数
【推荐】轻量又高性能的 SSH 工具 IShell:AI 加持,快人一步
· 全程不用写代码,我用AI程序员写了一个飞机大战
· MongoDB 8.0这个新功能碉堡了,比商业数据库还牛
· 记一次.NET内存居高不下排查解决与启示
· DeepSeek 开源周回顾「GitHub 热点速览」
· 白话解读 Dapr 1.15:你的「微服务管家」又秀新绝活了