pipeline pilot(BIOVIA Discovery Studio)基础用法
搬运来源: https://blog.csdn.net/weixin_43364556/article/details/106780358
pipeline piplot就是相当于一堆元器件,我们可以用这些元器件组成各种各样的电子产品。
分类
pipeline piplot中左侧的工具栏中分为三个部分(见左下部分),第一个部分为用户的自定义的工具(也就是用户自己组装好的电子产品,这一部分以用户的名称进行命名)。第二部分为DS中常用的流程(命名为Protocols),第三部分为各种组件的名称,不同的组件完成的功能不同(相当于各种元器件,命名为Components)。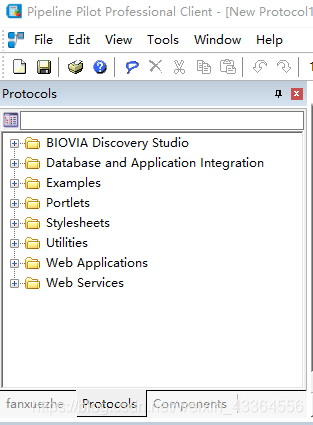
Chemistry
Chemistry中的组件的主要功能是跟化学相关的(如下图),比如分子读取,文库设计,文件格式的转化(比如二维转化为三维的格式),分子的操作(分子的筛选,比如lipinski五规则,相似性的搜索等),化合物聚类(基于结构)等等。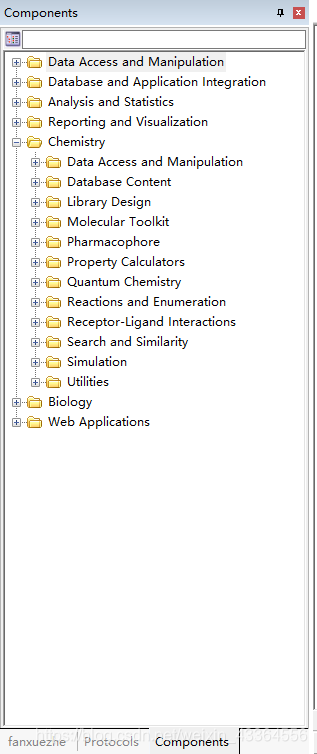
案例
参考文献:
A combinatory screen of the CLOUD uncovers a synergy targeting the androgen receptor.
文献是通过FDA通过的化学分子药物,希望通过这些分子的组合或者其他方式找出其不同的发挥功能的方向。文献收集了308个小分子药物,The CeMM Library of Unique Drugs (CLOUD)是一个整合了前体药物以及在药用中的活性形式,适合用来进行药物配对的筛选,找出能杀死癌细胞的药物组合。最后文献发现两种药物的组合(flutamide(氟他胺) and phenprocoumon (PPC:苯丙香豆素))能够稳定癌细胞的AR受体,并且使前列腺癌细胞的AR受体对氟他胺变得更加敏感。
文章首先通过手机FDA通过的药物整合成一个数据库,剔除大分子,提取通过配体受体方式相互作用的药物,剔除盐离子,最后通过聚类剔除冗余的分子(剔除me too药物,因为结构和作用方式相似)。作者根据化学结构进行聚类,并且提出其中位于中心位置的分子药物。最后从2万多个药物中提取出176类,239个代表性的药物。另外,通过查阅文献找出其中的35个前体药物,把这些前体药物的活化形式加入库中。另外加入其它34个靶标未知的分子,形成最后的CLOUD库,含有308个分子。最后再利用高通量的方法进行筛选。
流程实例:读取chemDraw格式文件并且转化为3D格式
一、搜索目标模块
直接在component模块的左上方的搜索数据框中进行搜索。比如找出能够读取chemDraw分子结构文件的读取程序,直接搜索reader(这个应该与经验有关,用的次数多了应该会更熟练一些)会显示出与reader相关的组件。writer/filter的模块也是同样的操作。在这里我们找出chemDraw reader,双击其将其添加到protocal框中(双击自动添加,不影响原始文件)。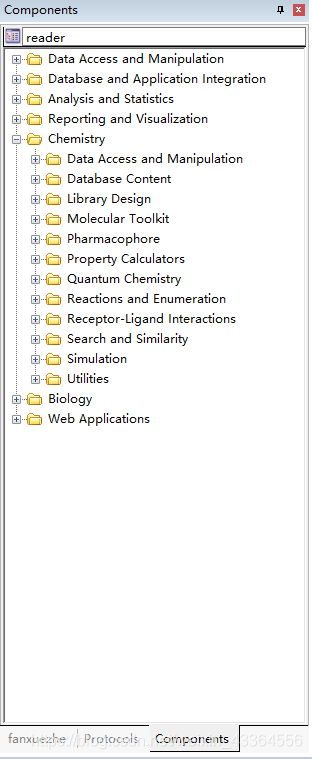
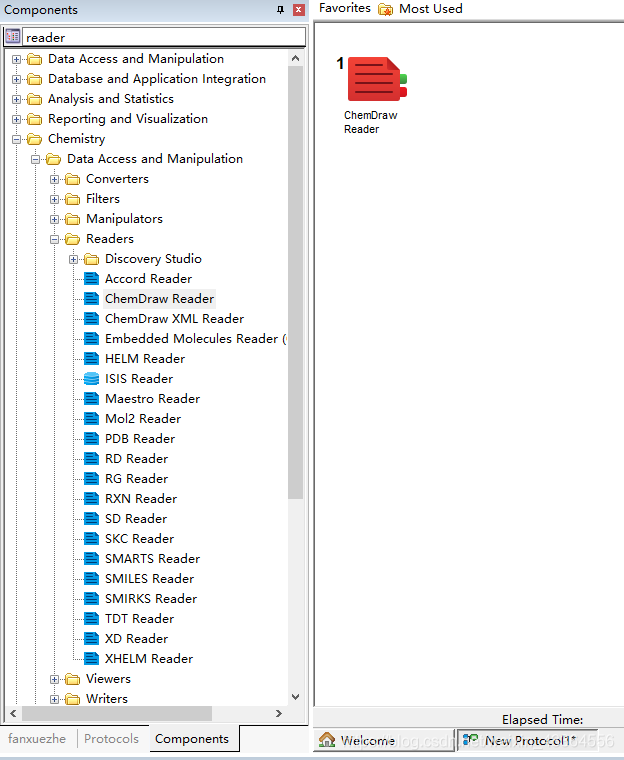
可以看到刚刚添加的组件是红色的,是因为一些必要的参数没有设定,该模块无法正常工作。我们将其中需要的参数进行设置之后就可以正常运行了(变成了蓝色)。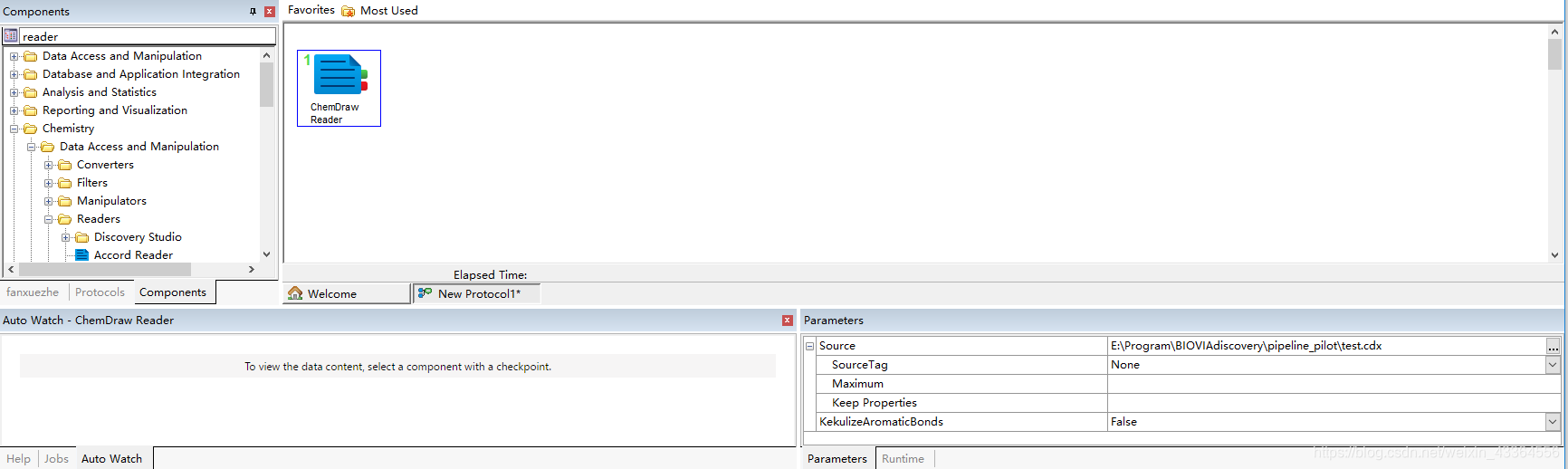
这里的文件设成自己文件就可以,路径也要带上。我随便做了一个,可以在这里下载https://pan.baidu.com/s/14kD9A9dU8FKLDJMUI-EbxQ,提取码fa0v。
二、添加3D coods模块
3D coods模块能够将二维的数据转化为三维的坐标数据。直接双击,就能够将这个模块加到之前的模块上。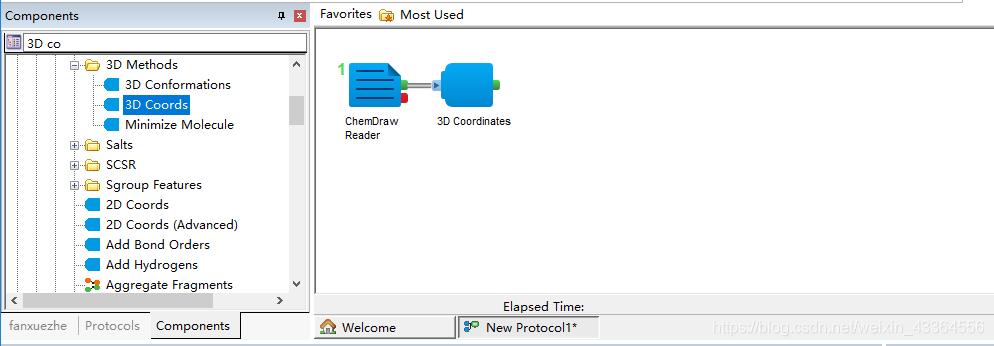
三、添加H原子(add Hydrogens)
原来的系统不含有H原子,这里添加H原子到系统中。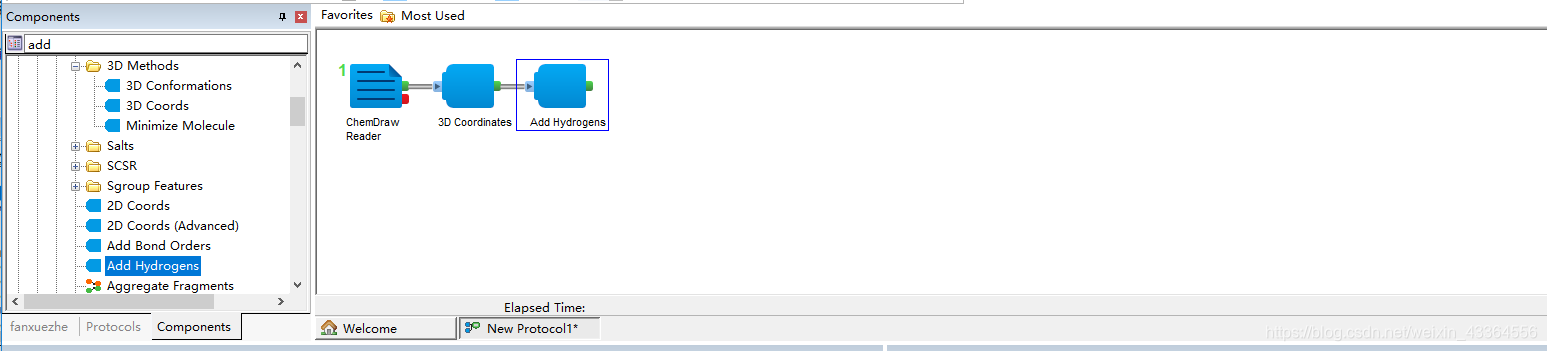
四、保存为SD格式文件(SD writer)
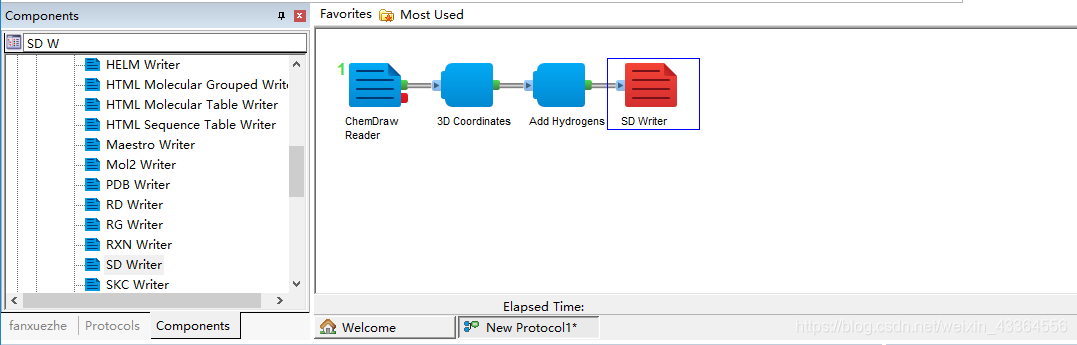
修改参数(指定文件的保存位置),使其变为蓝色正常状态。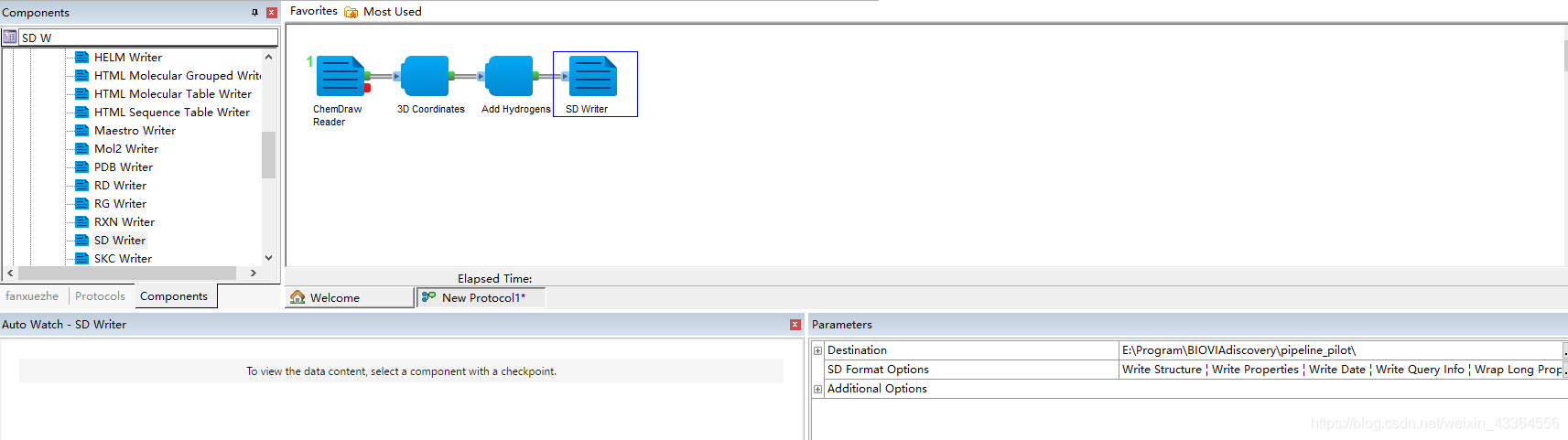
五、运行
点击上方的开始按钮,整套系统开始运行。假如系统报错那么久根据错误的类型来进行修改,比如我这里久报错说找不到目标文件,因为系统只能识别服务器上的文件,而不能识别本地机器上的文件。那么我就将文件上传到服务器,然后修改路径,再次运行。运行成功能看到绿色的小1。注意,我们也可以通过路径参数旁边的三个点号来选择路径。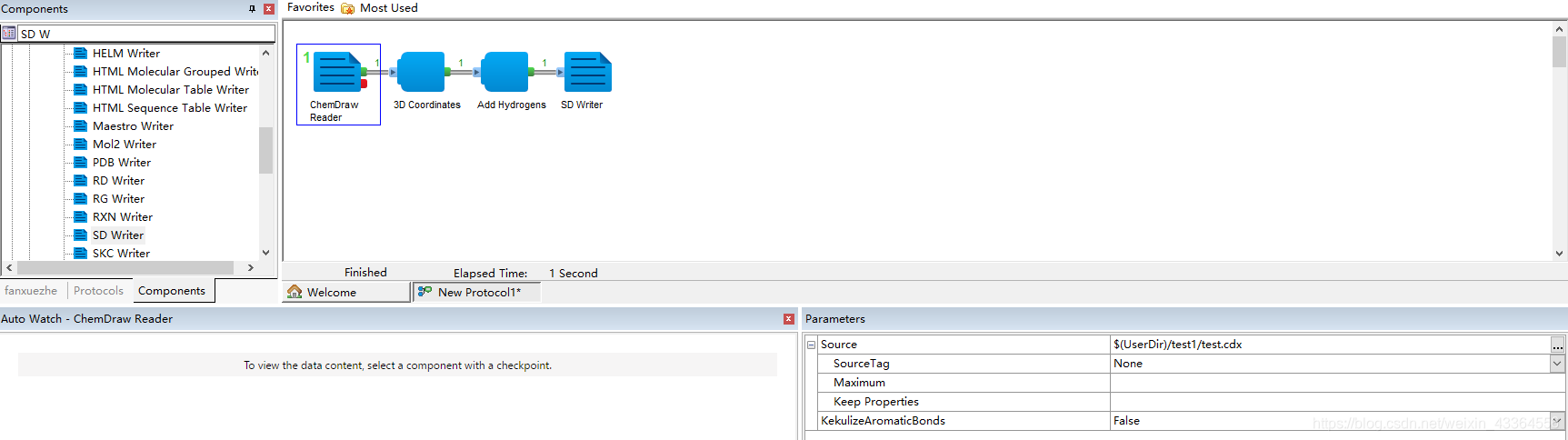




【推荐】还在用 ECharts 开发大屏?试试这款永久免费的开源 BI 工具!
【推荐】国内首个AI IDE,深度理解中文开发场景,立即下载体验Trae
【推荐】编程新体验,更懂你的AI,立即体验豆包MarsCode编程助手
【推荐】轻量又高性能的 SSH 工具 IShell:AI 加持,快人一步