scikit-learn使用PCA降维小结
本文在主成分分析(PCA)原理总结和用scikit-learn学习主成分分析(PCA)的内容基础上做了一些笔记和补充,强调了我认为重要的部分,其中一些细节不再赘述。
Jupiter notebook版本参见我的github: https://github.com/konatasick/machine_learning_note/blob/master/pca.ipynb
PCA的思想
PCA(Principal components analysis,主成分分析)是一种降维算法,它通过使样本间方差尽量大来尽可能保留原始数据的相关关系。
PCA的算法
1) 对所有的样本进行中心化 2) 计算样本的协方差矩阵 3) 对协方差矩阵进行特征值分解 4)取出最大的m个特征值对应的特征向量, 将所有的特征向量标准化后,组成特征向量矩阵W。 5)对样本集中的每一个样本转化为新的样本
scikit-learn的sklearn.decomposition.PCA参数介绍
官方文档:sklearn.decomposition.PCA
Parameters:
n_components:这个代表了需要降维的维度。当它是整数时,代表了保留的维度数量。当它是一个介于0~1之间的数时,代表了主成分的方差所占的最小比例,例如0.95代表取超过95%的量的维度。当它为‘mle’,同时svd_solver == ‘full’时,系统会根据MLE算法自动选择维度。(此时svd_solver == ‘auto’将会被解读为svd_solver == ‘full’)
svd_solver:默认是‘auto’,即在剩下的‘full’, ‘arpack’, ‘randomized’中根据情况选一个。‘full’是传统的PCA,‘arpack’, ‘randomized’适用于数据量大的场景,其区别在于前者是通过scipy.sparse.linalg.svds实现。
Attributes*:
components_ : 主成分的投影坐标,代表了数据的最大方差的方向,根据explainedvariance由大到小排列。维度是m*n,其中n是原始数据的维度,m是降维后的维度。
explainedvariance和explained_varianceratio:前者是每一维的方差,后者是所占比例:
explained_varianceratio=explained_variance/sum(explainedvariance)
维度是m, 当m=n时,sum(explained_varianceratio)=1。
mean_:每个feature的平均值。在pca算法的第一步,需要对feature归一化,此时的平均值保留在这里。
ncomponents :模型实际的降维数,即m。
PCA实例
用scikit-learn学习主成分分析(PCA)中示范了降维的操作。
首先我们生成随机数据并可视化,代码如下:
import numpy as np import matplotlib.pyplot as plt from mpl_toolkits.mplot3d import Axes3D %matplotlib inline from sklearn.datasets.samples_generator import make_blobs # X为样本特征,Y为样本簇类别, 共1000个样本,每个样本3个特征,共4个簇 X, y = make_blobs(n_samples=10000, n_features=3, centers=[[3,3, 3], [0,0,0], [1,1,1], [2,2,2]], cluster_std=[0.2, 0.1, 0.2, 0.2], random_state =9) fig = plt.figure() ax = Axes3D(fig, rect=[0, 0, 1, 1], elev=30, azim=20) plt.scatter(X[:, 0], X[:, 1], X[:, 2],marker='o')
输出如图:
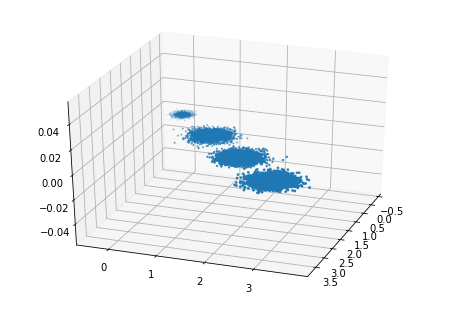
现在我们来进行降维,从3维降到2维,代码如下:
from sklearn.decomposition import PCA pca = PCA(n_components=2) pca.fit(X) X_new = pca.transform(X) plt.scatter(X_new[:, 0], X_new[:, 1],marker='o') plt.show()
输出如图:

在很多应用中,当我们将数据降维并用于训练后,训练出来的模型之后的输出也是降维后的数据,需要还原回原始维度。这时候需要将pca算法进行逆运算:
X_old=np.dot(Xnew,pca.components)+pca.mean_
即将新数据和components_相乘并加上平均值。
使用上文的例子,代码如下:
X_old=np.dot(X_new,pca.components_)+pca.mean_ fig = plt.figure() ax = Axes3D(fig, rect=[0, 0, 1, 1], elev=30, azim=20) plt.scatter(X_old[:, 0], X_old[:, 1], X_old[:, 2],marker='o')
输出如图:

可以看到,数据即是投影到最大方差方向但并未进行降维时的样子。
*parameter的命名后面没有下划线,而attribute的命名后面都有下划线,以此区分。



