REST easy with kbmMW #17 – Database 6 – Existing databases
kbmMW已经包含了非常精细的功能来确定和解释数据库中表的元数据。
在下一版本中,这个功能将得到进一步加强,可以导入现有数据库中的表,自动创建与表相匹配的ORM实体类。
这意味着你能够使用kbmMW的ORM智能来查询表,将数据保存回表并操纵表元数据,例如添加,更改或删除字段、索引以及需要能够升级数据库用于匹配已更改的ORM表实体类的更高版本的表。
当你意识到需要更改数据库表以允许其他或不同的字段时,ORM的设计目标之一始终是抽象掉所需的所有枯燥工作。在kbmMW中,它就像更改ORM类一样简单,并使用kbmMW的ORM来创建表的CreateOrUpgrade。这已经在早期的REST相关文章中介绍过。
在下一个即将发布的kbmMW版本中,ORM本身实现了生成Delphi类的代码能力,该代码与ORM实体类对应的任何表相匹配。由于ORM已经支持分析现有的表结构以确定现有的ORM类是否与实际的表结构不同,为了能够自动升级它,它是下一个逻辑步骤,能够使用有关表结构的知识和元数据生成全新的数据库表Delphi实体类。
ImportDBSchema工具实现了ORM实体类的Delphi代码生成,现在我来演示这个工具的用法:
因为复杂的部分已经在kbmMW的内部实现,所以该工具的代码就变的非常简单了。基本上它使用FireDAC支持的任何数据库,包括:SQLite,MSSQL,MySQL,Oracle,Interbase / Firedac和PostgreSQL,用户可以从连接的数据库中选择一个或多个表,然后调用kbmMW的ORM相关代码,最终生成这些表的实体类代码。
表格表示kbmMW语言,从表中建立足够的元数据信息,以便能够使用kbmMW的ORM注册TkbmMWORMTable,启用查询等,而无需实际拥有ORM类。
但由于TkbmMWORMTable包含kbmMW的ORM运行所需的所有元数据,因此它也可以用作生成新ORM表类的源。ORM类显然使您可以非常轻松地将表的字段和记录作为具有属性的常规对象进行访问。
事实上,导入包括生成ORM类的表,归结为调用这两个ORM方法:
var tbl:TkbmMWORMTable; begin ... tbl:=MyORM.SurfaceDynamicTable('actualdbtablename',true); MyORM.GenerateDelphiClass(tbl...); ... end;
让我们尝试利用一个MySQL数据库导入一个表。
在这种情况下,我们使用匿名用户名在服务器ensembldb.ensembl.org上选择名为aedes_aegypti_core_48_1b的数据库。
可以在此页面上找到连接信息:https://www.ensembl.org/info/data/mysql.html
单击“连接到数据库”将打开FireDAC对话框,在该对话框中设置数据库的连接信息:

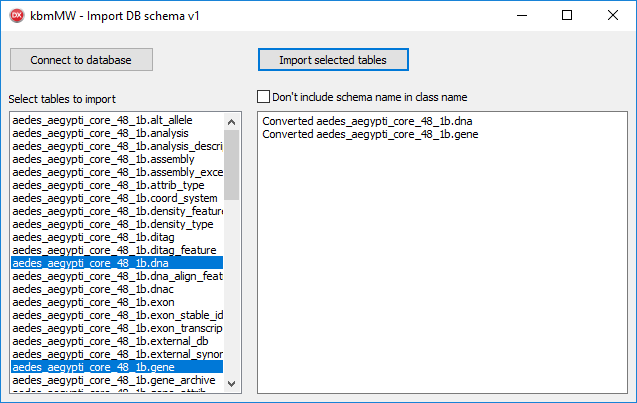
单击“确定”,将显示该数据库中的所有表的列表,选择其中一些表,然后单击“导入所选表”。
现在,系统会提示我们输入单元文件名,用于保存生成的实体类。
由于我们选择了两个表,因此该单元将包含两个类。单击“确定”后,将开始导入。可能需要一段时间。
让我们看看生成的单元,我称之为ensembl.pas
unit ensembl; interface uses DB, Math, Classes, System.Generics.Collections, kbmMWGlobal, kbmMWObjectMarshal, kbmMWNullable, kbmMWORM, kbmMWRTTI; type [kbmMW_Table('name:aedes_aegypti_core_48_1b.dna')] TAedes_aegypti_core_48_1b_dna = class private FSeq_region_id:integer; FSequence:string; public [kbmMW_Field('name:seq_region_id, primary:true', ftInteger)] [kbmMW_NotNull] property Seq_region_id:integer read FSeq_region_id write FSeq_region_id; [kbmMW_Field('name:sequence', ftWideMemo)] [kbmMW_NotNull] property Sequence:string read FSequence write FSequence; end; [kbmMW_Table('name:aedes_aegypti_core_48_1b.gene')] TAedes_aegypti_core_48_1b_gene = class private FGene_id:integer; FBiotype:string; FAnalysis_id:word; FSeq_region_id:integer; FSeq_region_start:integer; FSeq_region_end:integer; FSeq_region_strand:byte; FDisplay_xref_id:kbmMWNullable<integer>; FSource:string; FStatus:kbmMWNullable<string>; FDescription:kbmMWNullable<string>; FIs_current:byte; public [kbmMW_Field('name:gene_id, primary:true', ftInteger)] [kbmMW_NotNull] property Gene_id:integer read FGene_id write FGene_id; [kbmMW_Field('name:biotype', ftString, 40)] [kbmMW_NotNull] property Biotype:string read FBiotype write FBiotype; [kbmMW_Field('name:analysis_id', ftSmallInt)] [kbmMW_NotNull] property Analysis_id:word read FAnalysis_id write FAnalysis_id; [kbmMW_Field('name:seq_region_id', ftInteger)] [kbmMW_NotNull] property Seq_region_id:integer read FSeq_region_id write FSeq_region_id; [kbmMW_Field('name:seq_region_start', ftInteger)] [kbmMW_NotNull] property Seq_region_start:integer read FSeq_region_start write FSeq_region_start; [kbmMW_Field('name:seq_region_end', ftInteger)] [kbmMW_NotNull] property Seq_region_end:integer read FSeq_region_end write FSeq_region_end; [kbmMW_Field('name:seq_region_strand', ftShortInt)] [kbmMW_NotNull] property Seq_region_strand:byte read FSeq_region_strand write FSeq_region_strand; [kbmMW_Field('name:display_xref_id', ftInteger)] property Display_xref_id:kbmMWNullable<integer> read FDisplay_xref_id write FDisplay_xref_id; [kbmMW_Field('name:source', ftString, 20)] [kbmMW_NotNull] property Source:string read FSource write FSource; [kbmMW_Field('name:status', ftWideString, 19)] property Status:kbmMWNullable<string> read FStatus write FStatus; [kbmMW_Field('name:description', ftWideMemo)] property Description:kbmMWNullable<string> read FDescription write FDescription; [kbmMW_Field('name:is_current', ftShortInt)] [kbmMW_NotNull] property Is_current:byte read FIs_current write FIs_current; end; implementation initialization TkbmMWRTTI.EnableRTTI([TAedes_aegypti_core_48_1b_dna, TObjectList<TAedes_aegypti_core_48_1b_dna>, TAedes_aegypti_core_48_1b_gene, TObjectList<TAedes_aegypti_core_48_1b_gene>]); kbmMWRegisterKnownClasses([TAedes_aegypti_core_48_1b_dna, TObjectList<TAedes_aegypti_core_48_1b_dna>, TAedes_aegypti_core_48_1b_gene, TObjectList<TAedes_aegypti_core_48_1b_gene>]); end.
您可能会注意到,每个类名都包含架构/数据库名称和表名称。如果您只想将表名作为类名的一部分,请在导入之前选中“不要在类名中包含模式名称”复选框。
在一些相当罕见的情况下,数据库表可能已经定义了某些kbmMW未知的数据类型。在这种情况下,导入仍将发生,但有问题的字段将使用UNKNOWN数据类型进行标记。
kbmMW还尝试确定其他索引和音序器信息,并生成与之匹配的相关属性。然而,如果推断这些类型的信息失败,那么它将被忽略优先级,它将被忽略,因此继续生成基本的ORM表类。
现在,通过将此单元添加到您的应用程序,并设置MySQL kbmMW连接池以指向我们用于导入的相同(或类似)服务器,我们现在可以使用ORM轻松地从这两个表中查询数据。
var lst:TObjectList<TAedes_aegypti_core_48_1b_dna>; begin lst:=MyORM.QueryList<TAedes_aegypti_core_48_1b_dna>([2000],mwoqoGT); ... end;
在上面的示例中,我们将获得大于2000的主键的TAedes_aegypti_core_48_1b_dna对象列表。
如果对列表中的任何对象进行了任何更改,则可以通过以下方式轻松将更改解析回数据库
MyORM.Persist(lst);
使用ORM,使用这两个表的正确结构创建我们自己的数据库也非常容易。数据库甚至不必是基于MySQL的数据库。它可能是kbmMW的ORM支持的任何一个。
MyOtherORM.CreateOrUpgradeTable([TAedes_aegypti_core_48_1b_dna,TAedes_aegypti_core_48_1b_gene])
关于如何以一种新的现代面向对象方式轻松地重用现有数据库,这就结束了这一点。
如果您喜欢我所展示的内容,请通过分享我们的博客和艺术品来传播关于kbmMW的信息。
https://components4developers.blog/2018/11/15/rest-easy-with-kbmmw-17-database-6/


 浙公网安备 33010602011771号
浙公网安备 33010602011771号