层次聚类
目录
1.层次聚类的原理
2.层次聚类优缺点
3.聚类实例
1.层次聚类的原理
层次聚类可以分为两种主要类型:凝聚型(agglomerative)和分裂型(divisive)。
1.凝聚聚类:它也被称为AGNES(凝聚嵌套)。 它以自下而上的方式工作。 也就是说,每个对象最初被认为是单元素簇(叶子)。 在算法的每个步骤中,将最相似的两个群集组合成新的更大的群集(节点)。 迭代此过程,直到所有点都只是一个单个大簇(root)的成员(参见下图)。 结果是一棵树,可以绘制为树状图。
2.分裂层次聚类:它也被称为DIANA(Divise Analysis),它以自上而下的方式工作。 该算法是AGNES的逆序。 它以root开头,其中所有对象都包含在单个集群中。 在迭代的每个步骤中,最异构的集群被分成两个。 迭代该过程,直到所有对象都在它们自己的集群中(见下图)。
其中,凝聚聚类擅长识别小聚类。 分裂层次聚类擅长识别大型集群。

然而,我们如何衡量两组观测之间的差异? 已经开发了许多不同的聚类聚集方法(即连接方法)来回答这个问题。 最常见的类型方法是:
- 最大或完全链接聚类(Maximum or complete linkage clustering):它计算聚类1中元素和聚类2中元素之间的所有成对不相似性,并将这些不相似性的最大值(即最大值)视为两个聚类之间的距离。 它倾向于产生更紧凑的簇。
- 最小或单链接聚类(Minimum or single linkage clustering):它计算聚类1中的元素和聚类2中的元素之间的所有成对不相似性,并将这些不相似性中的最小值视为链接标准。 它倾向于产生长而“松散”的簇。
- 平均或平均链接聚类(Mean or average linkage clustering):它计算聚类1中的元素与聚类2中的元素之间的所有成对不相似性,并将这些不相似性的平均值视为两个聚类之间的距离。
- 质心连锁聚类(Centroid linkage clustering):它计算聚类1的质心(长度为p变量的平均向量)与聚类2的质心之间的不相似性。
- Ward的最小方差方法(Ward’s minimum variance method):它最小化了整个群内方差。 在每个步骤中,合并具有最小簇间距离的一对簇。
2.层次聚类优缺点
优点:
- 距离和规则的相似度容易定义,限制少
- 不需要预先制定聚类数
- 可以发现类的层次关系
- 对大样本数据效果较好
缺点:
- 计算复杂度太高
- 奇异值也能产生很大影响
- 算法很可能聚类成链状
3.聚类实例
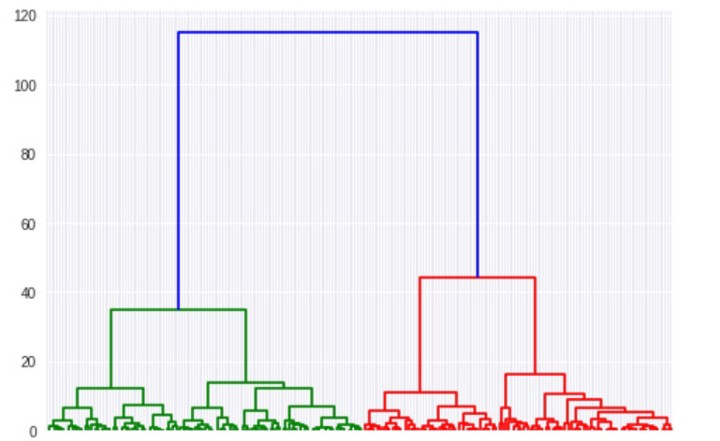
树形图绘制出每个簇和距离。 我们可以使用树形图找到我们选择的任何数字的聚类。 在上面的树形图中,很容易看到第一个簇(蓝色),第二个簇(红色)和第三个簇(绿色)的起点。 这里只对前3个进行了颜色编码,但是如果你查看树形图的红色边,你也可以发现第4个簇的起始点。 树形图一直运行,直到每个点都是它自己的单个簇。
让我们看看凝聚层次聚类如何在Python中运行。 首先,让我们从scipy.cluster.hierarchy和sklearn.clustering导入必要的库。
# import statements from sklearn.datasets import make_blobs import numpy as np import matplotlib.pyplot as plt # create blobs data = make_blobs(n_samples=200, n_features=2, centers=4, cluster_std=1.6, random_state=50) # create np array for data points points = data[0] # create scatter plot plt.scatter(data[0][:,0], data[0][:,1], c=data[1], cmap='viridis') plt.xlim(-15,15) plt.ylim(-15,15) plt.show()
现在,让我们创建我们的树形图(我已经在上面展示过),确定我们想要多少个簇,并保存这些簇中的数据点以将它们绘制出来。
#define distance using euclidean disMat = sch.distance.pdist(points,'euclidean') #define the linkage_matrix using ward clustering pre-computed distances linkage_matrix=sch.linkage(disMat, method ='ward') #optonal :average ward etc fig, ax = plt.subplots(figsize=(15, 20)) # set size ax = sch.dendrogram(linkage_matrix, orientation="right") #, labels=titles);#可添加label plt.tick_params(\ axis= 'x', # changes apply to the x-axis which='both', # both major and minor ticks are affected bottom='off', # ticks along the bottom edge are off top='off', # ticks along the top edge are off labelbottom='off') plt.tight_layout() #show plot with tight layout
这段代码显示的图像要比上边的图像更美观,更清晰,由于样本数较多,就不在此展示了。
还可以利用散点图展示聚类的结果:
from sklearn.cluster import AgglomerativeClustering # create clusters hc = AgglomerativeClustering(n_clusters=4, affinity = 'euclidean', linkage = 'ward') # save clusters for chart y_hc = hc.fit_predict(points) plt.scatter(points[y_hc ==0,0], points[y_hc == 0,1], s=100, c='red') plt.scatter(points[y_hc==1,0], points[y_hc == 1,1], s=100, c='black') plt.scatter(points[y_hc ==2,0], points[y_hc == 2,1], s=100, c='blue') plt.scatter(points[y_hc ==3,0], points[y_hc == 3,1], s=100, c='cyan')

也可以展示热力图:
import seaborn as sns sns.clustermap(points,method ='ward',metric='euclidean')
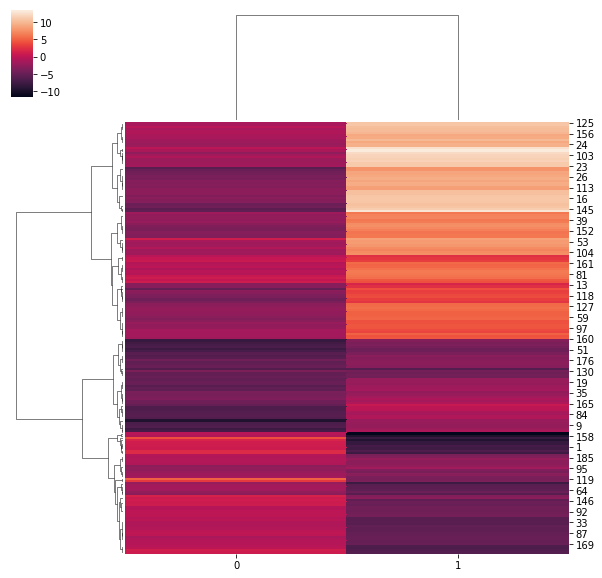
参考:
https://blog.csdn.net/qq_19528953/article/details/79133889(距离公式详解)
https://blog.csdn.net/enigma_tong/article/details/79081449(聚类函数参数详解)




