Leetcode 187. 重复的DNA序列 哈希
地址 https://leetcode-cn.com/problems/repeated-dna-sequences/
所有 DNA 都由一系列缩写为 'A','C','G' 和 'T' 的核苷酸组成,例如:"ACGAATTCCG"。在研究 DNA 时,识别 DNA 中的重复序列有时会对研究非常有帮助。
编写一个函数来找出所有目标子串,目标子串的长度为 10,且在 DNA 字符串 s 中出现次数超过一次。
示例 1:
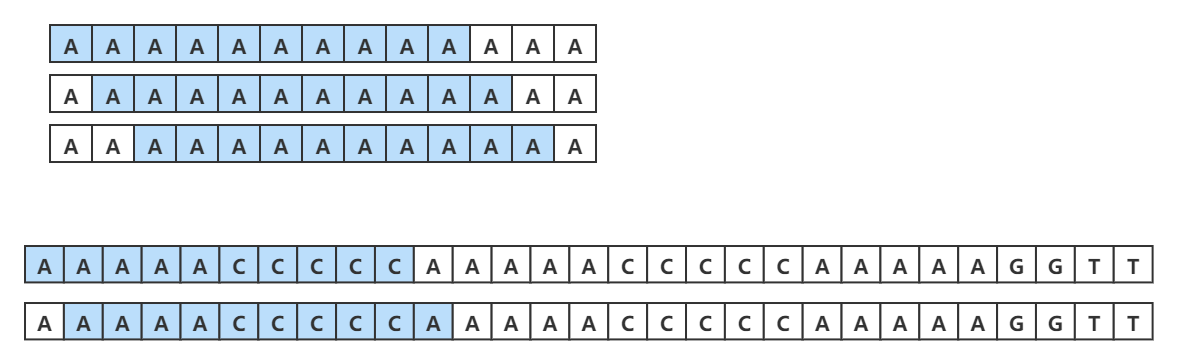
输入:s = "AAAAACCCCCAAAAACCCCCCAAAAAGGGTTT"
输出:["AAAAACCCCC","CCCCCAAAAA"]
示例 2:
输入:s = "AAAAAAAAAAAAA"
输出:["AAAAAAAAAA"]
提示:
0 <= s.length <= 105
s[i] 为 'A'、'C'、'G' 或 'T'
解答
需要获取字符串中每个连续10个字符的出现次数
那么使用哈希记录 查找也比较快速
代码
class Solution {
public:
vector<string> findRepeatedDnaSequences(string s) {
unordered_map<string,int> mm;
vector<string> ans;
if(s.size()<10) return ans;
for(int l = 0; l<s.size()-9;l++){
string subs = s.substr(l,10);
if( mm[subs]==1) { ans.push_back(subs); }
mm[subs]++;
}
return ans;
}
};
作 者: itdef
欢迎转帖 请保持文本完整并注明出处
技术博客 http://www.cnblogs.com/itdef/
B站算法视频题解
https://space.bilibili.com/18508846
qq 151435887
gitee https://gitee.com/def/
欢迎c c++ 算法爱好者 windows驱动爱好者 服务器程序员沟通交流
如果觉得不错,欢迎点赞,你的鼓励就是我的动力


欢迎转帖 请保持文本完整并注明出处
技术博客 http://www.cnblogs.com/itdef/
B站算法视频题解
https://space.bilibili.com/18508846
qq 151435887
gitee https://gitee.com/def/
欢迎c c++ 算法爱好者 windows驱动爱好者 服务器程序员沟通交流
如果觉得不错,欢迎点赞,你的鼓励就是我的动力




 浙公网安备 33010602011771号
浙公网安备 33010602011771号