Brain Full-featured, real-time database searching platform enables fast and accurate multiplexed quantitative proteomics (功能齐全的实时数据库搜索平台可实现快速,准确的多重定量蛋白质组学)【分享人:翁海玉】
文献名:Brain Full-featured, real-time database searching platform enables fast and accurate multiplexed quantitative proteomics (功能齐全的实时数据库搜索平台可实现快速,准确的多重定量蛋白质组学)
期刊名:Journal of Proteome Research
发表时间:(2019年6月)
IF:3.78
单位:
- Department of Cell Biology, Harvard Medical School, Cambridge, MA
- University of Washington Proteomics Resource, Seattle, WA
- Thermo Scientific LSMS, San Jose, CA
研究对象: 构建功能齐全的实时数据库搜索平台
技术:SPS-MS3
一、 概述:(用精炼的语言描述文章的整体思路及结果)
复杂蛋白质组定量方法(SPS-MS3)是最准确的定量方法,但采集工作周期较长。本文建立了一个新的实时数据库搜索(RTS)平台Orbiter,以应对SPS-MS3方法更长的工作周期。
Orbiter的RTS可以消除SPS-MS3扫描的没有肽段匹配给定的光谱。利用Orbiter的在线蛋白质组学分析管道(包括RTS和错误发现率分析),可以在不到10毫秒的时间内处理单个频谱数据库搜索。使得Comet匹配过滤肽谱更快速,有效,并且更有效地量化目标蛋白质。而且,使用Comet的肽谱匹配可以进行全特征搜索,包括翻译后修饰的分析,并给出众所周知且广泛验证的评分。
在这项工作中,作者测试了该自适应数据采集平台的实用性,以提高多重定量实验的效率和准确性。发现RTS使质谱数据采集效率提高了2倍。Orbiter的RTS能够在一半的SPS-MS3分析时间(RTS为18小时,SPS-MS3为36小时)中量化10个蛋白质组中的8000多种蛋白质。
二、 研究背景:(简要介绍研究进展动态、研究目的和意义)
多重定量方法不断尝试平衡采集速度和前体分离纯度。使用多重同位素试剂的初步实验实现了基于HRMS2方法进行多重定量分析,并依靠单个前体分离来尝试消除共分离离子。尽管这些方法相对较快,但由于母离子的共分离,所得的定量结果受到子离子共分离干扰现象的困扰。
为了消除干扰,开发了SPS-MS3方法(采用第三次扫描来分析子离子)。
SPS-MS3方法大大改善定量准确性,但每个扫描周期添加第三次定量扫描减慢了仪器采集速度。虽然有其他方法可以减少前体共隔离干扰和/或增加速度,这些方法仍依赖于HRMS2方法或SPS-MS3方法。
最近一个验证原理显示,实时频谱匹配作为一种新颖的手段来实现以HRMS2的速度分析并拥有SPS-MS3的定量准确度。实时搜索(RTS)有潜力大大提高多重定量分析的采集效率,在有且只有肽谱匹配(PSM)时才允许定量。这种智能采集策略是基于一个二项式检索分值。通过将该策略专门应用于多重分析并通过SPS-MS3选择性消除,本研究证明了扫描采集速度的巨大提升和多重定量的准确性改善。
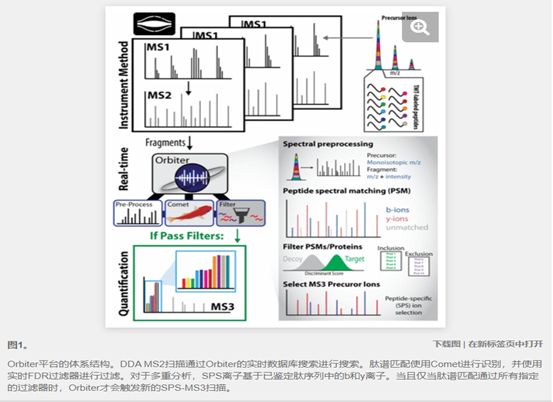
在当前的工作中,我们扩展了RTS策略在多重定量蛋白质组学中的鲁棒性和灵活性。我们构建了称为Orbiter 的完整分析流程-单同位素峰优化,数据库搜索和FDR过滤,这些都以毫秒为单位。Orbiter分析的速度使实时决策能够仅在观察到PSM时才对采集SPS-MS3扫描。我们选择了开源的彗星搜索引擎,数据库检索和评分。对于这项工作,对Comet进行了修订,以实现快速的实时光谱分析,同时保持对高度灵活的搜索的支持(例如,翻译后修改,多个同位素包络和灵活的碎片方案)。我们评估了Orbiter RTS相对于标准SPS-MS3的改进性能。与标准的SPS-MS3方法相比,Orbiter的采集速度提高了2倍,并提高了定量精度。
三、 实验方法:
构建Orbiter平台
在获取SPS-MS3之前执行RTS并仅在观察到PSM时才触发SPS-MS3扫描,可以显着减少生成的伪SPS-MS3扫描的数量。此过程将节省仪器时间来获取更多与MS2扫描相匹配的肽段,从而增加蛋白质组覆盖率,进而触发有用的SPS-MS3扫描。我们构建了Orbiter平台来执行此RTS决策。Orbiter包含功能齐全的分析蛋白质组学流水线,其运行速度足够快,可以与仪器扫描采集同时无缝集成并通知未来的扫描决策(即何时触发SPS-MS3)。
Orbiter平台内置于C#(.NET 4.6.5+)中,可在MS2扫描采集的毫秒内执行光谱预处理,使用Comet搜索引擎的RTS,SPS离子选择以及实时错误发现率过滤(图1)。三个主要组件实现了快速,自适应的数据库搜索。首先,在数据库搜索之前对前体MS1扫描进行处理有助于单同位素前体质量的分配和实时修正。其次,Comet数据库搜索算法的最新开发版本实现了快速单频谱搜索。第三,使用线性判别分析构建实时错误发现率(FDR)过滤,以使用用户定义的FDR设置过滤PSM。
四、研究成果:(重点图表展示)
1、Comet脱机运行时,Orbiter也能够在几毫秒内以高保真度在整个有机数据库快速搜索每个新的MS2光谱,比较SEQUEST离线搜索,我们发现高度相关的相似。
2、Comet的RTS修订版保留了可脱机使用的灵活数据库搜索功能,允许使用用户定义的变量修饰,即使在完整的人类数据库中,我们也观察到了中等的搜索时间,在50,000分辨率SPS-MS3扫描(86毫秒)的默认进样时间下,搜索时间下降得很好。
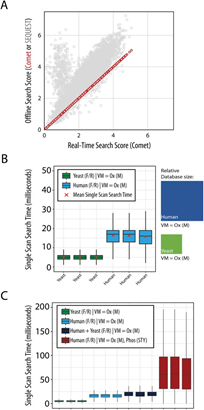
图2
3、LDA产生最终的判别分数,能够有效地将目标与诱饵的PSM分开。
4、为了确保该LDA不会产生偏差,我们在同一运行中比较了在线LDA和离线LDA。高度相关的LDA系数说明偏差很小。
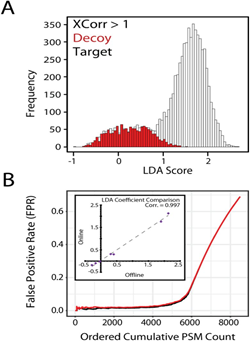
图3
5、SPS-MS3方法在36小时内定量了8455种蛋白质。在短短18小时内,Orbiter RTS方法量化了8166(SPS-MS3方法的97%)。从每小时用SPS-MS3定量的234种蛋白质增加到每小时用Orbiter RTS定量的454种蛋白质。SPS-MS3与Orbiter定量结果之间的比率高度相关(Pearson相关= 0.946)
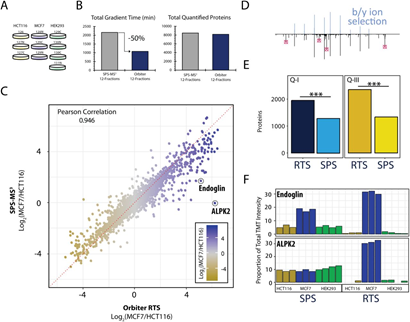
图4
五、文章亮点(结论讨论):
Orbiter平台在获取SPS-MS3之前执行RTS并仅在观察到PSM时才触发SPS-MS3扫描,大大节约仪器时间来获取更多与MS2扫描相匹配的肽段,从而增加蛋白质组覆盖率。在消除了共分离前体离子干扰的前提下,还使效率提高了一倍。
Orbiter包含功能齐全的分析蛋白质组学流水线,其运行速度足够快,可以与仪器扫描采集同时无缝集成并通知未来的扫描决策。
同时作者还对比了离线状态下的搜索匹配准确性,说明该方法在离线状态下也能很好地工作。充分考虑实际实验环境。
阅读人:翁海玉


 浙公网安备 33010602011771号
浙公网安备 33010602011771号