Journal of Proteome Research | SAAVpedia: identification, functional annotation, and retrieval of single amino acid variants for proteogenomic interpretation | SAAV的识别、功能注释和检索 | (解读人:徐洪凯)
文献名:SAAVpedia: identification, functional annotation, and retrieval of single amino acid variants for proteogenomic interpretation(SAAVpedia:蛋白质基因组解释的单氨基酸突变的识别、功能注释和检索)
期刊名:Journal of Proteome Research
发表时间:2019年10月
IF:3.78
单位:
- 韩国基础科学研究所,生物融合分析研究中心
- 韩国化学技术研究所,药物信息平台中心
- 韩国大田忠南国立大学,分析科学与技术研究所
物种:人
技术:蛋白基因组学,SAAVpedia
一、 概述:
新一代的基因组测序技术使得许多疾病或药物反应相关的非同义单核苷酸变异(Nonsynonymous Single Nucleotide Variants, nsSNVs)的发现成为可能,这些变异改变了蛋白质的氨基酸序列。目前已有研究试图描述致病的nsSNVs,但很少有研究在蛋白质水平上被确认为单氨基酸变异(Single Amino Acid Variants, SAAVs)。本研究开发了由四个模块(SAAVidentifier、SAAVannotator、SNV/SAAVretriever和SAAVvisualizer)组成的SAAVpedia平台,从蛋白质组和基因组数据中识别、注释和检索致病的SAAV候选。SAAVpedia在乳腺癌和恶性胶质瘤数据分析中的应用说明其在确认基因组变体是否存在于氨基酸序列中是一个比较合适的工具。
二、 研究背景:
得益于二代测序技术,大量的可能会引起蛋白功能变化的nsSNVs被鉴定到,目前已经有了许多对其致病机理和分子机制进行预测、解释的研究,但这些方法在不同数据集之间无法充分的复现。在一些研究中,结合蛋白质组数据在蛋白质水平上鉴定到的来自nsSNVs的变异,可能在实际的疾病发生过程中扮演重要的角色。但是由于没有开发出有关鉴定SAAV的综合蛋白基因组学分析平台,目前只有很少一部分SAAV能够与疾病相关的基因组数据和功能联系起来,大多数SAAV的生物、临床和药理功能仍然是未知的,进一步确认致病的SAAV是极其困难的。
三、 实验设计:
四、研究成果:
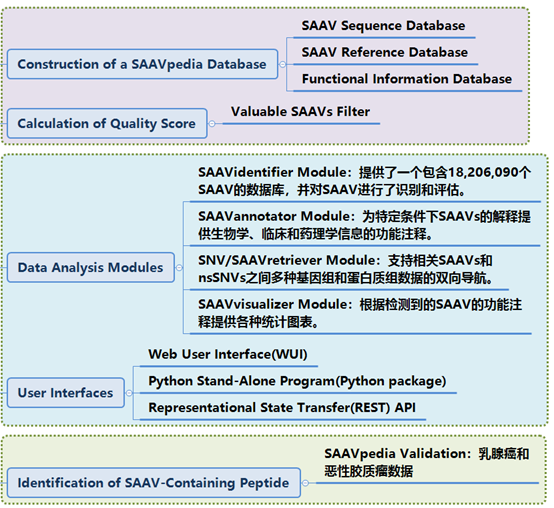
SAAVpedia是一个从蛋白质组和基因组数据中识别、注释和检索致病SAAV候选体的平台。该平台由四个模块组成:SAAVidentifier、SAAVannotator、SNV/SAAVretriever和SAAVvisualizer。SAAVidentifier提供了包含549,033个SAAVs的42,134个蛋白质亚型的参考数据库。SAAVannotator提供了24种信息,属于4个类别:i)基因组变异,ii)基因/转录本/蛋白,iii)生物学和iv)临床信息。它为解释特定条件下的SAAV提供了功能注释。SNV/SAAVretriever模块支持在相关条件特异性SAAVs和nsSNVs之间双向导航,具有多种基因组和蛋白组数据。最后,SAAVvisualizer根据检测到的SAAVs的功能信息提供了各种统计图。
SAAVpedia中包括三个数据库:SAAV序列数据库(SAAV Sequence Database)、SAAV参考数据库(SAAV Reference Database)以及功能信息数据库(Functional Information Database)。其中,序列数据库中包括来自neXtProt和GENCODE中所有含有变异肽段的蛋白质序列;参考数据库中包括neXtProt中全部SAAV的基因组和蛋白质组变异信息,基因组变异信息包括基因位置和来自dbSNP或COSMIC的基因组参考序列和改变的核苷酸序列,蛋白质组变异信息包括变异发生的位置,蛋白质参考序列和改变的氨基酸序列以及来自neXtProt的胰蛋白酶酶解肽段序列;功能信息数据库包括生物、临床、药理等信息。

图1. SAAVpedia平台的原理图模块。
该平台支持从蛋白质组和基因组数据中识别、注释和检索致病SAAVs。SAAVidentifier对SAAVs进行识别和质量评估。SAAVannotator通过对生物学、临床和药理信息进行注释,以解释SAAVidentifier已识别的SAAVs的功能。SNV/SAAVretriever模块自动构建具有基因组和蛋白质组变体列表的查询。它还可以双向导航相关条件特异性SAAVs和nsSNVs,具有多种表型的基因组和蛋白质组数据。SAAVvisualizer提供五类分析结果的图形可视化。除SAAVvisualizer模块外,所有模块都实现为Web应用程序、Python包和REST API。
利用CPTAC(Clinical Proteomic Tumor Analysis Consortium)乳腺癌研究中的925个LC-MS/MS数据以及Synapse中恶性胶质瘤样本的40个LC-MS/MS数据进行测试:
表1. CPTAC和NextPP搜索结果中乳腺癌和胶质母细胞瘤样本中鉴定到的SAAVs。
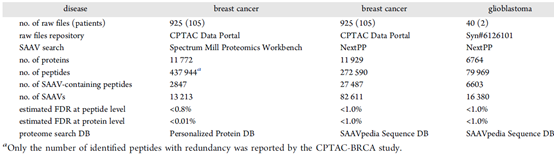

图2. 互作网络图。通过SAAVpedia结合临床信息和PPI网络的发现乳腺癌相关的新基因和致病的SAAV候选。PPI网络由乳腺癌相关基因和SAAVs基因组成。
在8,377个基因中对82,611个乳腺癌SAAVs进行鉴定、注释以及蛋白互作分析(PPI),最终构建的PPI网络由1326个基因节点(红色节点黄色文本)和1087个一级edge构成(A)。其中共有298个基因与BRCA1、TP53、BRCA2、ERBB2、ESR1、ESR2等乳腺癌相关基因有基本联系(B, C)。
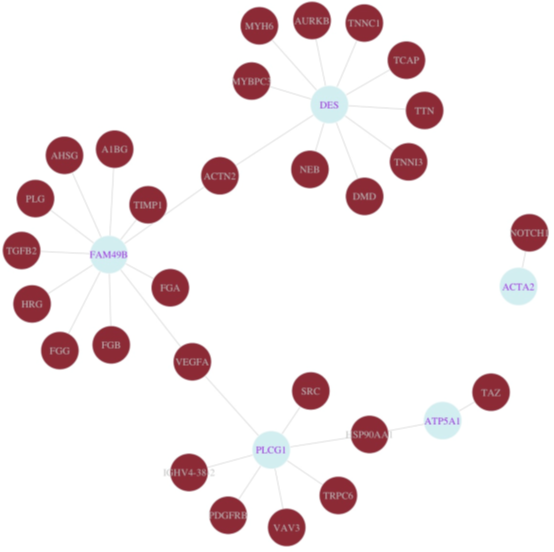
图3. 互作网络图。通过SAAVpedia结合临床信息和PPI网络的发现胶质母细胞瘤相关的新基因和致病的SAAV候选。PPI网络由胶质母细胞瘤相关基因和SAAVs基因组成。共有12个基因与胶质母细胞瘤驱动基因(红)相关。节点测试颜色为紫色,表示SAAV数量为1。
五、文章亮点(结论讨论):
SAAVpedia可以从大规模和高通量的基因组和蛋白质组数据中鉴定、注释和检索致病性SAAVs。SAAVpedia中灵活的蛋白质基因组解释流程可以帮助描述和定位实际条件特异性的致病SAAVs,而这些是不能通过比较和分析基因组和蛋白质组数据来预测的。SAAVpedia基于功能多样的生物医学知识数据库提供了足够的信息,但是由于所访问的知识库本身信息不足,一些查询并没有返回结果。通过使用知识数据库对功能信息数据库进行一致的扩展,可以解决这一问题。通过把SAAV数据库从各种蛋白质组数据扩展到进一步的生物医学研究,SAAVpedia能够在基于大量蛋白质基因组数据的致病生物标志物发现中发挥重要的作用。
阅读人:徐洪凯
原文链接:https://pubs.acs.org/doi/10.1021/acs.jproteome.9b00366
DOI:10.1021/acs.jproteome.9b00366



 浙公网安备 33010602011771号
浙公网安备 33010602011771号