Mol Cell Proteomics. | 用于鉴定新型融合转录本及其在癌细胞中的潜在翻译产物的多功能蛋白质组基因组学工具FusionPro
期刊:Molecular & Cellular Proteomics
发表时间:June 17, 2019
分享人:任哲
内容与观点:
大家好,本次分享的是发表在Molecular & Cellular Proteomics上的一篇关于蛋白质组基因组学工具的文章,题目是FusionPro, a Versatile Proteogenomic Tool for Identification of Novel Fusion Transcripts and Their Potential Translation Products in Cancer Cells,通讯作者是Yonsei University,Proteome Research Center的Young-Ki Paik教授,他长期从事蛋白质组基因组学的研究,发表了一系列蛋白质组学的工具流程和研究。
基因融合会产生各种融合的蛋白形式,FusionPro就是通过分析蛋白质组和转录组的数据来鉴定和注释相应融合蛋白形式的工具。在本文所述的研究中,研究者通过MS/MS数据来发现融合蛋白形式的证据,并通过FusionPro所获取的序列信息来分析融合蛋白的翻译模式。这个流程利用蛋白质组基因组策略鉴定来自融合基因翻译的肽段序列,并详细分析了致癌的基因融合形式。
基因融合现象一般很少发生,但是它的翻译产物,却在各种生物医学研究中发挥着重要的作用,比如,有些基因融合蛋白可能影响着肿瘤的发生发展,从而可以作为肿瘤标记物或者细胞周期的调控元件。尽管已经有很多研究者声称可以对基因融合及其翻译产物进行预测或者鉴定,但是从蛋白质组学水平进行研究的还鲜有报道。基于此,作者开发了FusionPro,它可以协助对基因融合转录本的构建和其翻译产物进行鉴定。利用该流程,作者成功地从白血病细胞系和卵巢癌组织中分别鉴定到了18个和3个融合导致的肽段,并成功地验证到了其中的两个蛋白融合形式和其翻译产物。此外,更深入的序列分析揭示了这些融合蛋白形式可能参与到了细胞循环过程。我们的结果也揭示了,融合基因通常具有多种融合的转录本,以及多种剪切位点,在染色体和基因水平相对随机。
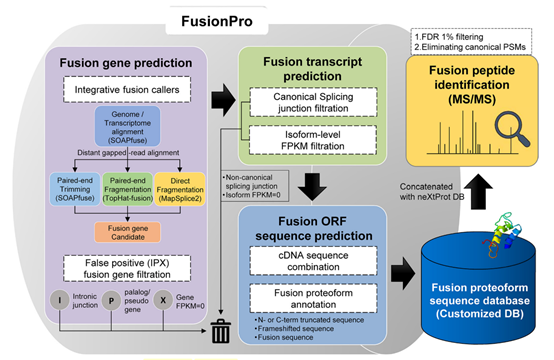
FusionPro共包含了8个模块,用于鉴定基因融合转录本和构建定制的数据库来搜索,它们分别是:CreateReference, RunSoapfuse, RunTophatfusionS, RunMapspliceS, RunCufflinks, ProcessResults, FindFusionProteoform, 和 MergeMultipleResults,各个模块之间详细的工作流程见下图:
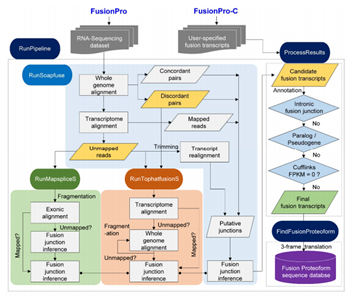
其中最重要的融合基因获取采用了SOAPfuse,TopHat-Fusion和MapSplice2。为了进一步降低获取的融合基因的假阳性,避免内含子部分的剪接,同源/假基因和未表达基因的干扰,所有融合基因结果将通过以下的条件进行过滤:1. 融合基因剪接位置不能来自内含子部分;2. 融合基因不能有间接同源或者假基因的关系;3. 融合基因转录本必须要有显著表达。
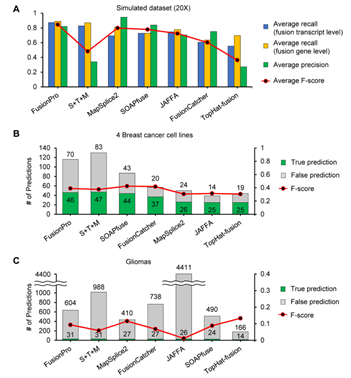
为了评估FusionPro的表现,结合多款基因融合软件的横向测评,结果见下图,在模拟数据集,乳腺癌细胞系和神经胶质瘤样品中的效果无论是最终获取的融合基因数目,还是准确率均在前列,证明了FusionPro在基因融合鉴定上的效果。
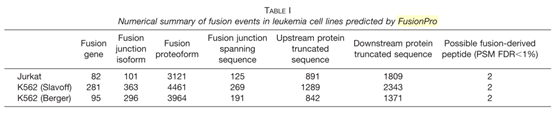
紧接着,作者将FusionPro应用在了白血病细胞系的研究中,在3个细胞系中,分别鉴定到了82,281和95个融合基因现象,它们具有很大的共享部分,最后利用Mascot对这些细胞系的质谱数据进行鉴定,共鉴定到了6条基因融合肽段(FDR<=0.01)。类似地,作者还在CPTAC的卵巢癌数据中进行类似的分析,得到了15条融合肽段(FDR<=0.01)。

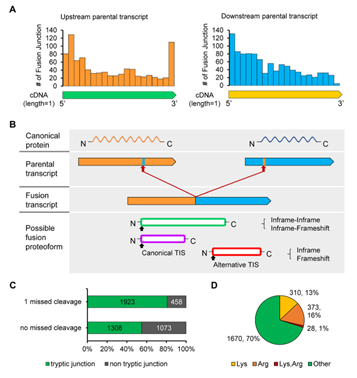
接下来,作者试着利用鉴定得到的基因融合现象来探索可能的基因融合剪接规律,由结果可见,融合基因的剪接位置相对于上游供体基因和下游供体基因来讲,都较为靠近5‘UTR,这也解释了为何融合基因一般都会保留下游供体基因的功能元件。但是对于上游供体基因来说剪接位置靠近3’端的也有很多,也许是由于上游供体基因在3‘UTR中含有一个未激活的翻译起始位点。作者还进一步地探索了漏切和胰酶肽段剪接的关系,由于胰酶肽段的特性(以KR结尾),所以不是所有融合形式都可以被胰酶肽段覆盖。另外,作者还观察到了在剪接位置高比例的Lys和Arg富集现象。
综上,FusionPro为蛋白组基因组学者提供了一套可靠的研究基因融合的工具。
作者:任哲
文章引用:



 浙公网安备 33010602011771号
浙公网安备 33010602011771号