Journal of Proteome Research | Prediction of an Upper Limit for the Fraction of Interprotein Cross-Links in Large-Scale In Vivo Cross-Linking Studies (分享人:张宇星)
题目:Prediction of an Upper Limit for the Fraction of Interprotein Cross-Links in Large-Scale In Vivo Cross-Linking Studies
期刊:Journal of Proteome Research
发表时间:July 3, 2019
DOI:10.1021/acs.jproteome.9b00189
作者及单位:
Andrew Keller——Department of Genome Sciences, University of Washington, Seattle, Washington 98195 United States
Juan D. Chavez——Department of Genome Sciences, University of Washington, Seattle, Washington 98195 United States
Kevin C. Felt——Department of Genome Sciences, University of Washington, Seattle, Washington 98195 United States
James E. Bruce——Department of Genome Sciences, University of Washington, Seattle, Washington 98195 United States
分享人:张宇星
概述:
化学交联质谱技术(Chemical cross-linking mass spectrometry)在研究蛋白构象和蛋白互作领域正飞速发展,随着越来越多可碎裂交联试剂的推出,在复杂样本中通过化学交联质谱大规模鉴定蛋白互作得到普及,蛋白样本化学交联后通过质谱鉴定可以得到两种结果:蛋白内交联(intraprotein)和蛋白间交联(interprotein),这其中蛋白间交联的鉴定对于蛋白互作的研究最为重要,然而究竟如何评判交联质谱鉴定结果的质量,目前还没有合适的标准。本文中作者通过计算所有的目前已有蛋白结构信息的各种交联结果,得到了理论上复杂样本中蛋白间交联所占比例的范围,为该领域的研究提供了可参考的标准。
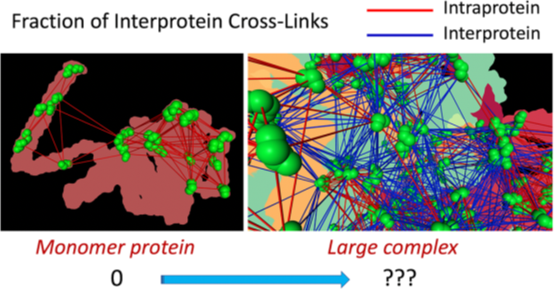
实验方法和结果:
1、 复杂样本理论蛋白交联的计算
作者从蛋白结构数据库 RCSB Protein Data Bank下载了共148,586个去除冗余和非高分辨率的蛋白及蛋白复合物的有效结构,由于目前的化学交联试剂主要是与赖氨酸侧链反应的,所以作者计算了所有蛋白复合体中理论上任意两个赖氨酸Cα之间的距离,包括直接计算欧式距离(Euclidean distance)和计算可接触表面距离(Solvent accessible surface distance,SASD),根据常用的可碎裂交联试剂允许的最长交联距离,统计其中符合距离要求的属于蛋白间交联的肽段数目。
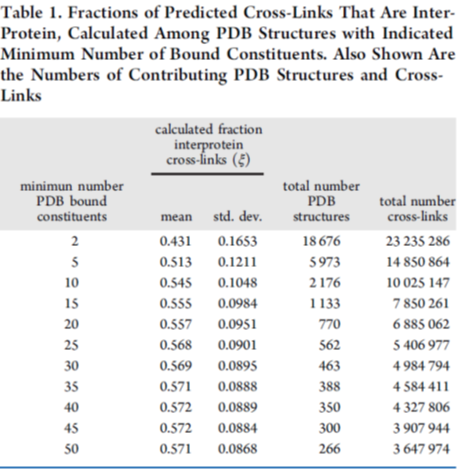
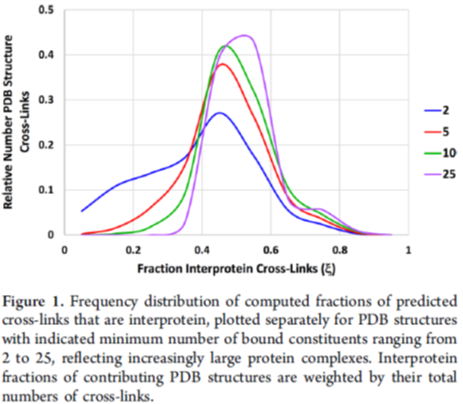
表1及图1展示了直接计算欧式距离的结果,蛋白间交联肽段在总交联肽段中的频率用ξ 表示,并且把PDB中的蛋白复合体按照复合体中组成蛋白的最少数目分别计算蛋白间交联肽段的频率,只由2个蛋白组成的复合体可以模拟真实实验中的简单样本,而又50个蛋白组成的复合体可用来表示实验中的复杂样本,结果显示,当复合体是由超过25个蛋白组成时,蛋白间交联肽段的频率 ξ 平均值趋于稳定,达到0.568;随后通过蒙特卡洛模拟方法验证了这个结果,得到的平均值为0.563,与0.568基本一致,证明结果的可靠。
由于直接计算欧式距离没有考虑到蛋白的位阻作用,所以作者又结算了SASD,但由于计算SASD十分耗时,作者只随机选择了1000个PDB数据进行计算,得到的结果与直接计算欧式距离的结果进行比较,结果如图2。
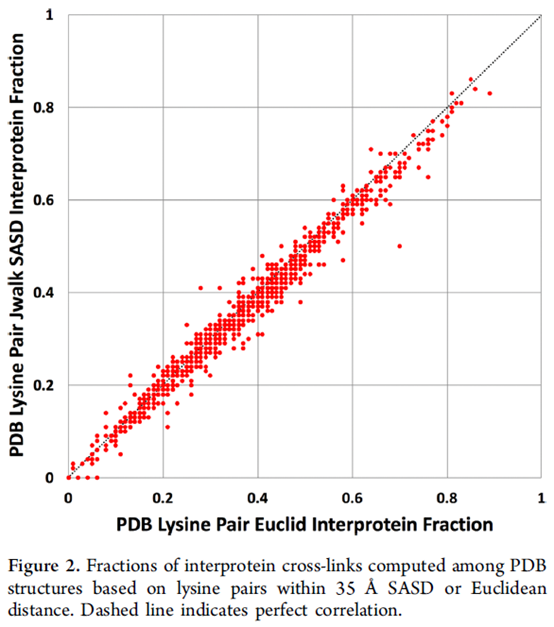
令人惊奇的是,SASD计算得到的结果和欧式距离计算的结果有高度的一致性,说明直接计算欧式距离的结果是可靠的。
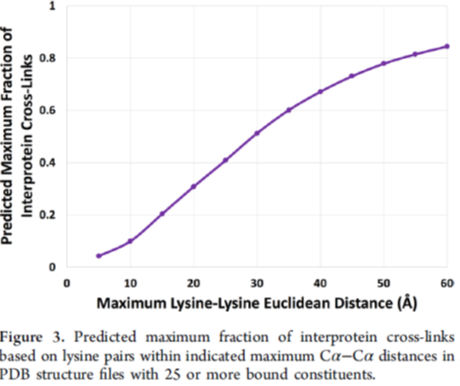
由于不同的交联试剂长度不一样,而交联到的肽段数目与交联试剂的长短有一定的关联性,所以作者也计算了不同的任意两个赖氨酸Cα之间的距离与蛋白间交联肽段频率的关系,结果如图3所示,确实当交联试剂长度越长时,蛋白间交联肽段的频率会提高,但两个赖氨酸Cα之间的距离大于50Å后增长幅度越来越小。
考虑到真实的蛋白质在生物体内发生作用时可能有各种构型,直接计算欧式距离得到的结果是否能代表真实实验中得到的结果,作者用蛋白交联数据库XLinkDB中使用交联试剂PIR和DSSO得到的真实蛋白交联结果,结合PDB中的蛋白三维模型和空间距离,计算了XLinkDB结果中交联肽段频率在PDB中的欧式距离,结果如图4。
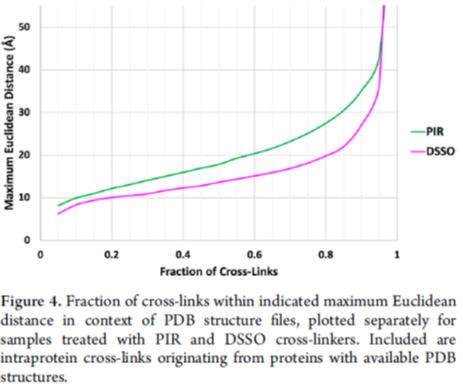
PIR可连接的两个赖氨酸Cα长度通常在35 Å内,而DSSO通常在27 Å内,由图4可知,在实验数据中,90%的交联肽段两个赖氨酸Cα之间的距离在所用交联试剂的最长连接范围内,剩下10%超过交联试剂连接范围的交联肽段可能是由于PDB中部分蛋白结构分辨率不足造成的误差,或者是一些在生物过程中出现的特殊形式构象导致的,还有可能是搜库中的假阳性结果,但总的来说,实验数据可以说明之前的理论计算结果是可靠的。
2、 有关交联肽段FDR的讨论
当假设所有的假阳性结果都是蛋白间交联肽段时,蛋白间交联肽段的FDR计算公式如下:
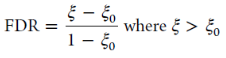
其中 ξ 为观测到的蛋白间交联肽段频率,ξ0 为实际上真实蛋白间交联肽段的频率,作者将XLinkDB数据库中的多组数据用上述公式校正后与常用的搜库用的FDR (Decoy-Estimated FDR)进行了比较,结果如图5,
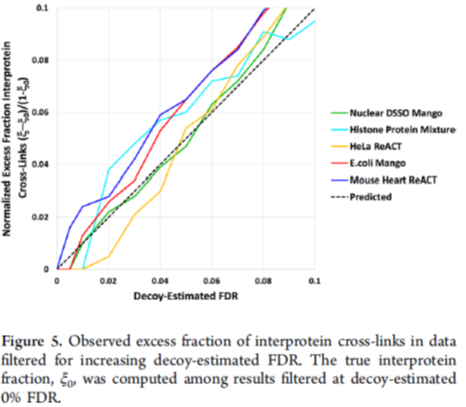
结果显示绝大部分交联肽段的实际FDR会比搜库时通过反库计算的FDR要高,如果考虑到假阳性交联肽段中还有可能包含蛋白内交联,那实际上蛋白间交联肽段的FDR会更高,这也是为什么在交联质谱实验的搜库中,分数较低的交联肽段中绝大部分时蛋白间交联肽段。
进一步将,由于作者通过PDB计算得到的交联肽段的最大频率与真实蛋白间交联肽段频率之间存在如下关系,ξmax ≥ ξ0 ,将此不等式带入上述公式可得:
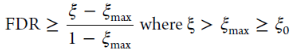
而PIR和DSSO作为交联试剂时的ξmax 已经分别算出为0.6和0.45,所以可以知道在使用这两种交联试剂时的FDR:
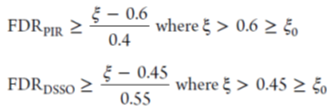
这样就有利于帮助科研人员更准确的计算交联数据的FDR,从而使结果更加可靠。
同时作者还提出了一个用来表述样品中蛋白复合体所占比例的公式:
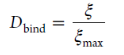
3、 in vivo与in vitro交联实验的结果差别
作者将Hela细胞作为材料,分别进行了直接在细胞上进行交联的in vivo交联实验和将细胞通过研磨、破碎后再交联的in vitro交联实验,随后都进行质谱分析,得到的结果如下:

经过计算,在in vivo实验中蛋白复合体所占比例要明显高于in vitro实验(0.43 vs 0.22),说明当细胞进行研磨、破碎等操作后,确实会破坏部分蛋白复合体的组成。
总结:
在化学交联质谱技术的发展中一直缺乏对鉴定结果质量的有效判断标准,尤其对于样本是否过度交联、鉴定结果假阳性率等问题时缺乏客观的标准,一些实验室通过将两种来源不同物种的蛋白通过和不同浓度交联试剂反应,来得到可使用的交联试剂浓度的最高限度以及搜库结果中假阳性交联肽段的得分,从而作为标准指导各自的实验。然而由于这样的实验结果跟预实验的样品关联度很高,并不一定适用于其他实验,所以不能广泛的推广。
本篇文章中作者通过对PDB数据库中蛋白复合体的计算,提供了理论上交联肽段的频率范围,为更多的实验人员审视自己的实验数据提供了参考,只是文章中只提供了PIR和DSSO两种交联试剂的相关结果,如果未来能提供更多交联试剂,相信会对交联质谱领域的发展提供更多帮助。


 浙公网安备 33010602011771号
浙公网安备 33010602011771号