解读人:李思奇,Development of a sensitive, scalable method for spatial, cell-type-resolved proteomics of the human brain. (一种用于研究人类大脑基于空间或细胞类型的蛋白质组学的灵敏方法)
发表时间:(2019年4月)
一、 概述:
本文报道了一种可研究人类大脑组织中特定神经细胞的蛋白质组学的方法。作者通过激光捕获显微切割技术(LCM)从逝者大脑中分离出目的神经元细胞,接着尝试了一系列不同的样品收集方法,裂解液和酶解方法,最终从10 um厚,60,000 um2大小的小脑分子切片中鉴定到了稳定重复的1500蛋白。用此方法,作者从分出的贝兹细胞(Betz)和浦肯野细胞(Purkinje)分别得到了高度特异表达的2800-3600个蛋白。
二、 研究背景:
蛋白质组学研究通常需要毫克级别的组织作为起始材料。大块的组织内含有多种细胞类型,得到的分析结果是不同类型细胞的平均表现。已有文献报道用流式细胞荧光分选技术(FACS)配合微量样品蛋白提取技术分析单个到数百个细胞。FACS不适用于分离体积较大、组成复杂的脑神经细胞。本文用LCM技术分出贝兹细胞和浦肯野细胞,以这两种神经细胞为例搭建实验流程,研究人类大脑的细胞类型特异性蛋白质组学。本文方法或对研究蛋白稳态失衡相关的神经退行性疾病有所帮助,也能对人类大脑的多组学研究提供信息。
三、实验设计:

四、研究成果:
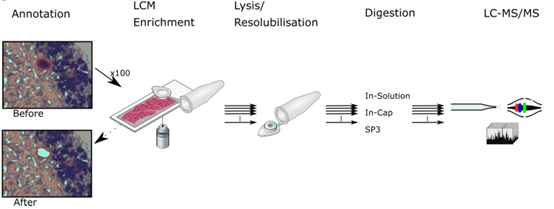
1. LCM-微量蛋白组学实验流程。用激光切割显微镜从10um厚的LCM切片上分离出浦肯野细胞,切下的样品落在离心管盖子里。在盖子里裂解样品后,用Single-Pot Solid-Phase-enhanced Sample Preparation (SP3)方法进行酶解。酶解后的肽段上质谱检测。
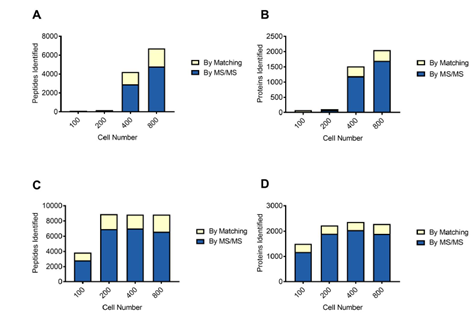
2. 比较回收样品的方法。从LCM切片上分出100,200,400和800个浦肯野细胞,图A\B中的样品通过离心方法从离心管盖子转移到离心管中后,进行裂解和酶解,图C\D中的样品直接在离心管盖子中进行裂解和酶解。在盖子中直接酶解的方法能大量提升样品的肽段和蛋白鉴定数,对少量细胞的效果更加明显。
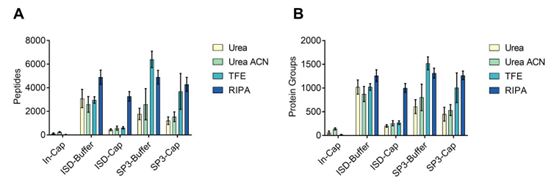
3. 比较回收样品和蛋白酶解的方法。用100个浦肯野细胞测试不同的样品收集、裂解液和酶解方法的组合。In-Cap表示收集时样品落在离心管盖上,ISD表示液态酶解。最佳方法组合是收集时让样品落在离心管盖里的溶液中,用50%TFE做裂解液,配合SP3酶解方法(buffer-TFE-SP3)。三次实验重复得到的平均值是肽段数6399,蛋白数1521。
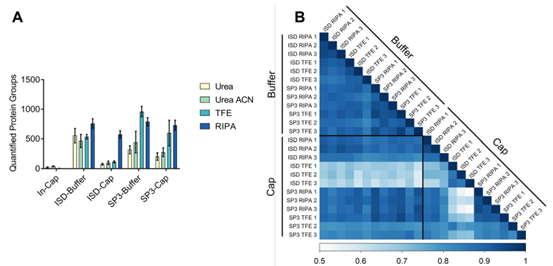
4. 定量相关性。图A,用buffer-TFE-SP3方法处理样品,Maxquant软件分析数据,从100个浦肯野细胞中得到可定量蛋白958个。图B,只用液态酶解或SP3酶解方法配合50%TFE或RIPA溶液得到结果,三次实验重复的组内Pearson相关性高。
文章亮点:
本文结合了LCM技术和微量蛋白提取技术,检测同一大脑组织中不同神经细胞的蛋白质组。本文描述的方法可以得到某一类神经细胞独特的蛋白质组学信息,并与其它种类的神经细胞进行比较。这一方法将为神经、神经疾病相关研究提供更多的信息。



 浙公网安备 33010602011771号
浙公网安备 33010602011771号