Mol Cell Proteomics. |王欣然| 基于微粒的蛋白聚合物捕获技术让能满足多种不同需求的蛋白质组学样品制备方法成为可能
大家好,本周分享的是发表在Molecular & Cellular Proteomics. 上的一篇关于蛋白质组学样本质谱分析前处理方法改进的文章,题目是Protein aggregation capture on microparticles enables multi- purpose proteomics sample preparation,通讯作者是丹麦哥本哈根大学的Jesper V. Olsen。
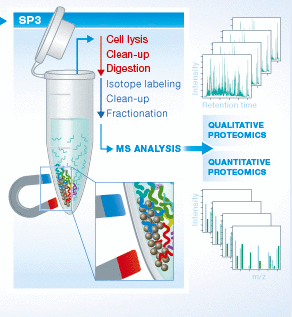
对于蛋白质组学研究人员来说,在进行质谱分析之前,一个良好的蛋白样本制备方法可以有效降低各类杂质对于液相和肽段离子化等在质谱测序过程中的干扰,从而获得更理想的检测结果。在样本制备过程中,为了能尽可能得到研究样本中所有的蛋白质,就需要使用强力的去污剂以完全裂解组织、细胞等结构——然而这些化学物质难以去除并且导致明显的质谱分析信号干扰。在本篇文章中,作者研究并完善了一种名为SP3 (Single‐Pot Solid‐Phase‐enhanced Sample Preparation)的技术。他们在理解技术原理的基础上,优化了各项参数达到从而使这项技术正式应用成为可能。
SP3技术是由德国欧洲分子生物学实验室C. S. Hughes等人开发并于2014年发表在Molecular System Biology杂志上,这一技术被认为能够代替FASP等制备手段并且具备高灵敏度。Hughes等人发现,通常用于某种肽段专一富集的磁珠,在有机溶剂的诱导之下可以无差别的固定溶液中所有蛋白和肽段,并且降低pH也可以促进这一进程。利用这一现象可能有效地在酶解之前就完全去除裂解液成分对后续步骤的影响。对于该现象背后的原理,Hughes等人认为可能是磁珠表面的羧基基团的亲水作用(HILIC,hydrophilic interaction chromatography)结果,有机溶剂的存在改变液体环境从而诱导蛋白和肽段与磁珠表面的相互结合。
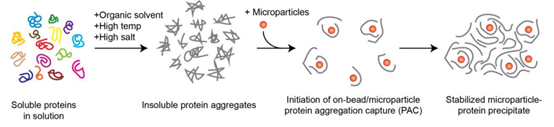
本文的研究者们首先对于这一现象进行了各种实验并试图理解该现象背后的原理。他们首先否定了HILIC原理下有机溶剂诱导蛋白固定化假说,因为该过程被发现不可逆——恢复水相环境不能使蛋白与微珠分离,但是LDS之类的增溶性缓冲液却可以。所以作者认为这是蛋白无法溶于水后形成的蛋白聚集物本身能使其与微珠结合,并且这一过程与微珠表面化学修饰类型或磁力性质没有关联,磁珠仅作为成核区或载体。为了进行验证,他们首先对已经加入磁珠的蛋白原液进行三种不同的蛋白聚合物产生方法,即有机溶剂(70%乙腈),高温(80度)和高盐条件(2.5M硫酸铵),并设置空白对照组;关于磁珠表面化学性质的影响,多种磁珠在蛋白固定上的效果通过SDS-PAGE进行了直观比较;磁珠磁力性质影响则通过纯铁粉进行验证。这些实验的结果都验证了假说,作者由此将其作为微珠蛋白固定化现象背后的一种机制,命名为protein aggregation capture (PAC)。
随后作者基于对SP3原理的理解,进行了一系列参数优化并展示了该技术在改进后的各项优势。首先通过对比试验和银染SDS-PAGE等方法,确定了合适的蛋白液浓度与微珠的相对比例,以及验证了该技术的极高的蛋白回收率,最终重新构建了一套完整的技术流程;接下来作者通过与其他制备方法LC-MS/MS结果对比证明了该技术可以用少于通常10倍用量的蛋白酶,达到比一般方法在标准蛋白酶用量下更低的漏切数(missed cleavage);该技术在蛋白翻译后修饰组学(如磷酸化蛋白质组)、器官和组织的蛋白质组学、蛋白互作分析和细胞分泌蛋白质组学上相对于其他制备方法有更优异的表现也通过LC-MS/MS和相应数据分析流程进行了比较验证。综上结果很好的展示了基于微珠蛋白固定化技术的样本制备流程相对现有方法,更适应多种起始样本量要求,成本更低并且简便省时。当然该方法也有局限性——不适合含有影响蛋白聚合的物质的样本。
文章解读:
文献分享二组-王欣然
文章引用:
doi:10.15252/msb.20145625
doi:10.1074/mcp.TIR118.001270
文章链接:
http://msb.embopress.org/content/10/10/757.long
https://www.mcponline.org/content/early/2019/03/04/mcp.TIR118.001270


