MIT Molecular Biology 笔记5 转录机制
视频 https://www.bilibili.com/video/av7973580?from=search&seid=16993146754254492690
教材 Molecular biology of the gene 7th edition J.D. Watson et. al
转录机制
一、RNA聚合酶和转录周期
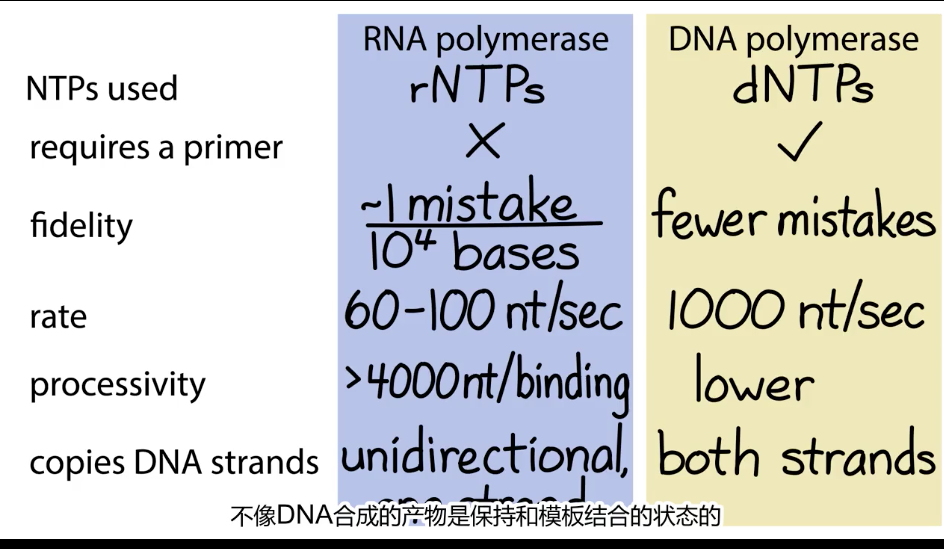
1、
- 细菌:一种转录酶
- 真核:三种转录酶(植物+2种)
- Pol I 大核糖体RNA前体
- Pol II 几乎转录所有蛋白质编码
- Pol III tRNA,小的核RNA基因,5S人RNA基因
- (Pol IV、V 植物中,参与转录小干扰RNA)
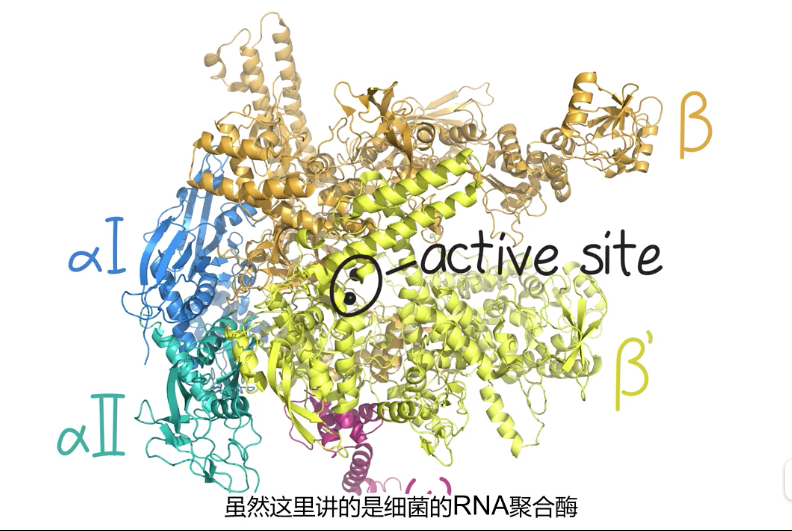
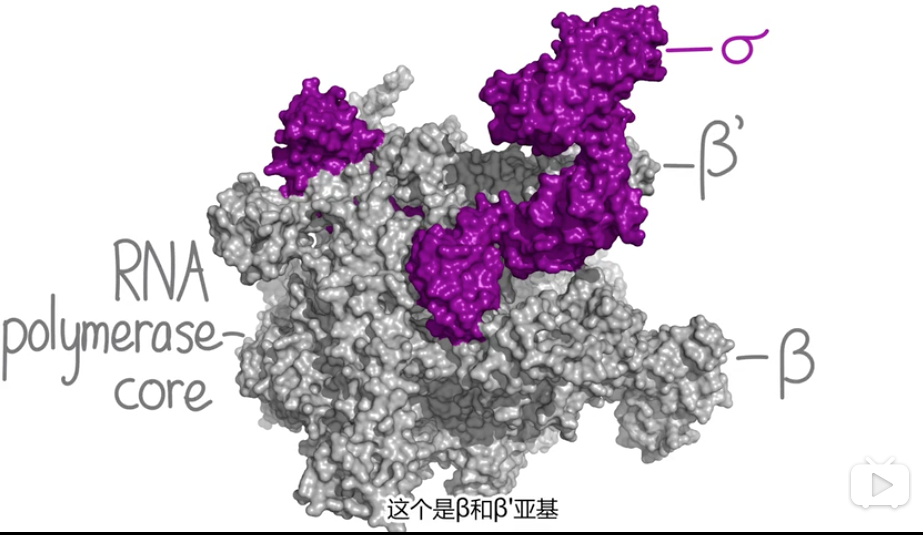
细菌聚合酶的核心酶可独立合成RNA
- 2 * α + β + β'大亚基 + ω
- 酶的形状像个蟹爪
- 钳子基部有活性中心裂隙
- 依靠双金属离子催化合成反应
- 位点结合一个Mg2+
- 另外一个离子随人NTP进入随PPi释放
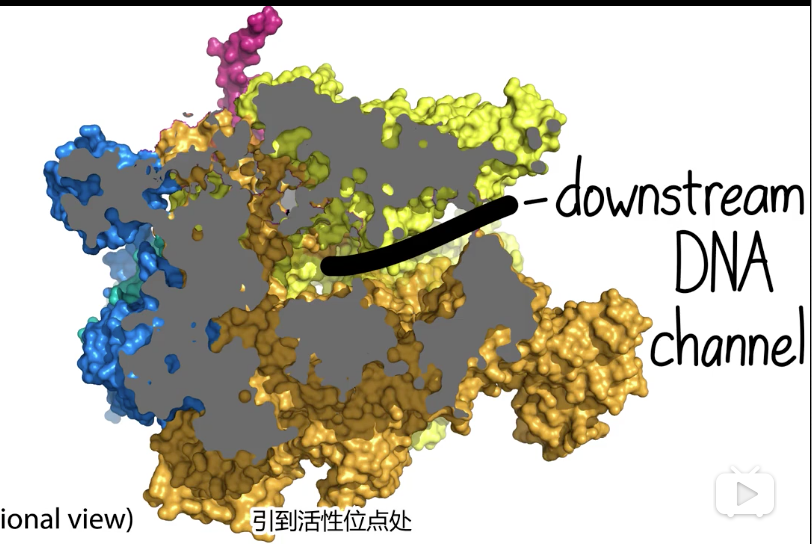
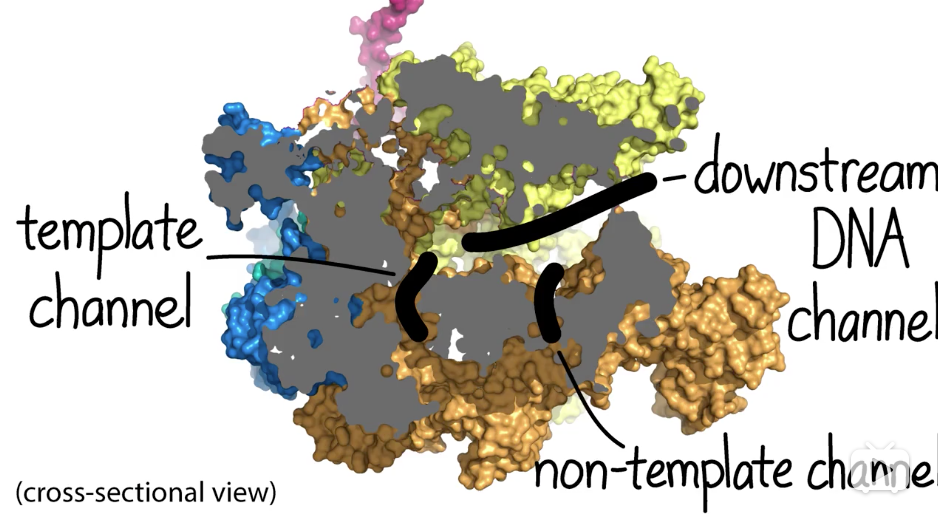
- 酶上有多种通道
2、由RNA聚合酶进行的转录是由一系列步骤完成的
- initiation
- promoter 最初结合RNA聚合酶的序列
- elongation
- 合成了10个碱基以后,便进入延伸阶段
- termination
3、转录的起始包含三个定义明确的步骤
- 闭合复合体的形成
- 双链状态
- 聚合酶结合在一个面上
- 转变为开放复合体
- 双链在起始位点13bp距离内形成转录泡
- 此时复合体叫:转录起始复合体(initial transcribing complex)
- 最初10bp合成效率相当低,会不断合成小片段释放
- 合成超过10bp,聚合酶逃离启动子
- 启动子逃离,进入转录起始阶段
原核的转录
二、细菌的转录周期
1、细菌的启动子在强度和序列上是多样的
- 原则上细菌RNA聚合酶核心酶可以在DNA分子的任意一点开始转录
- 但是聚合酶不能打开双链
- 但是在细胞内 ,有σ起始因子的参与,聚合酶只在启动子处转录
- σ + 核心酶 = 全酶(holoenzyme)
细菌启动子元件有不同的组合情况
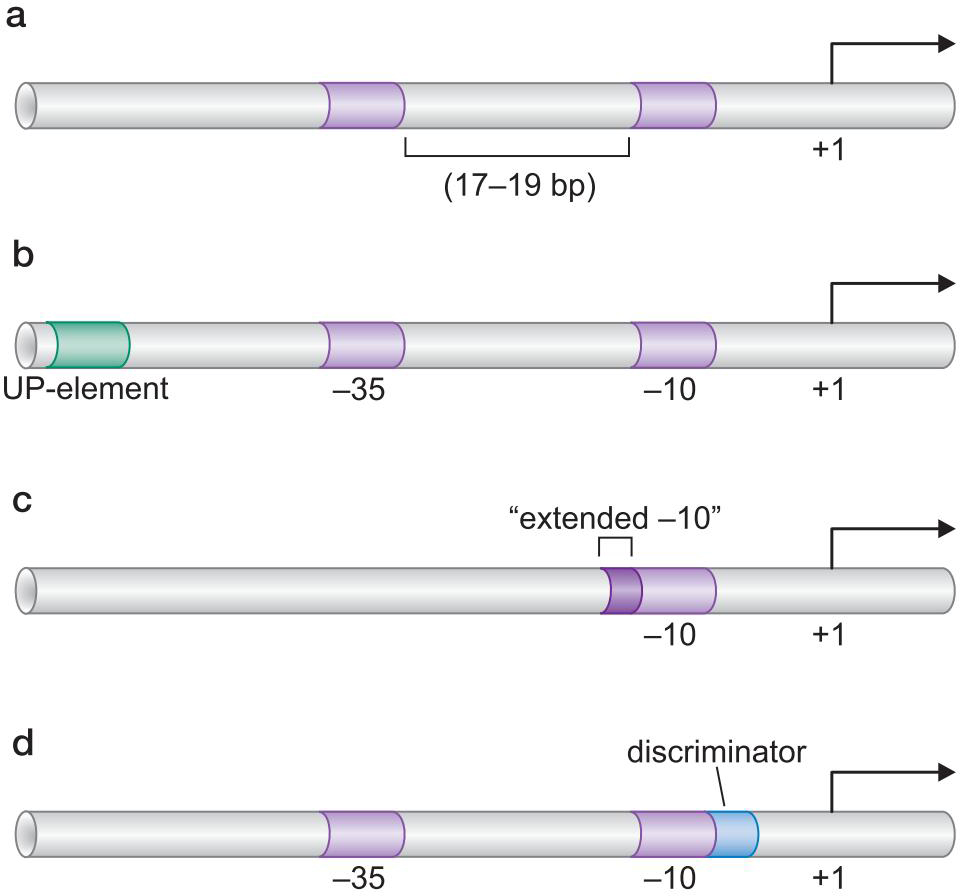
- 大肠杆菌最常见的σ因子是σ70 具有如下特征
- 两段6核苷酸长的保守序列
- -10, -35
- 被长度17-19bp的非特异性序列隔开
- 两段6核苷酸长的保守序列
- 启动的蛋白所识别的共有序列越相近,启动子越强
- 单位时间内转录产物越多
- 启动子的强度 与以下因素相关
- 启动子最初与聚合酶的结合程度
- 异构化作用的支持效率
- 此后聚合酶逃离的难易程度
- 这解释了表达水平的差异
- 较强的启动子中(如指导rRNA)有UP-element
- 提供额外的特异性
- 缺失-35区的σ70拥有上游延长-10区,以补偿
- 大肠杆菌gal基因
- -10下游有鉴别子(discriminator)(都有??)
- 与聚合酶作用影响稳定性
2、σ因子介导聚合酶与启动子的结合(形成闭合复合体)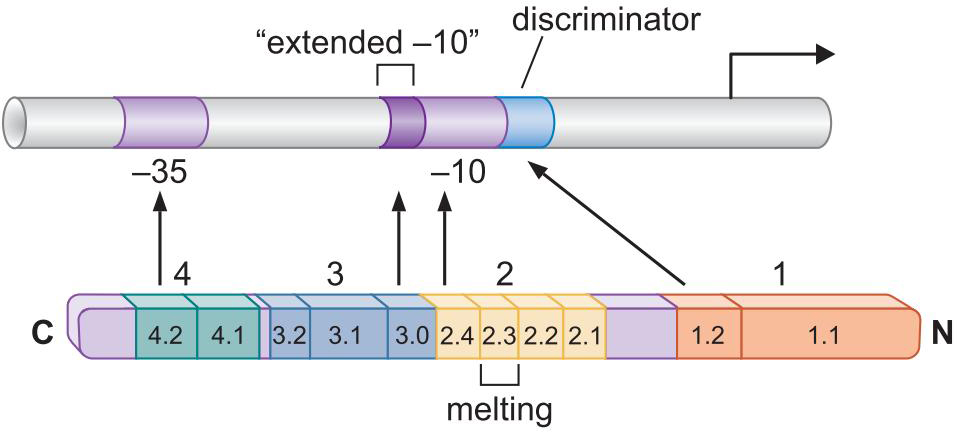
- σ70 有4个结构域
- -35 被区域4的两个螺旋形成 helix-turn-helix识别
- 一个插入-35 大沟,与碱基作用
- 另一个从大沟顶部横穿过,与骨架接触
- 与-35的结合提供能量确保聚合酶-启动子结合
- 这一种motif结构见于很多DNA结合蛋白(转录激活、抑制因子)
- -10 被区域2α螺旋识别
- 螺旋包含几个芳香氨基酸
- 可以与非模板链相互作用维持解旋DNA的稳定(类似SSB)
-
- 区域2结合一个单链状态的-10element
- 非模板链链上的两个碱基插入σ的口袋中
- -10 的延长序列被结构域3识别
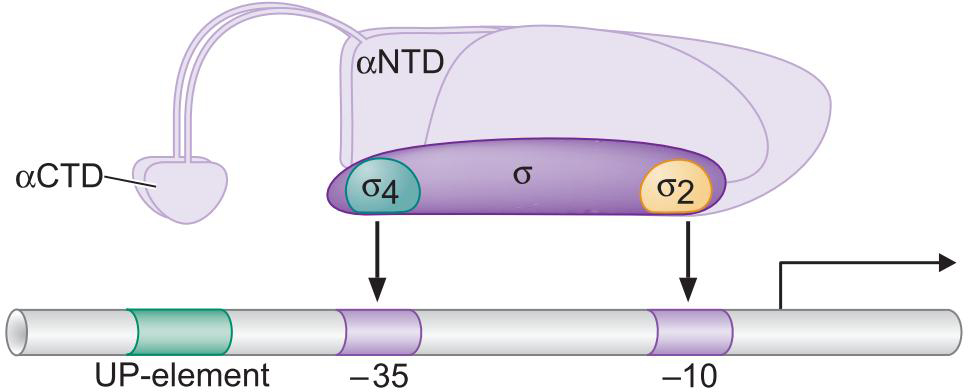
- 鉴别子由结构域1、2识别
- UP-element 不被 σ 识别,被α亚基羧基端αCTD识别
- 区域3.2被称为3/4连接区
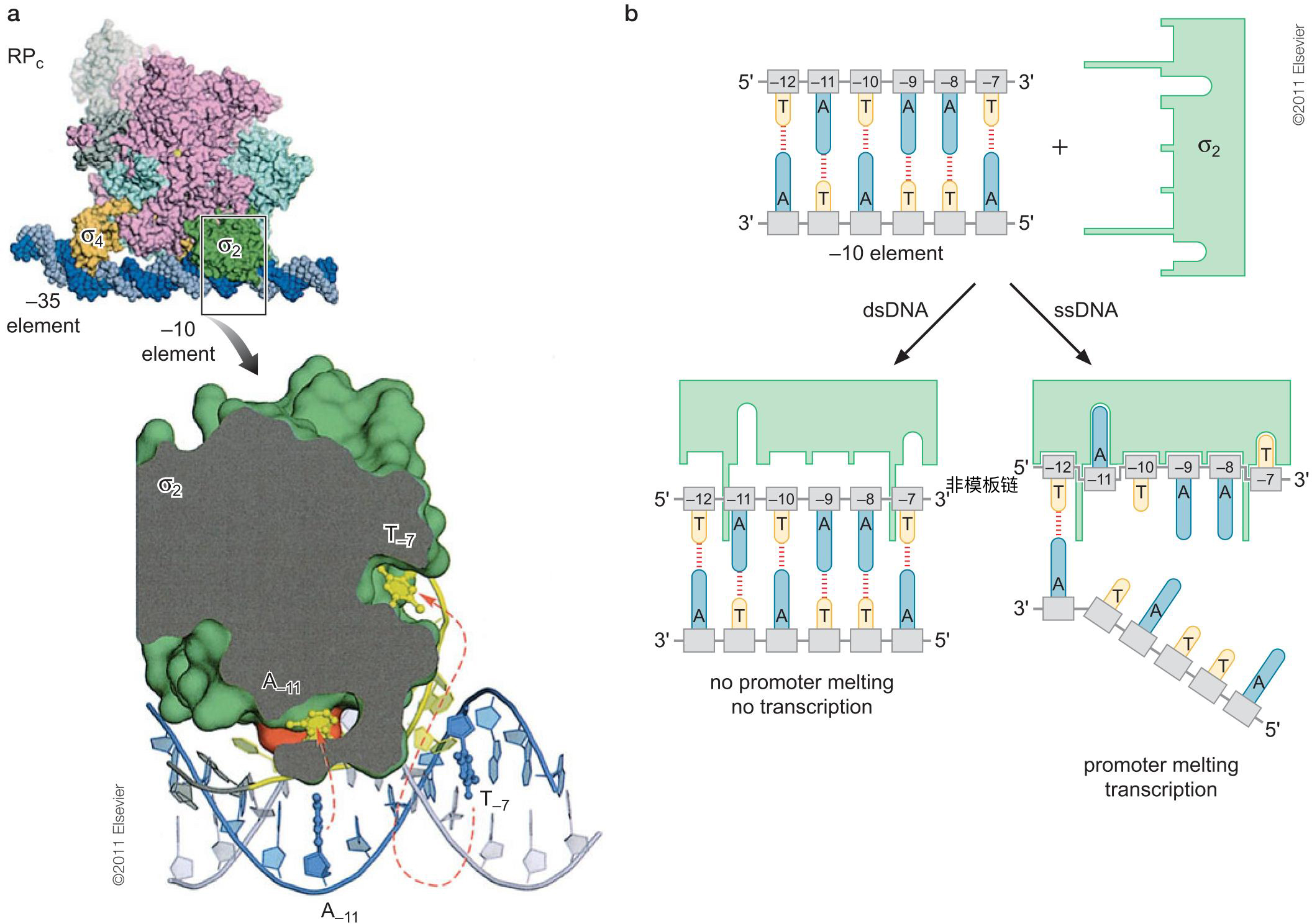
3、向开放式复合体的转变涉及RNA聚合酶和启动子DNA的结构变化
异构化作用(isomerization)
- 异构化作用不需ATP ,依靠优势构相驱动
- 异构化不可逆,开放式复合体形成后,转录随后起始
- 相反,闭合式复合体的形成是可逆的
- 在活性中心内,DNA从+3位置开始分离
- 聚合酶后的上游DNA在-11位置重新恢复双链 σ因子 N 端(1.1)起到分子模拟物(molecular mimic)的作用(+电)
- 结合DNA前,是1.1结合在活性中心
4、转录由RNA聚合酶起始,不需要引物
- 聚合酶需与一条或多条DNA模板链,起始核苷酸以及第二个核苷酸建立特异性作用
- 严格控制方向
- 大多数转录物以同一核苷酸开始
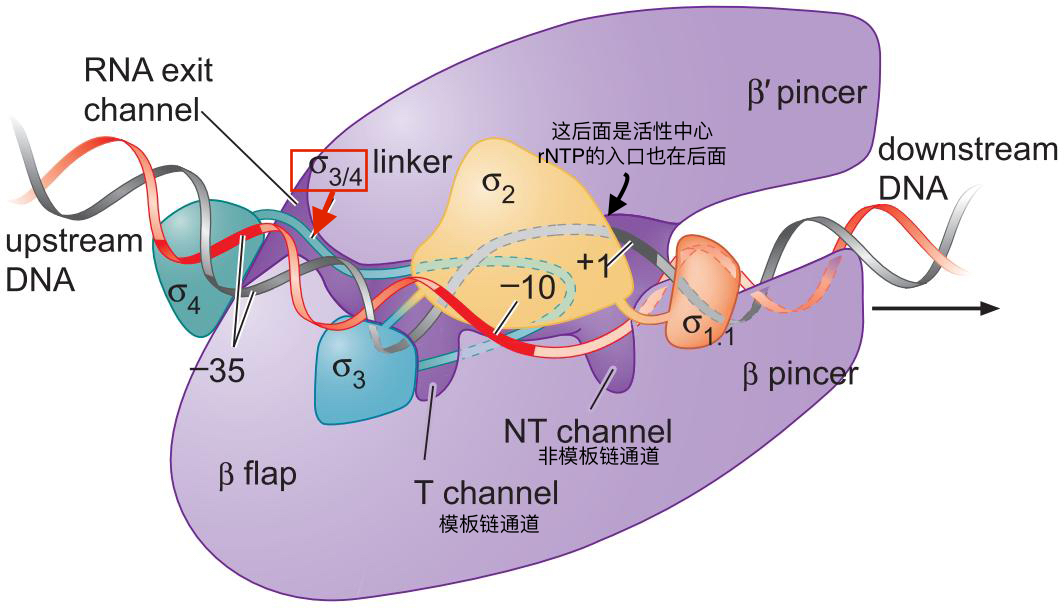
- σ的3/4linker 与模板链互作,,确保正确位置和构象启动转录
5、转录的起始阶段RNA聚合酶保持位置不变,并将下游DNA拉向自己
- 三种模型
- 瞬时漂移
- 蠕虫移动
- 蜷缩
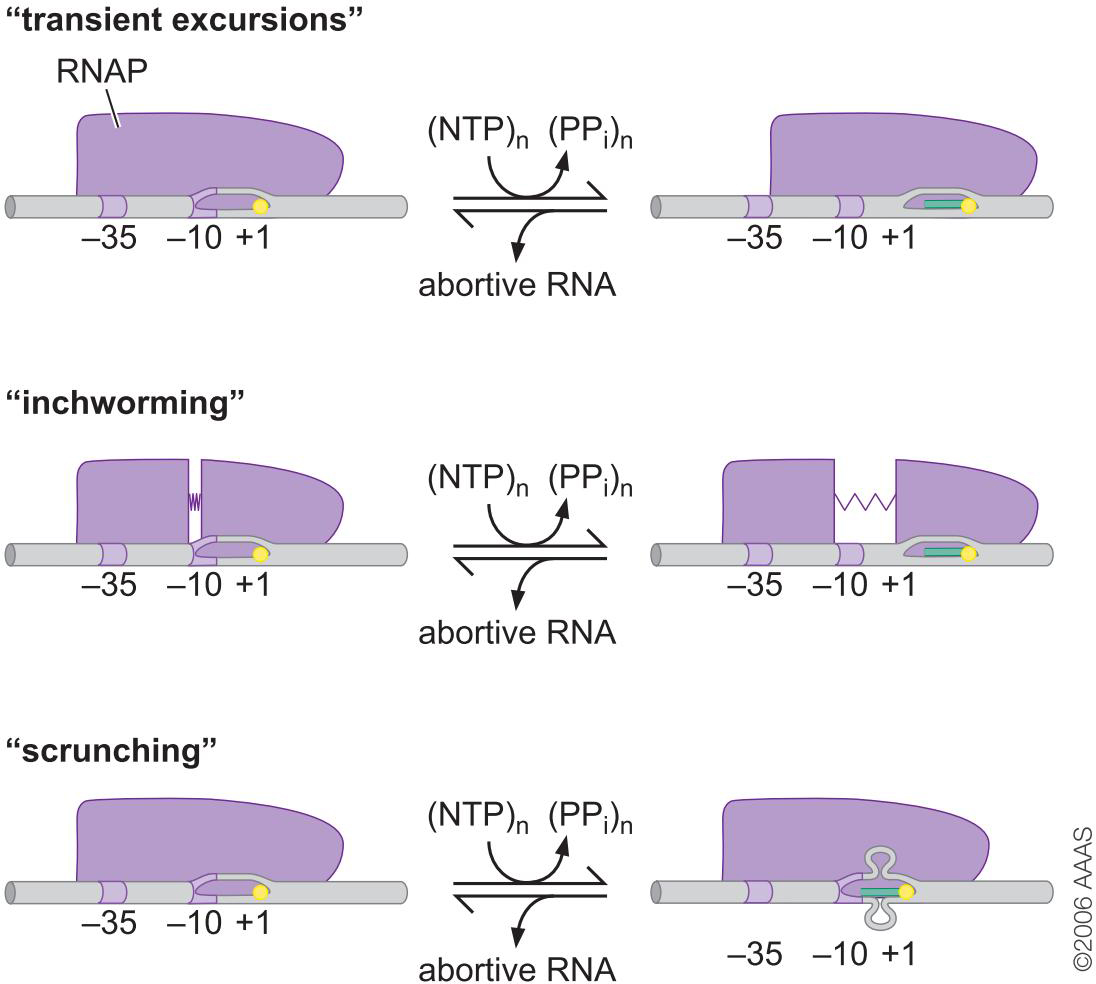
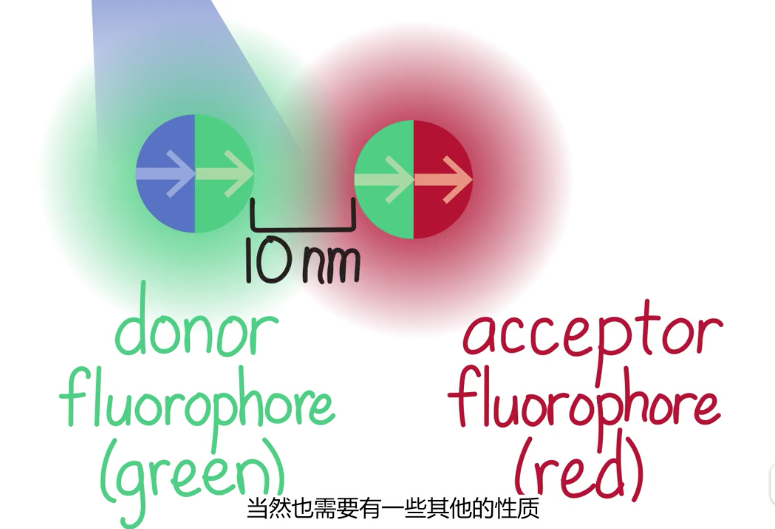
- 单分子实验(FRET)支持蜷缩模型
- 聚合酶下游的DNA被酶拉进来,在内部形成泡结构
6、启动子的逃离涉及聚合酶-启动子相互作用 以及
聚合酶核心-σ相互作用的破坏
- 一旦合成长度达到10bp,转录物变要穿入酶的RNA出口通道
- σ3/4作为分子模拟物占据RNA出口通道
- 合成达到10bp,RNA链便将σ3/4逐出
- σ也常常会从延长酶上丢失
- 逃离后,转录泡从22-24bp缩小为12-14bp
- 这会释放能量,让聚合酶离开启动子并驱逐σ因子
有些生物如大肠杆菌噬菌体T7是单亚基聚合酶,没有σ因子,但也有类似的结构变化以让合成超过10bp的RNA穿出RNA通道
7、延伸聚合酶是一个边合成边校对的机器
- 增长中的RNA有8-9个碱基与DNA保持互补
- 聚合酶每前进相当于单核苷酸长度的一步,3'端添加一个核苷酸
- 转录泡的大小不变
转录酶的校对机制(proofreading)
- 焦磷酸编辑 pyrophosphorolytic editing
- 是rNTP合成的逆反应
- 错配导致合成速度减慢(空间位点不正确)
- 聚合酶利用空间结构特性短暂限制PPi的离开
- PPi + RNAn = RNAn-1 + rNTP
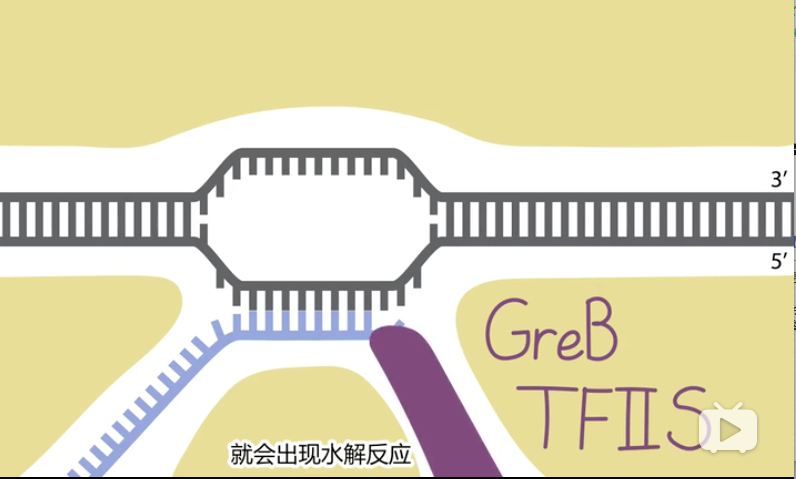
- 水解编辑 hydrolytic editing
- 聚合酶倒退入rNTP进入通道(利用环境中的热,0°C不发生,37°C发生)
- Gre因子(E.coli)、TFIIS(真核)进入
- 水解
8、聚合酶可能会因为模板损伤停滞不前,需要被挪走
TRCF挪走停滞的聚合酶,并且招募修复酶
- TRCF结合上游双链(依靠ATP),并且寻找到停滞的RNA聚合酶
- 推动聚合酶向前合成,或者令三重复合体解离
- 招募转录藕联修复酶(UvrA、B、C)
9、转录由RNA序列内部信号停止
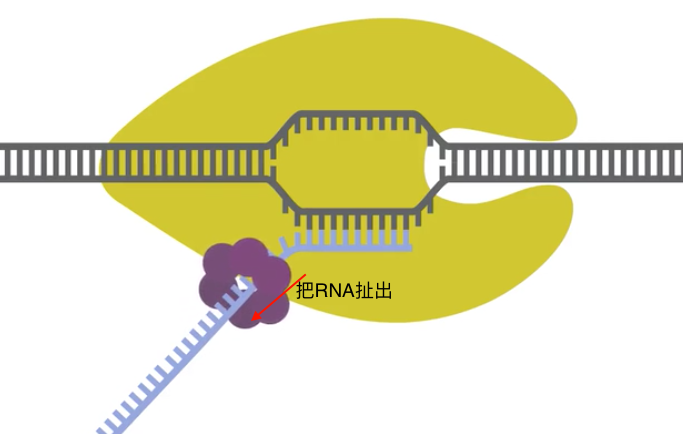
- Rho依赖型
- Rho(ATP依赖)从rut位点结合单链
- 寻找聚合酶
- 将RNA扯出,或像TRCF一样作用
Rho的结合有特异性:
- Rut位点理想:40bp,不折叠为二级结构,富含C
- 不结合正在翻译的RNA
- 故在细菌中,是等转录超过一个基因(编码序列)后才终止的
最近研究显示Rho因子是通过转录循环早期与RNA聚合酶结合
随着转录易位
易位导致紧绷
到一定程度,将酶终止
- Rho非依赖型
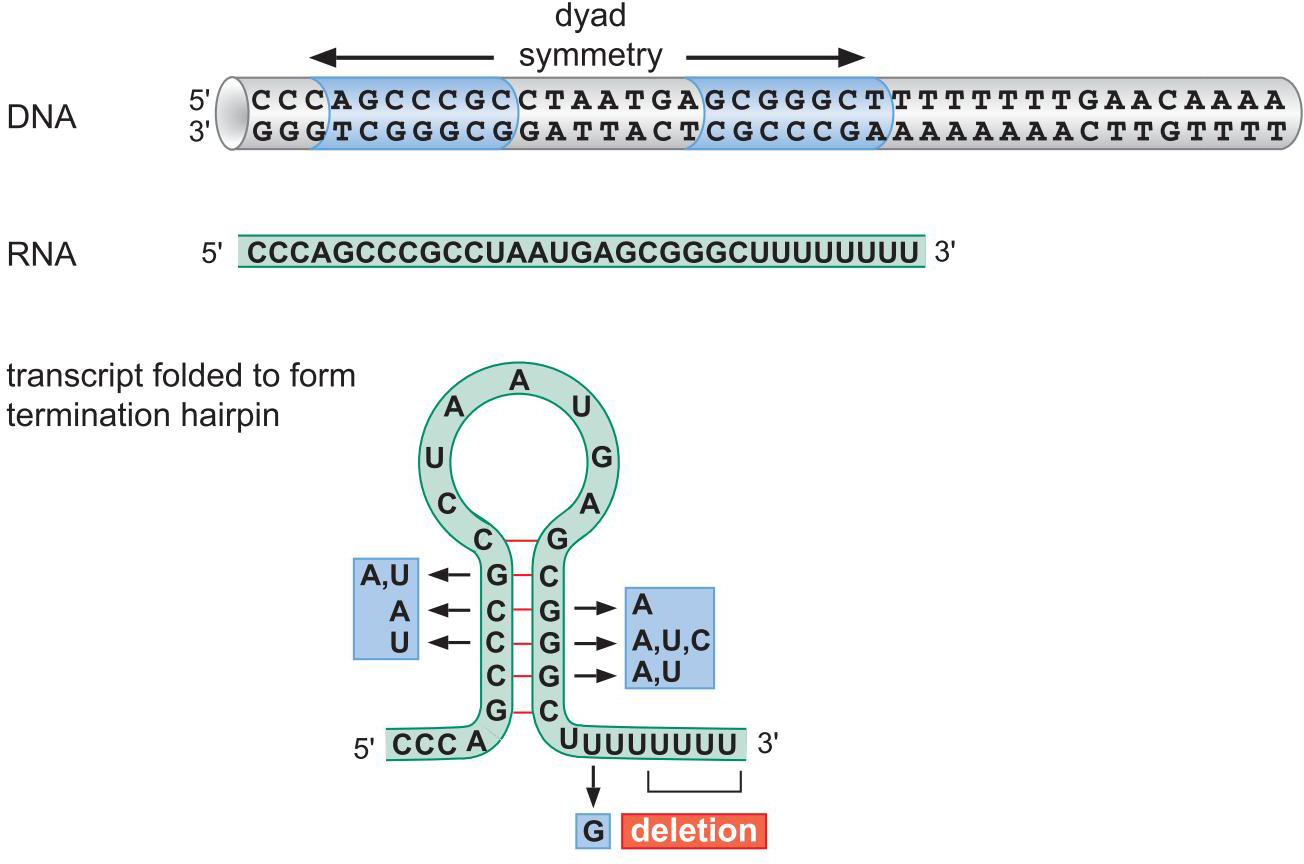
- 非依赖型终止子也称固有终止子(intrinsic terminator)
- 组成
- 反向重复序列
- 8个U
- 形成发夹,引发相关信号,转录物脱离
真核的转录
三、真核生物的转录
- 真核生物的转录除了需要聚合酶外,还需要通用转录因子General transcription factor,GTF
- 在体内,由于有核小体,转录还需要
- DNA结合调节蛋白
- 中介蛋白复合体
- 染色质修饰酶
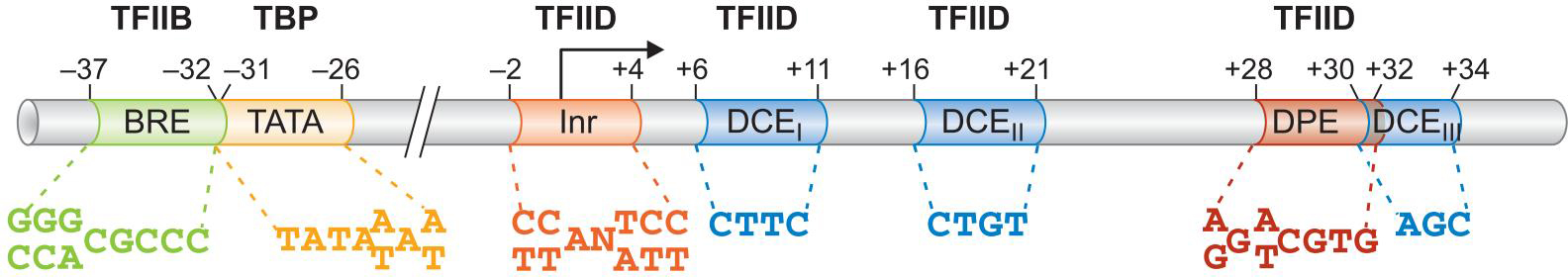
1、RNA聚合酶II的核心启动子由4个不同序列的元件构成
- TFIIB识别元件
- TATA元件
- 起始子(Inr)
- 下游启动子元件(包括DPE、DCE、MTE)
核心启动子之外,存在的在体内进行有效转录所需的其他序列元件。共同组成调节序列
- 启动子最近元件(promoter proximal element)
- 上游激活物序列(upstream activator sequence,UAS)
- 增强子
- 沉默着子
- 边界元件
- 绝缘子
2、RNA聚合酶II在启动子上和通用转录因子形成前起始复合物
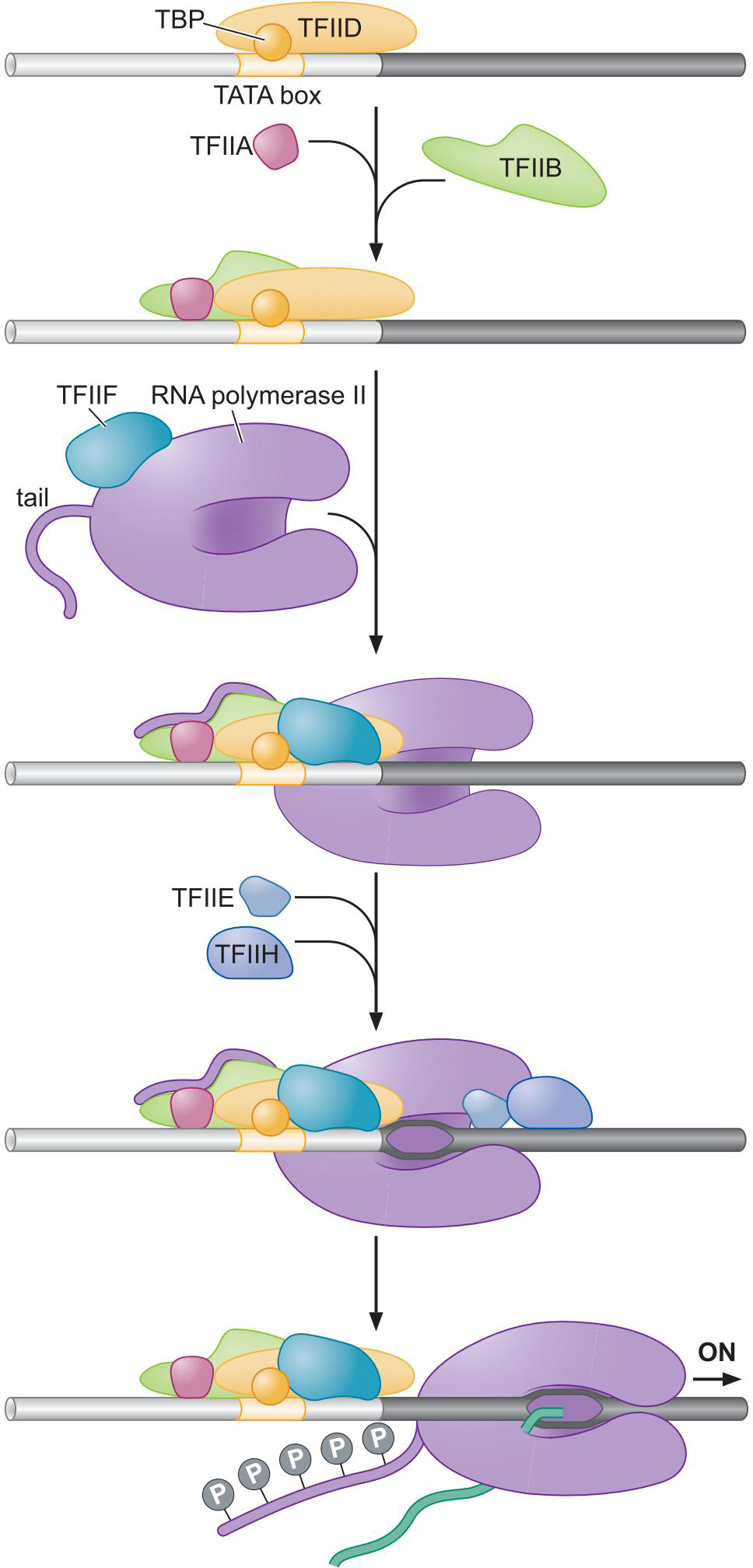
- TFIID由TBP(TATA Binding Protein)与TAF(TBP Associated Factor)组成
- TFIID 的TBP 识别TATA box
- 真核的启动子解旋需要TFIID的ATP水解介导
- 还需要聚合酶CTD磷酸化
3、启动子的逃离需要聚合酶的尾巴磷酸化
- Pol II 的CTD由连续重复7肽序列
- Tyr-Ser-Pro-Thr-Ser-Pro-Ser
- 每个重复序列都有激酶磷酸化位点
- TFIIH的一个亚基由激酶功能
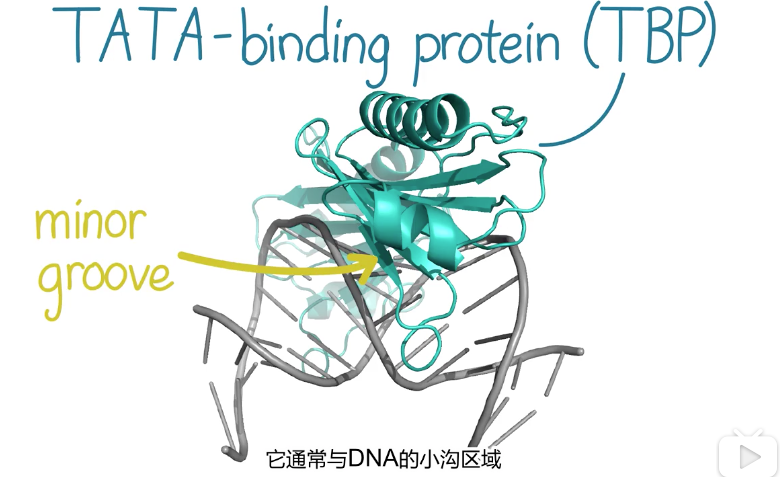
4、TBP用插入双螺旋小沟的β折叠来结合并扭曲DNA
- 结合的特异性来自插入到识别序列两端的碱基对,这由驱动DNA发生强烈弯曲的两对苯丙氨酸的侧链实现
5、其他通用转录因子在起始中也有特殊作用
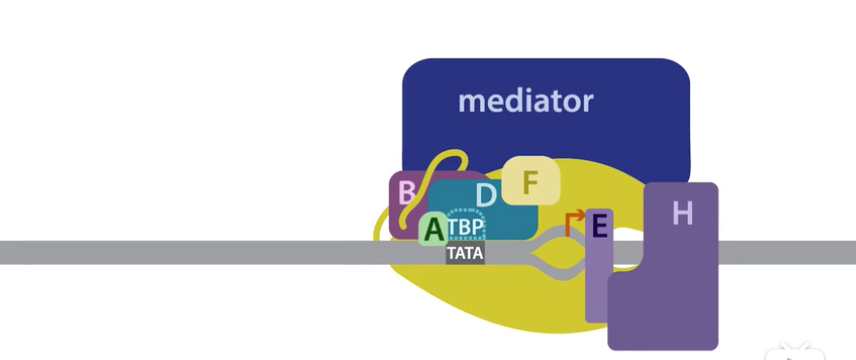
6、体内的转录需要其他蛋白的参与,包括中介蛋白复合体
聚合酶的CTD尾巴结合有mediator,它有助于聚合酶分子定位到启动子
- mediator的一个表面结合Pol II CTD尾巴
- mediator另外的表面结合其他起始因子
- 如果缺失中介蛋白,常会导致一些基因失去表达
- 中介蛋白把聚合酶定位到不同的启动子上
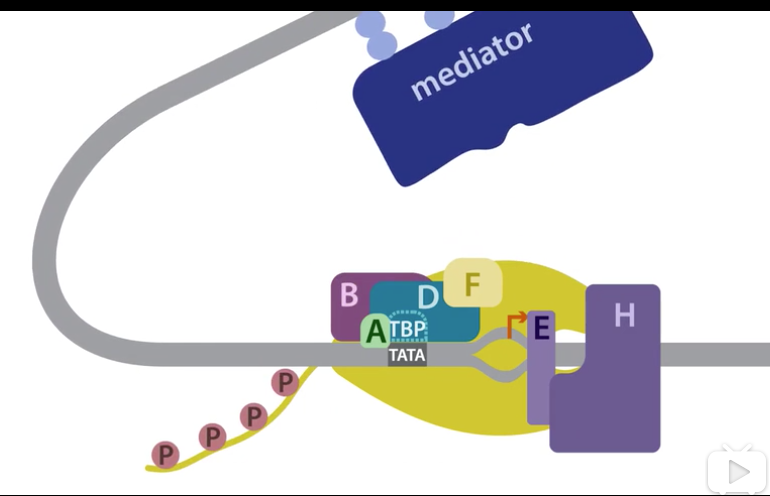
- 在TFIIH水解ATP的介导下,双链打开,转录起始
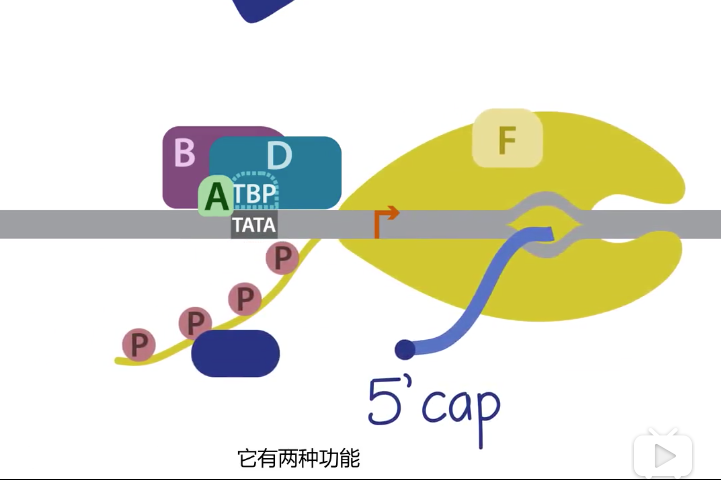
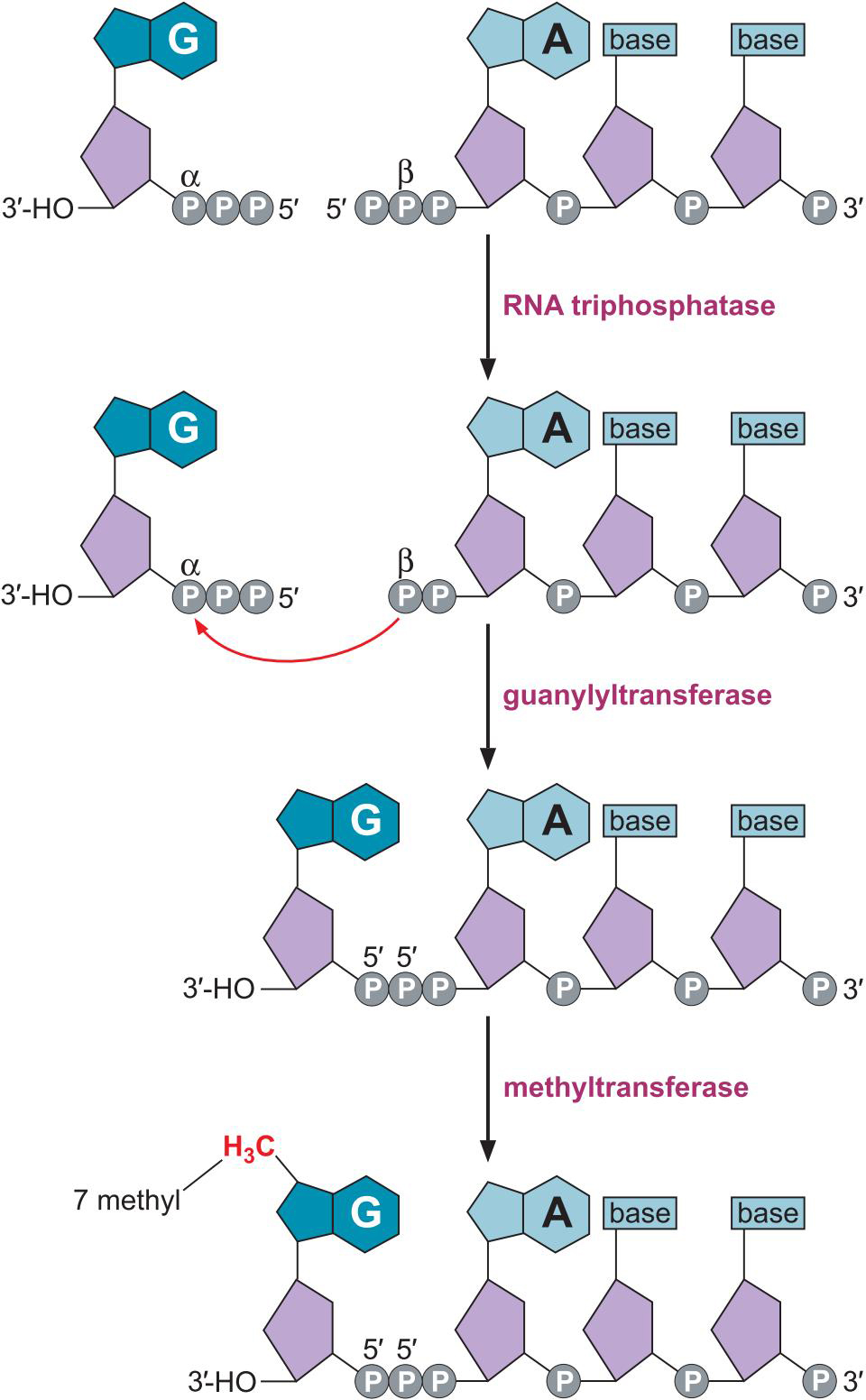
- 加帽酶5’端加帽
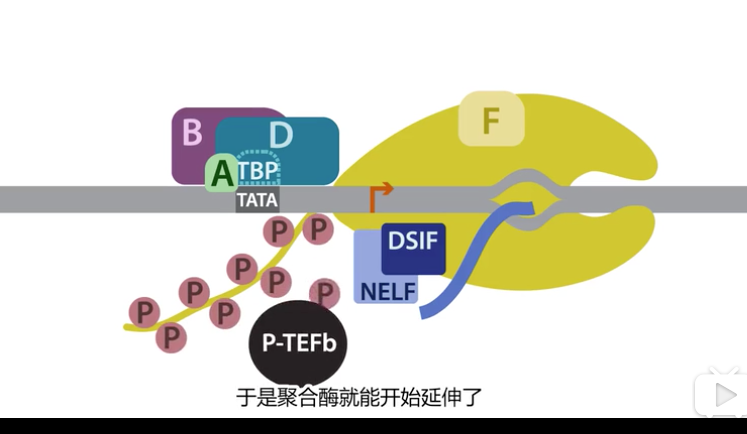
7、一组新的因子激发Pol II 延伸 和RNA校对(proofreading)
- NELF :负延伸因子
- DSIF :DRB敏感因子
- 它们抑制早期转录,使得转录在+25 ~ +60处停止
- P-TEFb会使得重新开始转录
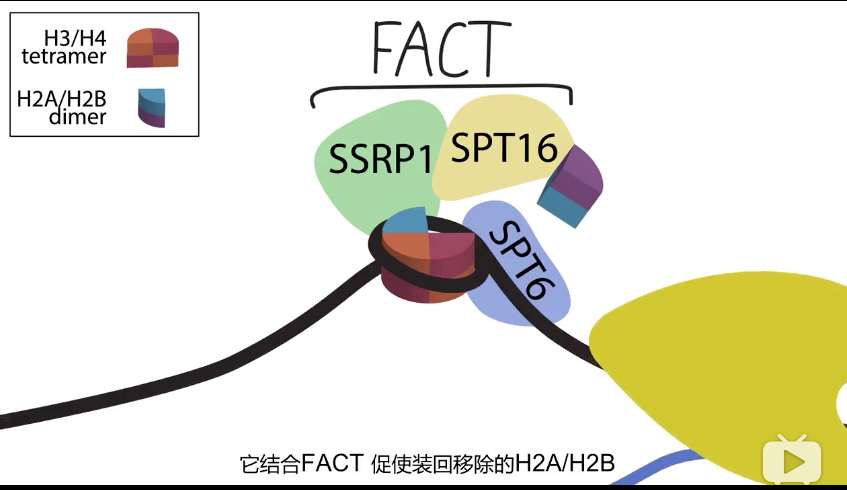
8、聚合酶延伸过程中对抗组蛋白的阻碍
- FACT 通过暂时挪开一个H2A·H2B二聚体方便转录
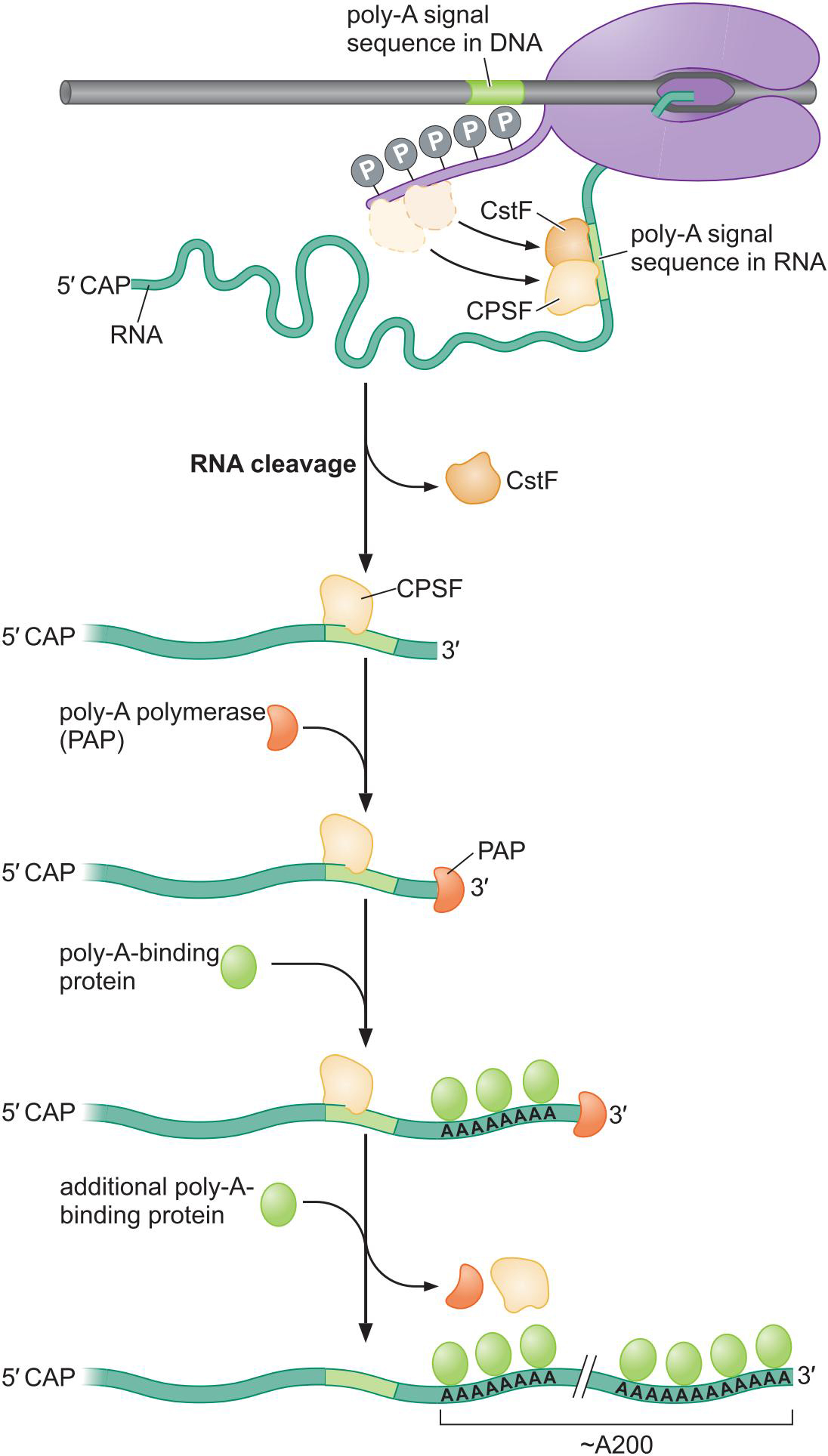
9、延伸聚合酶与各种RNA加工所需的一组新蛋白质相互作用
- 5'端加帽
- 转录poly-A信号
- 相关蛋白募集
- CSTF
- CPSF
- 切割
- 多聚腺苷酸化
- 终止
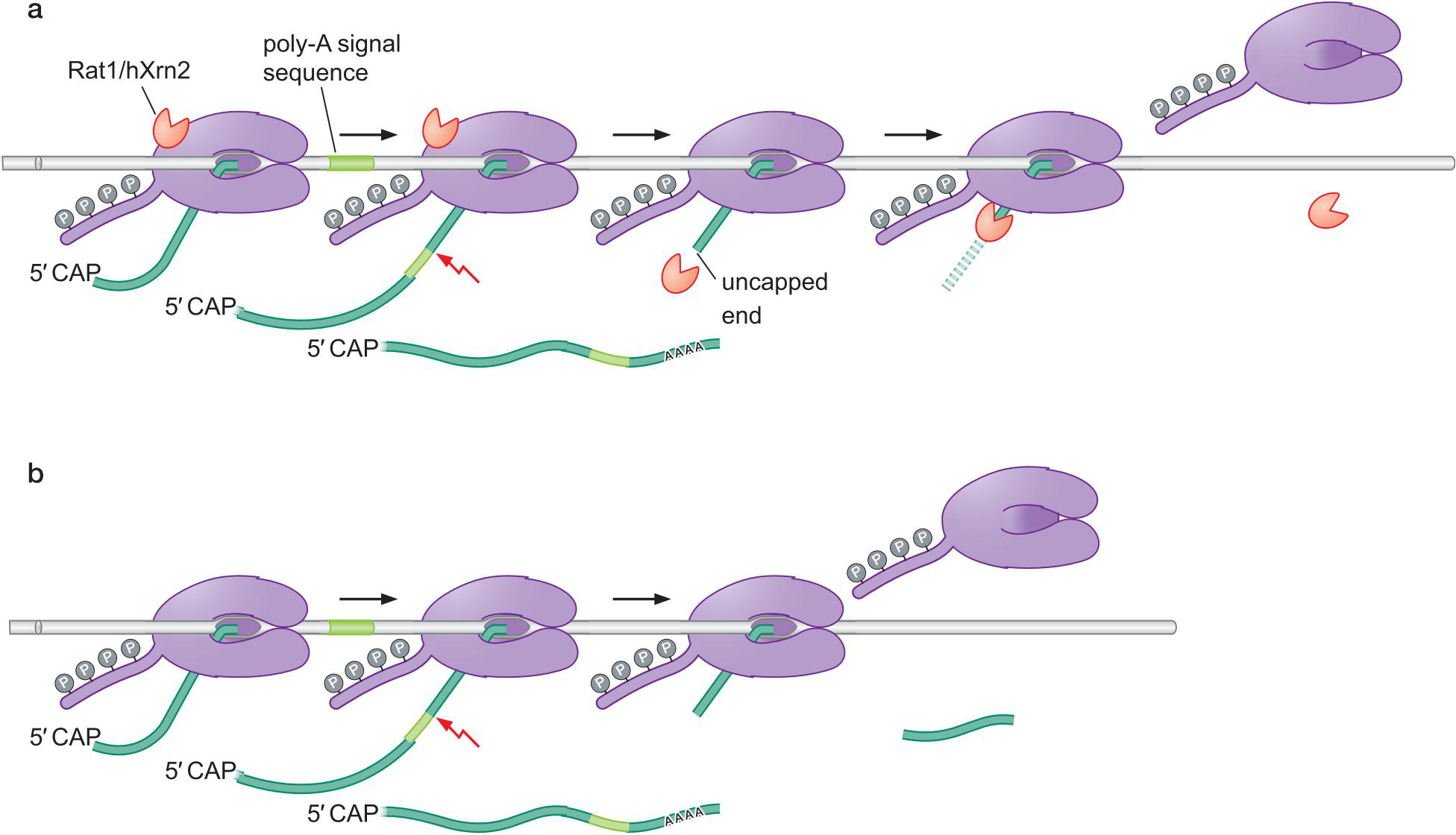
- 鱼雷模型较受认可
- Rat快速消化正在合成的5'没有帽的RNA
- 达到聚合酶时,将RNA链扯出消化
四、由聚合酶I和聚合酶III催化的转录
1、聚合酶I 聚合酶III识别不同的启动子,但仍需TBP
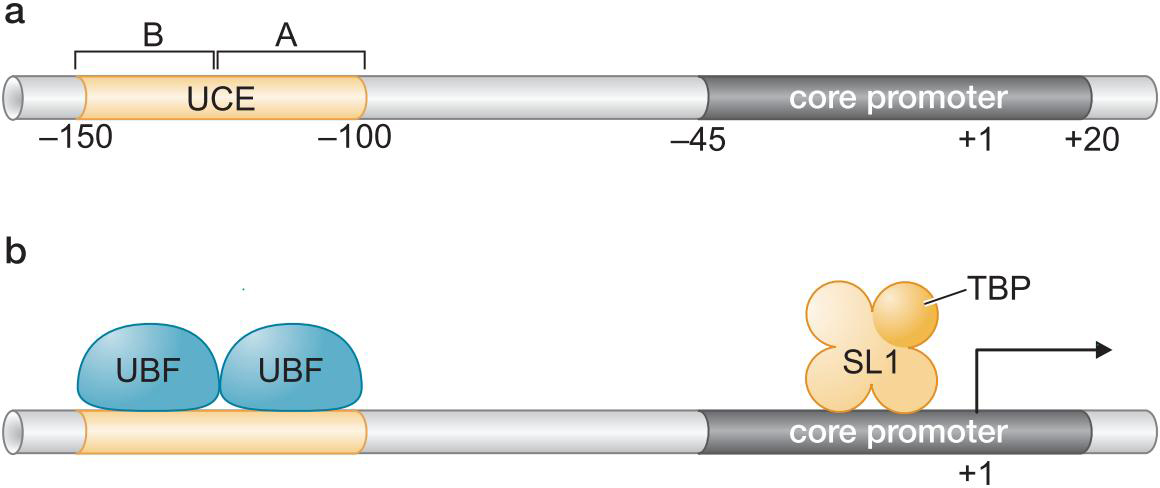
2、Pol I 只用于转录RRNA基因
- UBF结合UCE
- 引入SL1(由TBP与TAF组成)
- 募集Pol I 至核心启动子,激发转录
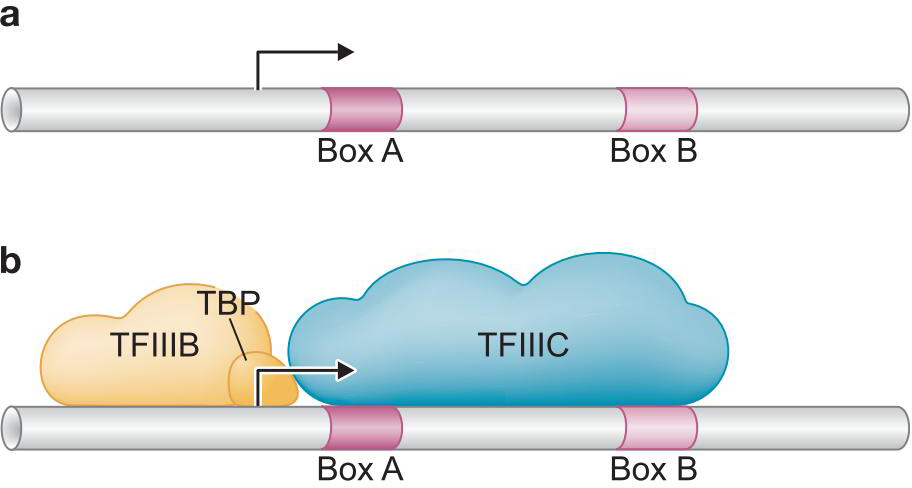
3、Pol III 的启动子位于转录起始点的下游
posted on 2018-10-16 10:58 iojafekniewg 阅读(3943) 评论(0) 编辑 收藏 举报