11.分类与监督学习,朴素贝叶斯分类算法
2020-05-13 21:22 广商吴彦祖 阅读(145) 评论(0) 编辑 收藏 举报1.理解分类与监督学习、聚类与无监督学习。
简述分类与聚类的联系与区别。
分类就是按照数据的属性给对象贴上标签,再根据标签来分类,属于无监督学习,聚类就是指事先定义好类别,然后通过某种度量(比如距离)将他们分类。
监督学习:利用一组已知类别的样本调整分类器的参数,使其达到所要求性能的过程,也称为监督训练或有教师学习。正如人们通过已知病例学习诊断技术那样,计算机要通过学习才能具有识别各种事物和现象的能力。用来进行学习的材料就是与被识别对象属于同类的有限数量样本。
无监督学习则是另一种研究的比较多的学习方法,它与监督学习的不同之处,在于我们事先没有任何训练样本,而需要直接对数据进行建模。这听起来似乎有点不可思议,但是在我们自身认识世界的过程中很多处都用到了无监督学习。比如我们去参观一个画展,我们完全对艺术一无所知,但是欣赏完多幅作品之后,我们也能把它们分成不同的派别。
2.朴素贝叶斯分类算法 实例
利用关于心脏病患者的临床历史数据集,建立朴素贝叶斯心脏病分类模型。
有六个分类变量(分类因子):性别,年龄、KILLP评分、饮酒、吸烟、住院天数
目标分类变量疾病:
–心梗
–不稳定性心绞痛
新的实例:–(性别=‘男’,年龄<70, KILLP=‘I',饮酒=‘是’,吸烟≈‘是”,住院天数<7)
最可能是哪个疾病?
上传手工演算过程。
|
|
性别 |
年龄 |
KILLP |
饮酒 |
吸烟 |
住院天数 |
疾病 |
|
1 |
男 |
>80 |
1 |
是 |
是 |
7-14 |
心梗 |
|
2 |
女 |
70-80 |
2 |
否 |
是 |
<7 |
心梗 |
|
3 |
女 |
70-81 |
1 |
否 |
否 |
<7 |
不稳定性心绞痛 |
|
4 |
女 |
<70 |
1 |
否 |
是 |
>14 |
心梗 |
|
5 |
男 |
70-80 |
2 |
是 |
是 |
7-14 |
心梗 |
|
6 |
女 |
>80 |
2 |
否 |
否 |
7-14 |
心梗 |
|
7 |
男 |
70-80 |
1 |
否 |
否 |
7-14 |
心梗 |
|
8 |
女 |
70-80 |
2 |
否 |
否 |
7-14 |
心梗 |
|
9 |
女 |
70-80 |
1 |
否 |
否 |
<7 |
心梗 |
|
10 |
男 |
<70 |
1 |
否 |
否 |
7-14 |
心梗 |
|
11 |
女 |
>80 |
3 |
否 |
是 |
<7 |
心梗 |
|
12 |
女 |
70-80 |
1 |
否 |
是 |
7-14 |
心梗 |
|
13 |
女 |
>80 |
3 |
否 |
是 |
7-14 |
不稳定性心绞痛 |
|
14 |
男 |
70-80 |
3 |
是 |
是 |
>14 |
不稳定性心绞痛 |
|
15 |
女 |
<70 |
3 |
否 |
否 |
<7 |
心梗 |
|
16 |
男 |
70-80 |
1 |
否 |
否 |
>14 |
心梗 |
|
17 |
男 |
<70 |
1 |
是 |
是 |
7-14 |
心梗 |
|
18 |
女 |
70-80 |
1 |
否 |
否 |
>14 |
心梗 |
|
19 |
男 |
70-80 |
2 |
否 |
否 |
7-14 |
心梗 |
|
20 |
女 |
<70 |
3 |
否 |
否 |
<7 |
不稳定性心绞痛 |
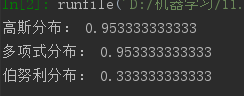
3.使用朴素贝叶斯模型对iris数据集进行花分类。
尝试使用3种不同类型的朴素贝叶斯:
- 高斯分布型
- 多项式型
- 伯努利型
并使用sklearn.model_selection.cross_val_score(),对各模型进行交叉验证。
from sklearn.datasets import load_iris #导入花的数据
from sklearn.naive_bayes import GaussianNB #导入高斯分布
from sklearn.naive_bayes import MultinomialNB #导入多项分布
from sklearn.naive_bayes import BernoulliNB #导入伯努利分布
from sklearn.model_selection import cross_val_score
iris_data= load_iris()
modelG = GaussianNB() # 高斯分布型
modelB = BernoulliNB() # 伯努利型
modelM = MultinomialNB() # 多项式型
score_G = cross_val_score(modelG, iris_data.data, iris_data.target, cv=10)
score_B = cross_val_score(modelB, iris_data.data, iris_data.target, cv=10)
score_M = cross_val_score(modelM, iris_data.data, iris_data.target, cv=10)
print("高斯分布:", score_G.mean())
print("多项式分布:", score_M.mean())
print("伯努利分布:", score_B.mean())