生信工具: DESeq2的原理
DESeq2步骤
DESeq2原理
2014原理论文发布在Genome Biology。
Estimate dispersion的策略:
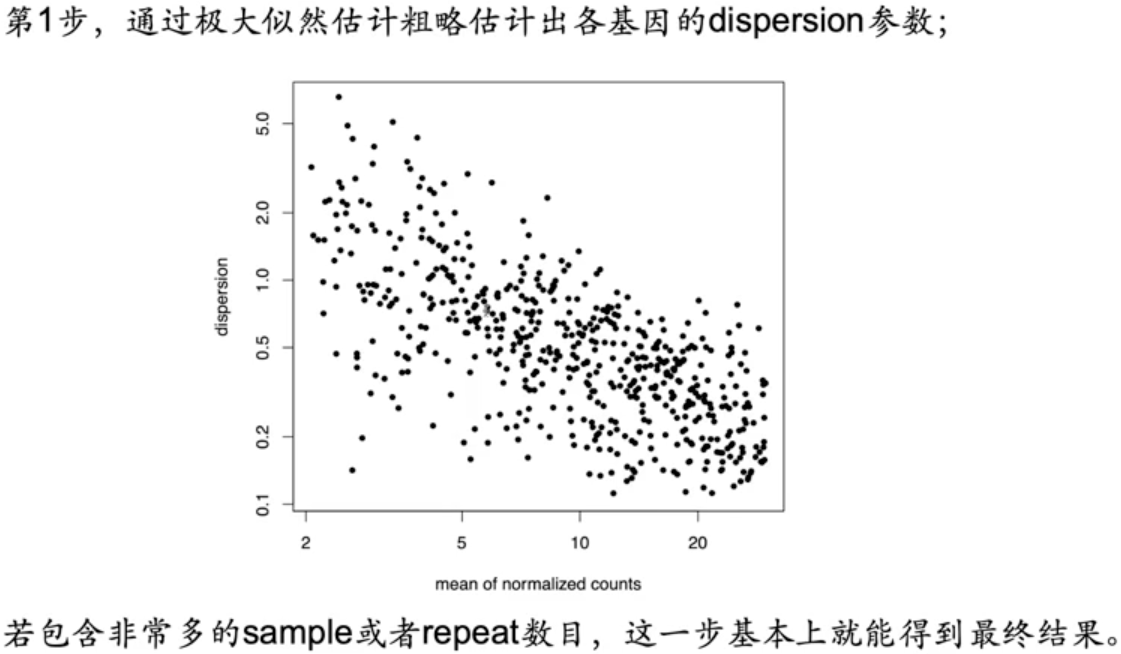
第一步,通过极大似然估计粗略估计出各基因的 dispersion 参数。
(若包含非常多的sample 或者 repeat 数目, 这一步基本就能得到最终结果)
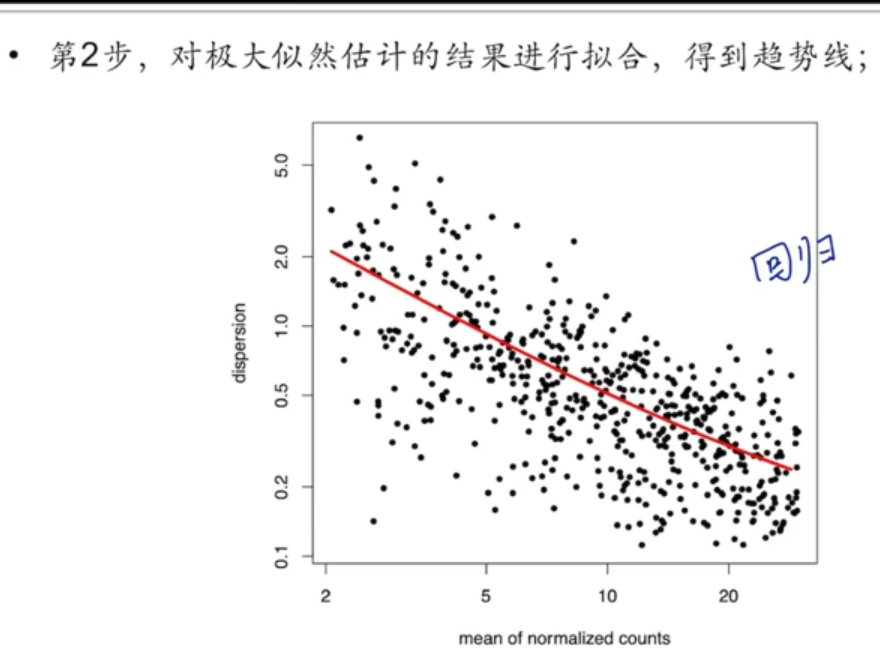
第二步,对极大似然估计的结果进行拟合,得到趋势线。
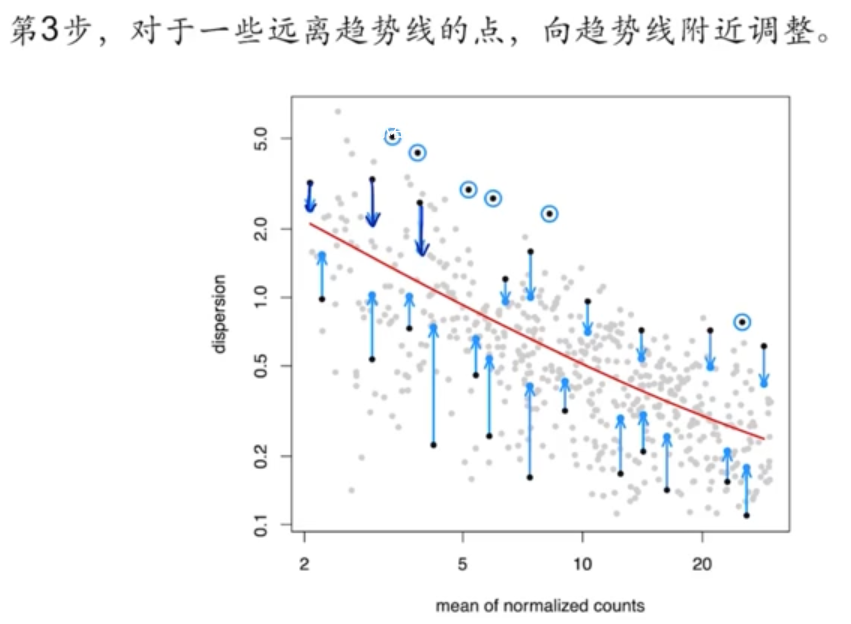
第三步,对于一些远离趋势线的点,向趋势线附近调整。
接下来对上面三步实操:
使用DESeqDataSetFromMatrix获取DESeq2的对象后,如果需要一步获取则使用DESeq函数即可
deseq2.obj = DESeqDataSetFromMatrix(countData = count_df.filter, colData = sample_df, design = ~condition)
deseq2.obj
deseq2.obj = DESeq(deseq2.obj)
可以看到DESeq的输出为:

分别是这么几步:
estimating size factors 做 normalization 用的 RLE 方法
estimating dispersions 通过极大似然估计各基因的 dispersion 参数
gene-wise dispersion estimates 拟合极大似然估计得到的结果,得到趋势线
mean-dispersion relationship 对于一些远离趋势线的点,向趋势线附近调整





【推荐】国内首个AI IDE,深度理解中文开发场景,立即下载体验Trae
【推荐】编程新体验,更懂你的AI,立即体验豆包MarsCode编程助手
【推荐】抖音旗下AI助手豆包,你的智能百科全书,全免费不限次数
【推荐】轻量又高性能的 SSH 工具 IShell:AI 加持,快人一步
· winform 绘制太阳,地球,月球 运作规律
· AI与.NET技术实操系列(五):向量存储与相似性搜索在 .NET 中的实现
· 超详细:普通电脑也行Windows部署deepseek R1训练数据并当服务器共享给他人
· 上周热点回顾(3.3-3.9)
· AI 智能体引爆开源社区「GitHub 热点速览」