学习笔记431—freesurfer下载安装,常用术语和recon-all命令
freesurfer下载安装,常用术语和recon-all命令
1 基础知识
1.1 简介
freesurfer是一个分析和可视化大脑结构成像和功能成像的工具包,可以处理MRI、fMRI数据,进行大脑解剖学数据测量等。
1.2 安装freesurfer
目前该软件包仅支持Linux和Mac OS系统,且官方推荐下载最新版本。官网下载指南
STEP1: 以unbuntu22为例,在该页面选择下载:
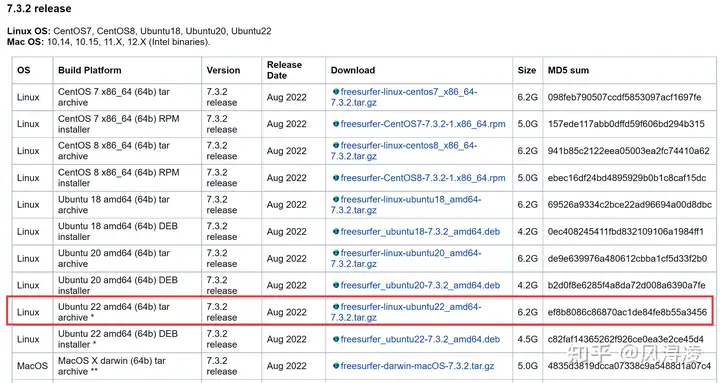
STEP2: 将下载数据移动到"/usr/local"文件夹下,并解压。
sudo mv 上面下载的压缩包名 /usr/local
cd /usr/local
sudo tar xzvf 上面下载的压缩包名STEP3: 在官网注册账户并申请许可证→链接,之后会有一份license.txt文件发送到填写的邮箱里,将其下载并移动到/usr/local/freesurfer文件夹中。
1.3 环境配置
每次打开终端运行之前,首先配置两个参数:FREESURFER_HOME和SUBJECTS_DIR。FREESURFER_HOME指FreeSurfer的位置,SUBJECTS_DIR在运行Freesurfer时索引数据的位置。
export FREESURFER_HOME=/usr/local/freesurfer
export SUBJECTS_DIR=path to your data然后运行setup文件:
cd $FREESURFER_HOME
source SetUpFreeSurfer.sh看到如果配置正确的话,就可以运行其他的命令啦~
1.4 快速上手的一个示例
STEP1: 下载官方tutorial数据集
# 下载数据集
curl https://surfer.nmr.mgh.harvard.edu/pub/data/tutorial_data.tar.gz -o tutorial_data.tar.gz
# 解压和删除压缩包
tar -xzvf tutorial_data.tar.gz
rm tutorial_data.tar.gzSTEP2:
# 解压后的tutorial数据在哪就写到哪
export TUTORIAL_DATA=/path/to/your/tutorial/dir
ls $TUTORIAL_DATA如果看到了类似于以下的输出,则说明环境已配置成功。
buckner_data fsfast-functional
diffusion_recons fsfast-tutorial.subjects
diffusion_tutorial long-tutorialSTEP3:
之后配置subjects dir:
export SUBJECTS_DIR=$TUTORIAL_DATA/buckner_data/tutorial_subjs
cd $SUBJECTS_DIRSTEP4:
复制以下代码
freeview -v \
good_output/mri/T1.mgz \
good_output/mri/wm.mgz \
good_output/mri/brainmask.mgz \
good_output/mri/aseg.mgz:colormap=lut:opacity=0.2 \
-f good_output/surf/lh.white:edgecolor=blue \
good_output/surf/lh.pial:edgecolor=red \
good_output/surf/rh.white:edgecolor=blue \
good_output/surf/rh.pial:edgecolor=red然后可以看到这样一个界面:
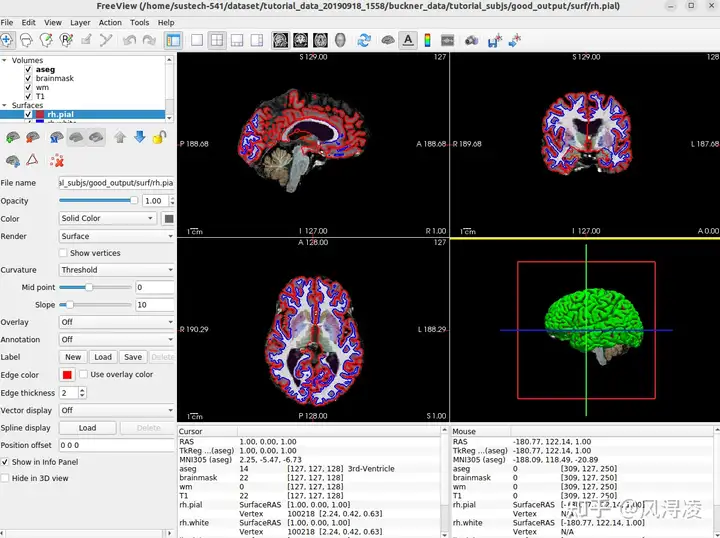
P.S. 上面命令的解释:

P.S. 更详细的更多的上手练习,请见官方教程文档以及官方教学课程
1.5 freesurfer/freeview qt包缺失解决方法
在执行上述freeview命令的时候需要用到qt5包,所以可能会出现一些库缺失导致无法运行的情况。以我自己为例,我缺少两个库:libxcb-xinerama和lib。
解决方法:
1 2 3 4 5 6 7 8 9 10 | 修改配置文件:sudo gedit ~/.bashrc在打开的.bashrc文件末尾最后一行写入export QT_DEBUG_PLUGINS=1然后再重新运行一下bashrc文件:source ~/.bashrc这个时候会详细报错缺少的库,以我为例,安装缺少的包即可:sudo apt install libxcb-xinput0sudo apt install libxcb-xinerama0然后cd回SUBJECTS_DIR再执行上述操作即可。 |
1.6 常用术语
该部分内容撰写参考自视频"Introduction To FreeSurfer Joargon"→链接
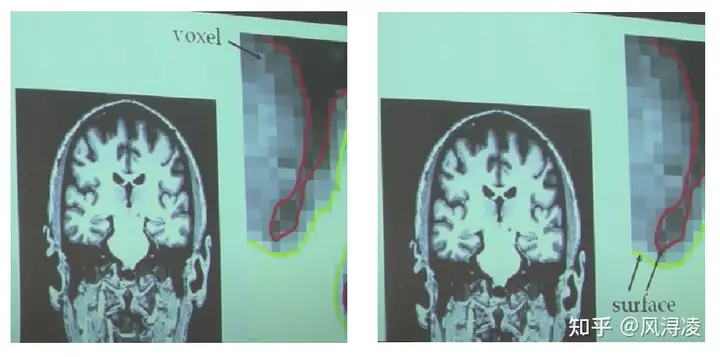
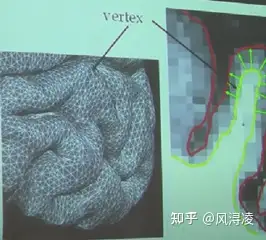
voxel & surface & vertex
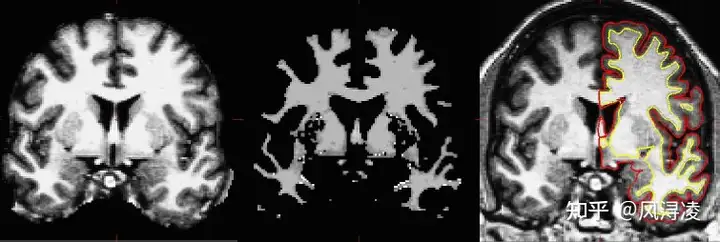
voxel为freesurfer处理的脑数据(MRI, fMRI)中的一个3d最小单元立方体,拥有体积(见下左图);surface意为灰质和白质之间的边界(见下右图黄线,也成为white surface)以及灰质和脑脊髓液之间的边界(见下右图红线,也称为pial surface)。
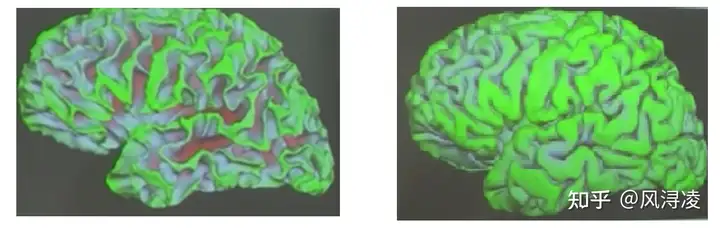
选择不同的surface对可视化的结果也是有一定影响的,如下左图为white surface的可视化,右图为pial surface的可视化。
在基于有限元的方法中,我们用~300000小三角形来近似建模大脑皮层,而每一个小三角形就是一个vertex。vertex对于surface来说,它由很多个vertex组成。
Recon
recon是reconstruction的缩写,执行recon命令可以大致理解为将原始皮层表面数据执行一整遍数据处理流程(详情见下述基于freesurfer的重建流程)。
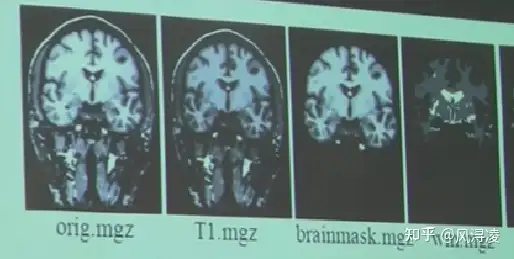
Volumes
称这些类似MRI结构数据格式的数据为volume,他们是freesurfer不同形式的数据输出。
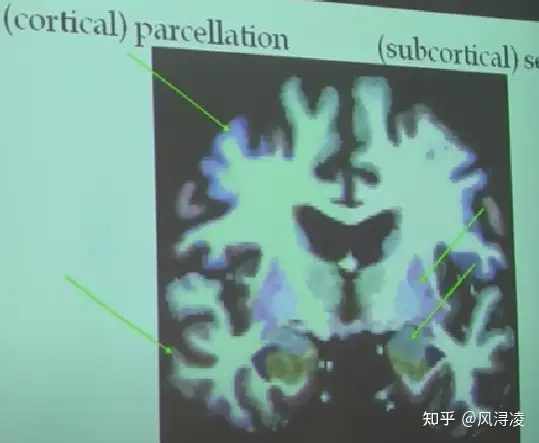
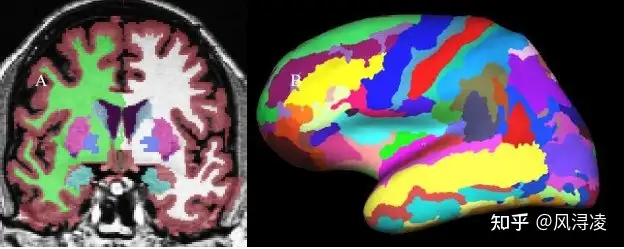
Parcellation & Segmentation
两者都是划分标记不同脑区,本质上没有区别;但是一般对于基于皮层的划分标注叫做parcellation,基于皮层下的划分标注叫做segmentation(见下图,右边遮挡字样应为“segmentation”)。
registration & morph & deform & transforms
大致指代同一个意思,意为对数据作转换。
registration是当在处理两份或以上数量的数据(eg. MRI)时,找到一个它们都可以映射上去的一个共同的坐标系统。映射上去之后,可以在共同的坐标系下对数据进行比较分析。
2 基于freesurfer的重建
2.1 重建流程
网址:RecommendedReconstruction - Free Surfer Wiki (harvard.edu)
Freesurfer分析的流程主要包括表皮和皮层下的解剖学分析。
Surface-based stream
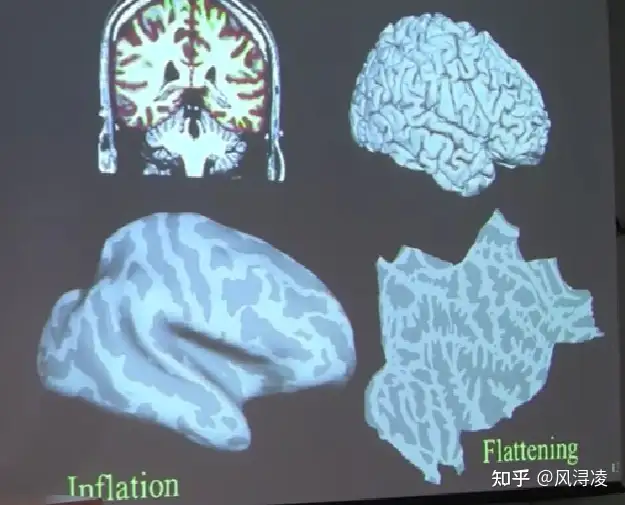
在该分析流程中,freesurfer可以为白质和灰质之间的边界建模,构建出边界之后,就可以做进一步的解剖学测量或分析(比如说测量皮质厚度,获得皮层表面积、曲率、皮层上每个点的法向量等);
除此之外,也可以对表面采用扁平或膨胀的变换方式,基于表面的皮层图谱可以根据特定的方式映射配准到球面上,以获得在不同使用情景下更好的可视化效果或者利于后续的进一步分析。
volume-based (subcortical) stream
该流程做MRI volumes数据的预处理和分类标记不同类别的subcortical tissue,包括基于MNI305坐标的放射配准,以及一个初始体积标记。
进行标签图谱重建的算法同时基于与被试无关的概率图谱(一个预先构建好的概率模型)和特定于被试的测量值(该被试数据本来有的信息),具体方法见论文Automatically Parcellating the Human Cerebral Cortex和Whole brain segmentation : Automated Labeling of Neuroanatomical Structures in the Human Brain,其自动分割的方法已被证明与专业人员得手动分割在统计学上无显著差异。
GUI图形用户交互界面
FreeSurfer有几个交互式图形工具(主要是Freeview),用于数据可视化,分析和管理。该工具具有许多功能,其中包括:可视化 FreeSurfer 输出(表面、体积、ROI、时间过程、叠加层等)、编辑由Freesurfer生成的体积、分割和包裹,以及手动标记 MRI 数据的能力。
2.2 recon命令
2.2.1 概览
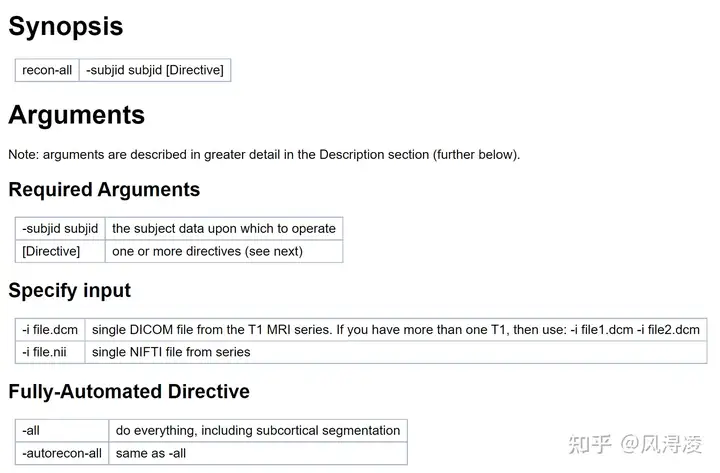
P.S. file.dcm是一个DICOM文件。DICOM既是一种通信协议,也是一种文件格式,这意味着它可以在一个文件中存储医疗信息,例如MRI图像以及患者的信息。该格式可确保所有数据保持在一起,并提供在支持 DICOM 格式的设备之间传输所述信息的能力。
2.2.2 快速上手的例子
recon-all -i file的名字.dcm或file的名字.nii -subject subject的名字 -all参考链接:https://zhuanlan.zhihu.com/p/588468017






【推荐】国内首个AI IDE,深度理解中文开发场景,立即下载体验Trae
【推荐】编程新体验,更懂你的AI,立即体验豆包MarsCode编程助手
【推荐】抖音旗下AI助手豆包,你的智能百科全书,全免费不限次数
【推荐】轻量又高性能的 SSH 工具 IShell:AI 加持,快人一步
· 分享4款.NET开源、免费、实用的商城系统
· 全程不用写代码,我用AI程序员写了一个飞机大战
· MongoDB 8.0这个新功能碉堡了,比商业数据库还牛
· 白话解读 Dapr 1.15:你的「微服务管家」又秀新绝活了
· 上周热点回顾(2.24-3.2)
2020-10-24 学习笔记177—PPT如何保存特定像素级的图片【四种方法】
2020-10-24 学习笔记176—PS 获得一个椭圆的某个部分
2018-10-24 学习笔记28—Python 不同数据类型取值方法
2018-10-24 学习笔记27—python中numpy.ravel() 和 flatten()函数