学习笔记18—circos应用集
一、在线画图(行列分别最大为75)
相信大家都听说过circos图,但是亲自画过的人可能就很少,这主要因为软件的安装和使用稍微有一点麻烦。其实,circos图也是可以在线绘制的,这样就简单多了!一起来了解一下吧!
在circos官网(http://circos.ca/)的最右方有个“CIRCOS ONLINE”选项,这里可以实现在线绘制部分circos图。
打开后界面如下:
以微生物多样性分析中样品与物种丰度circos图绘制为例,给大家讲解circos图的绘制功能。该图能够很直观的反映各样品中不同物种所占的比例,以及物种在不同分组或者样品中的分布关系。
绘制circos图
1.数据准备
首先我们要做的就是准备画图所用到的数据,所用数据为物种在各样品中的相对丰度,这里只选用丰度大于0.01的物种用于绘图,数据如下(列名A、B、C为样品,行名Acetobacteraceae等是科一水平的物种分类):
OTU A B C
Acetobacteraceae 0.506365321669611 0.596887241236994 0.457528142134733
Arcobacteraceae 0.000329490484604467 0.017913387252098 0.000426249200782749
Bacteroidaceae 0.0175209280769342 0.0455871811395347 0.352221339584988
Dysgonomonadaceae 0.00184297424905136 0.0256500300487296 0.0330226880824598
Lachnospiraceae 0.00569185760217826 0.0139020628633905 0.0173870923992018
Lactobacillaceae 0.17495220577586 0.237946115025089 0.0588340146862225
Pseudomonadaceae 0.0021326362135388 0.0286295607092948 0.0127991010016856
Ruminococcaceae 0.00312472844190829 0.00506121976120311 0.0274388235522058
Sphingomonadaceae 0.257860701561278 0.00711394623087561 0.00898610815104722
由于网站要求的数据格式为非负整数,故将所有的数据乘1000(系统会自动截掉小数点后的数据),输入数据则变为:
OTU A B C
Acetobacteraceae 506.365321669611 596.887241236994 457.528142134733
Arcobacteraceae 0.329490484604467 17.913387252098 0.426249200782749
Bacteroidaceae 17.5209280769342 45.5871811395347 352.221339584988
Dysgonomonadaceae 1.84297424905136 25.6500300487296 33.0226880824598
Lachnospiraceae 5.69185760217826 13.9020628633905 17.3870923992018
Lactobacillaceae 174.95220577586 237.946115025089 58.8340146862225
Pseudomonadaceae 2.1326362135388 28.6295607092948 12.7991010016856
Ruminococcaceae 3.12472844190829 5.06121976120311 27.4388235522058
Sphingomonadaceae 257.860701561278 7.11394623087561 8.98610815104722
2.绘图
数据准备好就可以来绘制circos图了,只需要导入数据就可以。
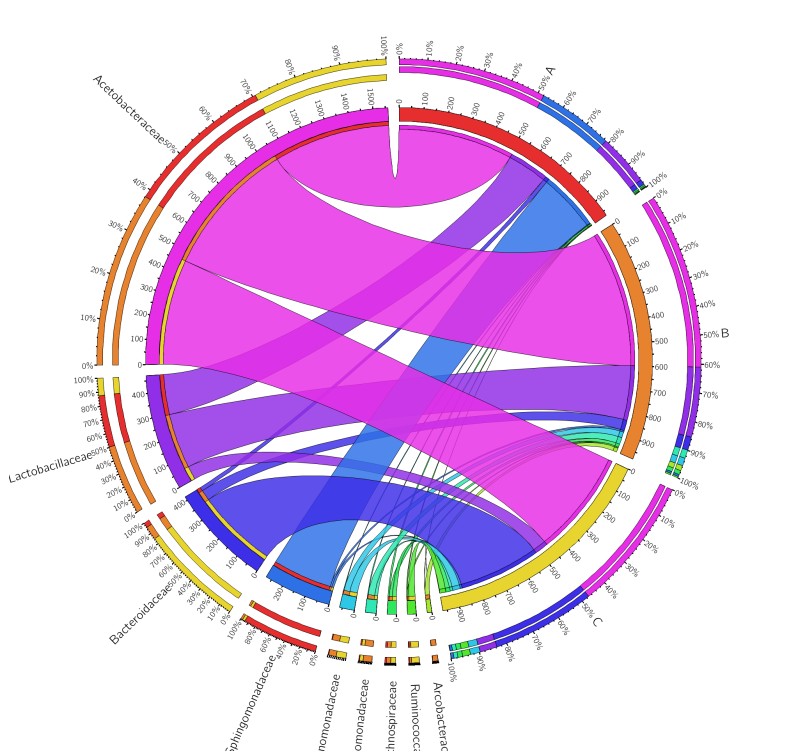
生成的图片如下:
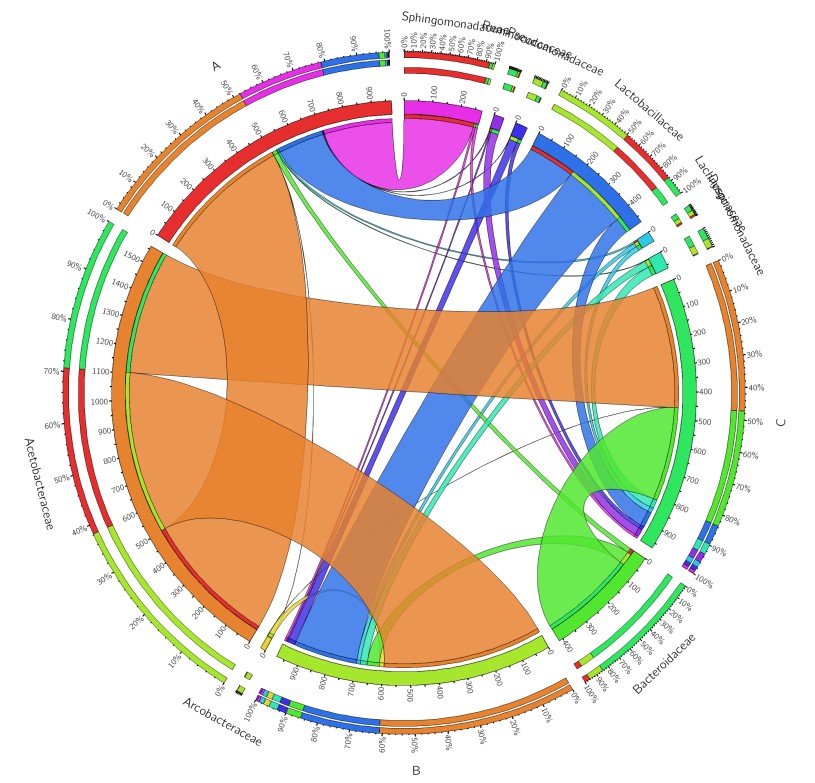
可以看到,图中的物种和样品完全是按照字母顺序排列的,我们希望物种和样品分别位列两边,这里可以人为的对其指定顺序。方法也很简单,就是在数据的第一行和第一列用数字来指定顺序。如下:
OTU OTU 1 2 3
OTU OTU A B C
12 Acetobacteraceae 506.365321669611 596.887241236994 457.528142134733
10 Bacteroidaceae 17.5209280769342 45.5871811395347 352.221339584988
8 Dysgonomonadaceae 1.84297424905136 25.6500300487296 33.0226880824598
6 Lachnospiraceae 5.69185760217826 13.9020628633905 17.3870923992018
11 Lactobacillaceae 174.95220577586 237.946115025089 58.8340146862225
7 Pseudomonadaceae 2.1326362135388 28.6295607092948 12.7991010016856
5 Ruminococcaceae 3.12472844190829 5.06121976120311 27.4388235522058
9 Sphingomonadaceae 257.860701561278 7.11394623087561 8.98610815104722
4 Arcobacteraceae 0.329490484604467 17.913387252098 0.426249200782749
第一行指定了样品的顺序,而第一列按丰度指定物种的顺序。生成图片时要勾选下图红框中的选项(排序所用),不然会报错哦!
新图如下:
图中由于部分物种丰度较低,导致物种名重叠,解决这个问题可以改变文字的布局。这时就需要进行设置了。
3.图片设置
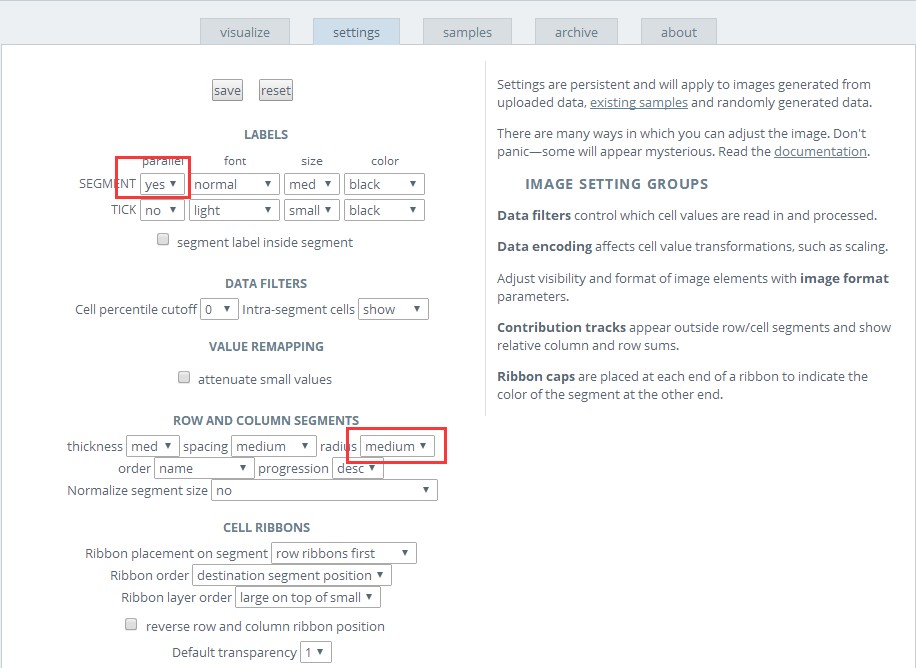
点击"settings"进入设置界面,会有很多的设置选项,可以对图片进行细调。
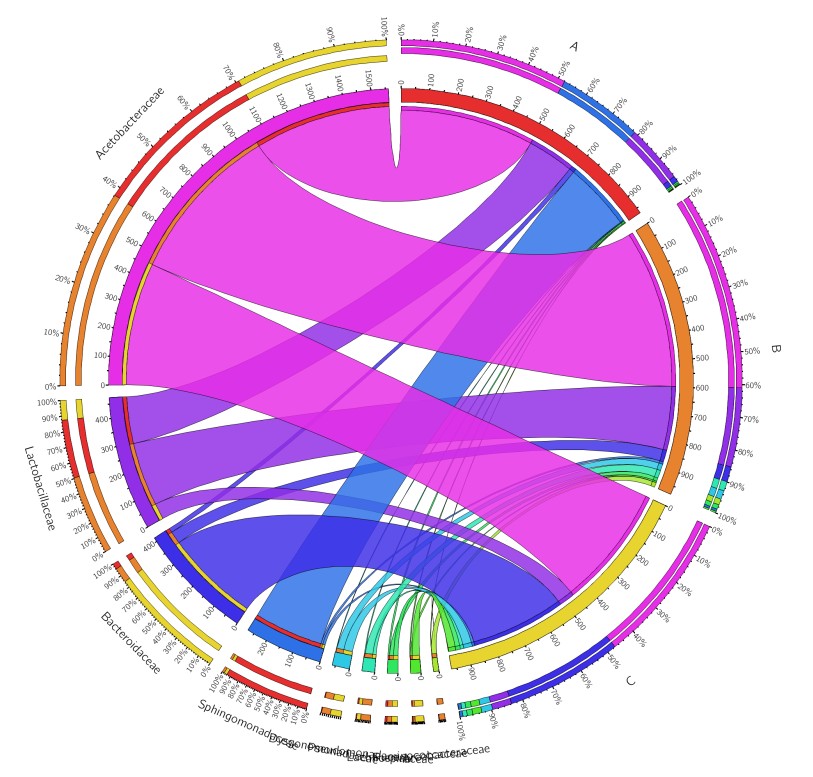
这里只需要修改两个地方即可,将下图第一个红框改为“no”,可以调整文字为垂直布局,避免重叠;但是如果物种名太长,又可能会超出图片范围,所以要缩小圆圈的半径,即将第二个红框改为small。
修改并保存设置后,重新生成图片:
注意:文章摘录自:http://www.omicsclass.com/article/354
二、线下软件画图
(1)染色体组型(karyotypes)是一类特殊的数据。一般的,它保存在名为xx.karyotype.txt文件当中。它将定义染色体的大小,ID,名称和颜色。每一行一条染色体,格式如下
数据格式:
#chr - ID label start end color
chr - hs1 1 0 17000000 chr1
chr - hs2 2 0 217000000 chr2
chr - hs3 3 0 451000000 chr3
...
chr - hs20 3 0 451000000 chr20
最开始的chr表示,这一行将定义一个染色体。然后是一个短线占位符。这个占位符通常用来定义所属关系,对于染色体来说,没有所属。
ID是染色体唯一且不能重复的标识。之后的LABEL是将来用于显示在图上的文本。如果一个染色体组型文件里面包含多个不同来源的染色体组,设置ID最好的办法就是使用前缀。比如hs=homo
sapiens, mm=mus
musculus等等。有时候你可以使用hs19做为前缀来明示数据来源版本。其实,即使是只有一个来源的染色体组,也最好使用前缀,以规范文件格式。
START和END值定义了染色体的大小。对于染色体组型文件,需要指明的是,这里的START和END应该是染色体本身的大小,而不是你想绘制部分的起止位置。指定绘制部分将由其它文件来定义。
COLOR是于定义显示的颜色。如果染色体组不以条纹(cytogenetic
bands)图谱覆盖的话,那么就会以这里设置的颜色显示。对于人类基因组而言,circos预设了与染色体相同的名字做为颜色名,比如chr1,
chr2, … chrX, chrY, chrUn.
效果图:

#Band Domin ID Label Start end color (可要可不要,如果要就和chr-...放入同一个text文件中)
band hs1 p36.33 p36.33 0 10 gneg
band hs1 p36.32 p36.32 10 15 gpos25
band hs1 p36.31 p36.31 15 17 gneg
band hs2 p25.3 p25.3 0 43 gneg
band hs2 p25.2 p25.2 43 70 gpos50
band hs2 p25.1 p25.1 70 217 gneg
band hs3 p25.3 p25.3 0 100 gneg
band hs3 p25.2 p25.2 100 250 gpos50
band hs3 p25.1 p25.1 250 451 gneg
cytogenetic bands的名称例子:1p36.33 其命名规则是之前的数字、字母为染色体代号,一般是数字或者X,Y。而之后会有字母p或者q。p代表短臂,q代表长臂。而每个band都会有颜色深浅的不同,这里主要以gpos和gneg来区别。
效果图:

(2)设置文件
circos的设置文件可以分为多个文件,也可以只写在一个文件里。一般地,为了人们阅读方便,都会写在多个文件,每个文件描述不同的部分,最后由一个主文件来调用就可以了。
这一节,需要设置三个方面,一,数据源,二如何画染色体,三如何画坐标,所以我们也将它分为三个文件,分别名为circos.conf,
ideogram.conf,和ticks.conf。由circos.conf来定义数据源,并调用ideogram.conf和ticks.conf。
circos.conf的内容
| <colors> white =255,255,255 black =0,0,0 blue =0,0,255<<include etc/colors.ucsc.conf>></colors> <fonts><<include etc/fonts.conf>></fonts> <<include ideogram.conf>><<include ticks.conf>> <image><<include etc/image.conf>></image> # specify the karyotype file here karyotype = karyotype.human.hg19.txt chromosomes_units =1000000 chromosomes_display_default = yes <<include etc/housekeeping.conf>> |
这里可以注意到它引入其它文件时使用的是<>的方式。注意,这里include会从circos所在的目录开始寻找,而不是当前目录。我们可以在circos的安装目录当中找到一个名为etc的目录,可以点击查看其中的colors.ucsc.conf,image.conf,以及housekeeping.conf的内容。
在circos当中使用karyotype=xxx语句来定义了karyotype的数据来源。chromosomes_units=1000000定义了基准单位。可以试着把这个值扩大10倍。
接下来我们看ideogram.conf。在ideogram.conf文件中,定义了如何显示染色体组型。
| <ideogram> <spacing> default = 0.005r break = 5u </spacing> #position configuration radius = 0.90r thickness = 100p fill = yes fill_color = black stroke_thickness =2 stroke_color = black #label configuration show_label = yes label_font = bold label_radius = dims(ideogram,radius)+ 0.07r label_with_tag = yes label_size =36 label_parallel =no #band configuration show_bands = yes fill_bands = yes band_stroke_thickness =2 band_stroke_color = white band_transparency =0 </ideogram> |
在ticks.conf当中定义了如何来绘制刻度。
| show_ticks = yes show_tick_labels = yes <ticks> skip_first_label =no skip_last_label =no radius = dims(ideogram,radius_outer) tick_separation = 3p label_separation = 1p multiplier = 1e-6 color = black thickness = 4p size = 20p <tick> spacing = 1u show_label =no thickness = 2p color = dgrey </tick> <tick> spacing = 5u show_label =no thickness = 3p color = vdgrey </tick> <tick> spacing = 10u show_label = yes label_size = 20p label_offset = 10p format=%d grid = yes grid_color = dgrey grid_thickness = 1p grid_start = 0.5r grid_end = 0.999r </tick></ticks> |
好了,有了这些文件,我们就可以让程序跑起来了。
(3)线条参数设置
数据格式:
#hs1 1000000 1700000 hs2 25000000 30000000 (线头就在(1000000+1700000)/2处)
hs17 1000000 1700000 hs1 0 100000000
hs17 1000000 1700000 hs2 0 100000000
hs17 1000000 1700000 hs3 0 100000000
hs17 1000000 1700000 hs4 0 100000000
hs17 1000000 1700000 hs5 0 100000000
hs17 1000000 1700000 hs6 0 100000000
在circos.conf文件中的设置(不同颜色的线条,应该并行的增加下面的句柄):
<links>
<link>
file = F:/BrainAging/result/Picture/circos/segdup.20regions.txt
color = black_a5
radius = 0.98r #线头与环柱之间相对的位置
bezier_radius = 0.01r
thickness = 10
</link>
</links>
/path/to/circos/bin/circos -conf ./circos.conf
参考链接:https://wenku.baidu.com/view/66bca61d8e9951e79a89273f.html;http://blog.sciencenet.cn/blog-442719-758826.html;http://blog.sina.com.cn/s/blog_5b8f74400102w8vy.html
官方链接:http://circos.ca/documentation/tutorials/ideograms/karyotypes/





 浙公网安备 33010602011771号
浙公网安备 33010602011771号