数据科学流程之维数约简
当数据集中包含大量特征时:
- 有些特征包含丰富的预测信息;
- 有些特征之间具有一定的相关性;
- 有些特征只包含噪声或不相关信息;
只保留有意义的特征不仅可以使数据集易于管理,而且可以使预测结果不受数据中噪声的影响,预测精度更好。
维数约简
消除输入数据集的某些特征,创建一个有限特征的数据集(包含所有需要的信息),以更有效的方式预测目标变量。
注:多维数组约简算法的一个主要假设:数据包含加性高斯白噪声。(加性高斯白噪声可以从我们的数据集中分离出来,它是一个线性噪声。)
ps:关于加性高斯白噪声指路博客:https://www.cnblogs.com/jiangkejie/p/10289876.html
维数约简可以减少噪声的集合跨度,以此减少噪声的能量。
一. 协方差矩阵
1. 协方差
协方差是对两个随机变量联合分布线性相关程度的一种度量。
- 两个随机变量越线性相关,协方差越大;
- 线性无关,协方差为零。
随机变量X,Y的协方差:
cov(X,Y) = E[(X-E[X])(Y-E[Y])]
cov(X,Y) = cov(Y,X) (协方差对称)
随机变量X与自身的协方差就是X的方差:
cov(X,X) = E[(X-E[X])(X-E[X])]
即
var(X) = cov(X,X) = E[(X-E[X])²]
2. 相关系数
相关系数(Correlation coefficient)是按积差方法计算,同样以两变量与各自平均值的离差为基础,通过两个离差相乘来反映两变量之间相关程度。
- 相关系数取值在-1到1之间;
- 相关系数为0时,称两个变量不相关;
- 相关系数为1时,称两个变量完全相关,即具有线性关系;
- 越接近于0说明两个变量的相似度越小;
- 越接近于1说明两个变量的相似度越大。
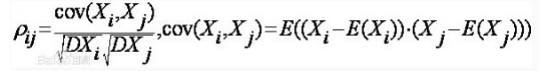
一般地,相关系数>0.8称为高度相关,相关系数<0.3称为低度相关,其他称为中度相关。
 (一.2)
(一.2)
1 from sklearn.datasets import load_iris #载入鸢尾花数据 2 iris = load_iris() 3 print(iris.feature_names) 4 print(iris.data.shape)
['sepal length (cm)', 'sepal width (cm)', 'petal length (cm)', 'petal width (cm)'] (150, 4)
1 import numpy as np 2 cov_data = np.cov(iris.data.T) 3 print(cov_data)
[[ 0.68569351 -0.042434 1.27431544 0.51627069] [-0.042434 0.18997942 -0.32965638 -0.12163937] [ 1.27431544 -0.32965638 3.11627785 1.2956094 ] [ 0.51627069 -0.12163937 1.2956094 0.58100626]]
1 import numpy as np 2 cov_data = np.corrcoef(iris.data.T) 3 print(cov_data)
[[ 1. -0.11756978 0.87175378 0.81794113] [-0.11756978 1. -0.4284401 -0.36612593] [ 0.87175378 -0.4284401 1. 0.96286543] [ 0.81794113 -0.36612593 0.96286543 1. ]]
输出矩阵的第一行第一个是数据集的'sepal length'和本身的协方差/相关系数;第一行第二个是数据集的'sepal length'和'sepal width'的协方差/相关系数;第二行第一个是数据集的
'sepal length'和'sepal width'的协方差/相关系数;第二行第三个是数据集的'sepal width'和'petal length'的协方差/相关系数。。。以此类推,所以会对称。
注:使用协方差矩阵和相关系数矩阵计算相关性的话,cov()和corrcoef()内输入数据必须每行是一个变量(特征),每列是一个样本,所以需要把数据进行转置。(具体算法参考例 一.2 )
cov()/corrcoef()是协方差/相关系数矩阵,是对称矩阵,每个元素是各个向量元素之间的协方差/相关系数。
3. 热力图
矩阵图形化表示——heatmap() 热力图
1 import seaborn as sns #导入seaborn包和可视化绘图工具 2 import matplotlib.pyplot as plt 3 sns.heatmap(cov_data,annot = True)
1 sns.heatmap(cov_data,annot = True,cmap = 'rainbow')
参数annot(annotate标注的缩写)默认取值False;如果是True,在热力图每个方格写入矩阵对应位置数据。
参数cmap从数字到色彩空间的映射。
发现:
- 主对角线元素为1,因为变量自相关系数为1
- 第1和第3,第1和第4,第3和第4特征之间具有高度相关性;
- 第2特征与其他特征相关度较低。潜在特征量应该为2.
相关特征可能包含相似属性,因而可以约简高度相关特征。
维数约减算法很多,如PCA,LFA,LDA,LSA,ICA,T-SNE等。
二. 主成分分析
1.主成分分析
主成分分析(Principal component Analysis, PCA),又称主元分析,主分量分析,旨在利用降维的思想简化数据。
PCA的主要思想是通过对原始变量相关矩阵内部结构的关系研究,找出影响效果某一要素的几个综合指标,使综合指标为原来变量的线性拟合。
将n维特征映射到p维上,这p维是新的正交特征,被称为主成分。
p维特征是在原有m维特征的基础上重新构造出来的,不仅保留了原始变量的主要信息,且彼此间不相关,又比原始变量具有某些更优越的性质。 ——抓住主要矛盾
注:p维是从协方差矩阵上提取出来的,因而具有正交属性
2.PCA算法步骤

PCA(n_components=None, copy=True, whiten=False, svd_solver='full')
参数:
n_components: 所要保留的主成分个数n,缺省时默认为None。赋值为int,如n_components=1,将把原始数据降到一个维度;赋值为string,如n_components='mle',将自动选取特征个数n,使得满足所要求的方差百分比。
copy: 表示是否在运行时将原始训练数据复制一份,缺省时默认为True。若为True,则在原始数据副本上进行运算,原始数据不改变。若为False,则在运行PCA算法后,原始训练数据的值改变。
whiten: 白化,使得每个特征具有相同的方差,缺省时默认False。
svd_solver: 指定奇异值分解SVD的方法,缺省时默认auto。有四个可选择的值:{'auto','full','arpack','randomized'}
返回值:
n_components_: 返回所保留的成分个数n;
components_: 返回具有最大方差的成分;
explained_variancce_ratio_: 返回保留的n个成分各自的方差百分比。
2.1 特征分解
特征分解是奇异值分解的一个特例,一般PCA库都是基于SVD实现。
svd_solver参数:
指定奇异值分解SVD的方法:’auto‘,'full','arpack','randomized'
'full':传统意义上的SVD,使用scipy库对应的SVD实现。
'randomized'和'arpack':一般适用于数据量大,数据维度多,同时主城分数比例又较低的PCA降维,使用了加快SVD的随机算法。
区别:randomized使用scikit-learn的SVD实现;
arpack使用scipy库的sparse的SVD实现。
'auto':PCA算法自行在前面三种中做权衡,选择一个合适的SVD算法来降维。
一般来说,使用默认值就够了。
2.2 Randomized PCA
scikit-learn提供了一种基于随机SVD(Randomized SVD)的更快的算法,它是一种更轻的,近似迭代分解的方法。
虽然使用随机SVD进行满秩重建并不十分理想,其基向量在每次迭代过程中会被局部优化,但是做矩阵分解时,随机SVD比经典SVD算法速度更快,只需要几个步骤就能与经典算法结果极其近似。因此,当训练数据集很大时,它是一个很好的选择。
当数据集的规模非常小时,Randomized PCA输出结果与经典PCA相当接近,但当这两种算法应用于大数据集时,其对比结果会显著不同。
1 from sklearn.decomposition import PCA 2 pca_2c = PCA(n_components=2)#初始化PCA,降为2维 3 X_pca_2c = pca_2c.fit_transform(iris.data)#把数据集里'data'部分进行标准化拟合,得到约简后的数据 4 X_pca_2c.shape
(150, 2)
1 plt.scatter(X_pca_2c[:,0],X_pca_2c[:,1],c=iris.target,alpha=0.8,
s=60,marker='o',edgecolors='white') #plt.scatter参数x,y:表示的是大小为(n,)的数组,也就是我们即将绘制散点图的数据点;c:可以是'color'也可以是一个二维数组(一行的那种);
alpha:0-1之间的实数;s:是一个实数或者是一个数组大小为(n,),这个是一个可选的参数;marker:数据点的形状,默认为'o'(圆点);
edgecolors:数据点边缘颜色
2 plt.show()
1 pca_2c.explained_variance_ratio_
array([0.92461872, 0.05306648])
pca_2c.explained_variance_ratio_.sum()
0.9776852063187949
pca_2c.components_
array([[ 0.36138659, -0.08452251, 0.85667061, 0.3582892 ],
[ 0.65658877, 0.73016143, -0.17337266, -0.07548102]])
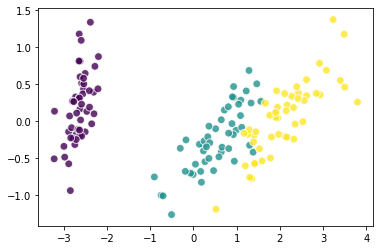
1 pca_2cw = PCA(n_components=2,whiten=True)#初始化PCA,降为2维并且白化 2 X_pca_2cw = pca_2cw.fit_transform(iris.data) 3 plt.scatter(X_pca_2cw[:,0],X_pca_2cw[:,1],c=iris.target,alpha=0.8, 4 s=60,marker='o',edgecolors='white') 5 plt.show()#数值范围变为2倍 6 pca_2cw.explained_variance_ratio_.sum()
0.9776852063187949
1 pca_1c = PCA(n_components=1)#初始化PCA,降为1维 2 X_pca_1c = pca_1c.fit_transform(iris.data) 3 plt.scatter(X_pca_1c[:,0],np.zeros(X_pca_1c.shape),c=iris.target,alpha=0.8, 4 s=60,marker='o',edgecolors='white') 5 plt.show() 6 pca_1c.explained_variance_ratio_.sum()

0.924618723201727
1 pca_95pc = PCA(n_components=0.95)#初始化PCA 2 X_pca_95pc = pca_95pc.fit_transform(iris.data) 3 print(pca_95pc.explained_variance_ratio_.sum()) 4 print(X_pca_95pc.shape)
0.9776852063187949 (150, 2)






【推荐】国内首个AI IDE,深度理解中文开发场景,立即下载体验Trae
【推荐】编程新体验,更懂你的AI,立即体验豆包MarsCode编程助手
【推荐】抖音旗下AI助手豆包,你的智能百科全书,全免费不限次数
【推荐】轻量又高性能的 SSH 工具 IShell:AI 加持,快人一步
· 基于Microsoft.Extensions.AI核心库实现RAG应用
· Linux系列:如何用heaptrack跟踪.NET程序的非托管内存泄露
· 开发者必知的日志记录最佳实践
· SQL Server 2025 AI相关能力初探
· Linux系列:如何用 C#调用 C方法造成内存泄露
· Manus爆火,是硬核还是营销?
· 终于写完轮子一部分:tcp代理 了,记录一下
· 别再用vector<bool>了!Google高级工程师:这可能是STL最大的设计失误
· 震惊!C++程序真的从main开始吗?99%的程序员都答错了
· 单元测试从入门到精通