Note1 基于MNE实现脑电信号的源定位(重建或成像)
写在最前
最开始接触mne还是在20年,那时候它的版本才刚刚开发到0.21。
几年过去他的正式版都已经发布了,而我还依旧是一个学术小白orz。
简单调研一下,发现网上关于mne的教程不多,看到脑机接口社区有推出一系列的epoch的mne教程,几位大佬撰写的mne中文手册,另外还有收费培训班。但作为情绪智能的科研从业者而言,截止目前没有需求去处理epoch的数据分析如ERP等,更多的还是把整段脑电预处理后在下游对接自己的模型或分析代码开发。预处理之前一直是拿EEGLAB实现的。倒是有魔改mne的平面电极拓扑展示,可以方便地在电极平面上展示自己想看的数据分布(颜色条也支持自定义哦),感兴趣的同学可以看这里~。
最近由于方向需要想要借用mne实现脑电的源定位算法,没有看到很好的教程,因此写下博客做一些记录。
环境准备
基于Anaconda的虚拟环境。github上有mne的官方仓库,传送门。仓库中有提到mne需要python版本3.9,因此在创建虚拟环境时要标注清楚。创建虚拟环境等操作都是在cmd命令行进行啦,如果不了解的话需要先调研补充下相关的基础知识。
第一次创建环境时由于安装顺序,库与库之间的依赖有些问题,因此删除虚拟环境并重新开始(虚拟环境牛逼):conda remove -n MNE --all。初次创建可直接忽略这步。这里我使用的环境名是MNE,当然也可以使用别的名字。
首先新建环境:
conda create --name MNE python=3.9
新建完毕后conda activate MNE进入虚拟环境。
pip install --upgrade mne Mark一下,编写教程此时安装的版本是1.6。
看到mne具有非常好看的绘图功能,因此需要安装QT等组件。
pip install PyQt6
安装pyvista以及一些相关的额外的依赖。
pip install pyvista[all]
pip install pyvistaqt
读取MRI数据(好像)时需要nibabel库,因此也给他安上。
pip install nibabel
好的,到这里就安装大成!可以开始运行一些教程了!至少经个人验证,上述环境依赖是足够跑通mne关于源定位的教程滴。此外,mne也提供了api可以方便查看自己目前具有哪些功能相关的依赖,如下所示。
import mne
print(mne.sys_info())
源成像/定位/重建(Source Imaging/Localization/Reconstruction)
根据文献,源成像过程总共可分为如下几步1:
- 基于个体和模板MRI生成头模型和源模型
- 将头模型、源模型、EEG/MEG传感器进行配准,计算导联场矩阵
- 使用EEG/MEG信号与导联场矩阵,选择合适的逆问题算法进行源成像
接下来,我们也将结合mne的代码来复现这几步。
教程实现与理解
通常来讲,我们需要对每个个体的MRT T1数据(大概就是脑皮层的结构数据),结合传感器分布进行针对性的配准,但事实上不是每个团队都既有EEG又有fMRI设备的。因此我们不能奢求太多,有个标准模板能凑合用就不错了。至于有没有效果,试了才知道。想要直接查看原始教程的,可以参考[EEG forward operator with a template MRI](EEG forward operator with a template MRI — MNE 1.6.0 documentation)与[Source localization with MNE, dSPM, sLORETA, and eLORETA](Source localization with MNE, dSPM, sLORETA, and eLORETA — MNE 1.6.0 documentation)。
使用自有数据的montage
我手边现有的电极空间数据有两份。一份是从neuroscan官网上下载的Scan-SynAmps2-Quik-Cap64, 一份是Curry8导出数据中sensor文件转为Quik_Cap_Neo.mat存储。这里提供了电极文件,data_load.py中也提供了我导入数据的方式。两份电极文件所对应的设备均为Neuroscan64导,并且删除了M1M2(因为我自己使用双侧乳突进行了重参考)。两种电极空间分布有一定差异,比如后者有前者没有的FT11,FT12。
直接使用上述电极导入会存在montage空间位置与头骨匹配不准的问题。我们将在后续看到成功配准的效果图。经实践,确认原因为电极空间坐标量纲不同,可能是米级或厘米级的区别。需对自有电极数据空间坐标单位调整,以下为实践结果。
read_dat函数中需缩小100倍,需对如下语句进行调整:
pos = np.array([float(item) for item in items[1:]]) / 100
read_montagemat函数调整如下。nasion, lpa, rpa 由于curry8没有提供,所以直接照搬的另一份的。因此此处须调整量纲两次。
nasion = np.array([0.100031, 9.518287, 0]) / 100
lpa = np.array([-6.902262, 0, 0]) / 100
rpa = np.array([6.902262, 0, 0]) / 100
channels[key] = sensorpos[i] / 1000
参数组调整后可视化展示时,发现电极空间坐标小红点会与头皮有一定偏移,两种电极坐标都有。后发现可视化函数mne.viz.plot_alignment中有一个参数eeg:eeg=dict(original=0.2, projected=0.8)或者eeg=["original", "projected"]。original即为小红点,projected即为自动配准到头皮的传感器,正如红色圆柱体在图中一样。这个图的实现函数会在后面展开。
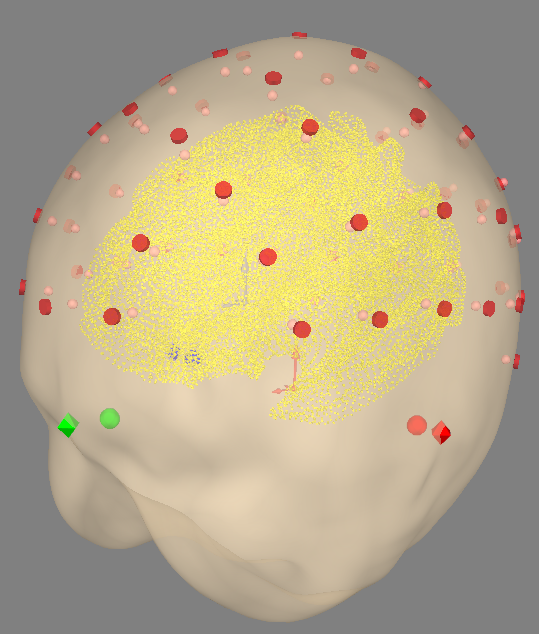
Montage认识强化
在MNE库中,nasion,lpa和rpa是用于定位电极的三个关键参考点,如上图的小绿点即为鼻根:
- nasion(鼻根,Nz):这是鼻子与前额交界处的凹陷部分,位于两眼之间。
- lpa(左耳前凹,LPA):这是左耳的前部,位于耳垂上方的凹陷处。
- rpa(右耳前凹,RPA):这是右耳的前部,位于耳垂上方的凹陷处。
(Bing生成)这三个点通常在实验开始时通过一种称为数字化的过程来获取,该过程使用特殊的设备(如Polhemus FastSCAN)来精确测量参考点和电极的位置。这些位置信息随后可以用于将电极数据与头部模型对齐,从而进行源定位等高级分析。在导入自有数据时,你需要提供这三个参数的坐标。这些坐标通常是在实验开始时通过数字化设备获取的。如果你没有这些坐标,你可能需要手动测量它们,或者使用一些估计方法来获取。
核心实现流程
MNE提供了标准模板,即被试fsaverage。首先抄袭教程如下语句。导入 transformation, sourcespace, bem(这些我也还不清楚是啥)。有的可能是从freesurfer(一款MRI处理软件好像)导入的。如果首次执行会下载相关配置文件到操作系统目录C:\Users\Your user \mne_data:
import os.path as op
import numpy as np
import mne
from mne.datasets import fetch_fsaverage
# Download fsaverage files
fs_dir = fetch_fsaverage(verbose=True)
subjects_dir = op.dirname(fs_dir)
# The files live in:
subject = "fsaverage"
trans = "fsaverage" # MNE has a built-in fsaverage transformation
src = op.join(fs_dir, "bem", "fsaverage-ico-5-src.fif")
bem = op.join(fs_dir, "bem", "fsaverage-5120-5120-5120-bem-sol.fif")
原始教程中使用提供的数据集eegbci进行。此处使用自定义的数据生成一个mne raw类,并配置数据与info文件。
from utils import read_dat
montage = read_dat("你的绝对路径\\"
"Scan-SynAmps2-Quik-Cap64\\SynAmps2 Quik-Cap64NoM1M2.DAT")
info = mne.create_info(montage.ch_names,200, "eeg") # 采样率为200
data = "输入你的数据读取方式" # data.shape [62, times]
raw = mne.io.RawArray(data, info)
raw.set_montage(montage)
raw.set_eeg_reference(projection=True) # 已经在M1M2 reference过了,但后面还是需要reference 不懂
通过下述语句可以检查电极传感器配准情况,也可以跳过。
# Check that the locations of EEG electrodes is correct with respect to MRI
mne.viz.plot_alignment(
raw.info,
src=src,
eeg=["original", "projected"],
trans=trans,
show_axes=True,
mri_fiducials=True,
dig="fiducials",
)
计算正向算子。
fwd = mne.make_forward_solution(
raw.info, trans=trans, src=src, bem=bem, eeg=True, mindist=5.0, n_jobs=None
)
print(fwd)
计算噪声协方差、逆向因子,并应用逆向因子。此处使用的方法为dSPM。根据官方教程可更换为其他的逆向求解算法。
# 噪声协方差
noise_cov = mne.compute_raw_covariance(raw)
# 逆向因子
from mne.minimum_norm import make_inverse_operator
inverse_operator = make_inverse_operator(
raw.info, fwd, noise_cov, loose=0.2, depth=0.8
)
del fwd
# 逆向因子应用
from mne.minimum_norm import apply_inverse_raw
method = "dSPM"
snr = 3.0
lambda2 = 1.0 / snr**2
stc = apply_inverse_raw(
raw,
inverse_operator,
lambda2,
method=method,
pick_ori=None,
verbose=True,
)
到这里就求解完啦!生成的stc就是类SourceEstimate,源估计。
逆向求解得到的数据存储于stc.data中。
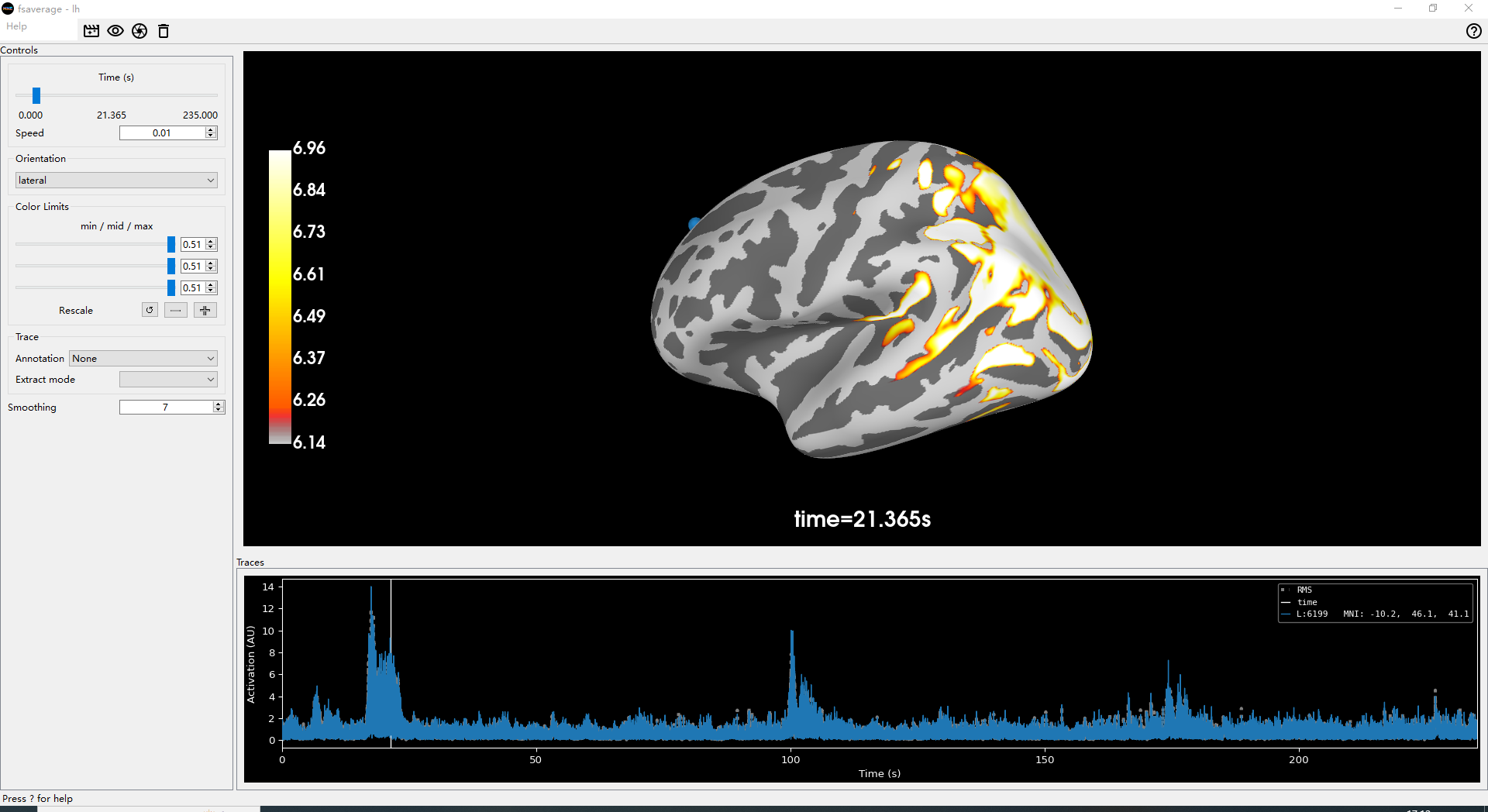
可使用如下命令对源估计进行可视化。不得不说,mne可视化做的是真好。可视化效果如下图所示。
initial_time = 0.1
brain = stc.plot(
subjects_dir=subjects_dir,
subject=subject,
initial_time=initial_time,
smoothing_steps=7,
)
源估计的保存
此外,由于针对一个影片(~4 min)的脑电片段生成source time course耗时约5min左右,因此我们最好能将其保存下来(较大警告)。
stc.save('test_stc')
stc = mne.read_source_estimate('test_stc')


 浙公网安备 33010602011771号
浙公网安备 33010602011771号