knn学习笔记
#knn算法学习笔记
1.定义
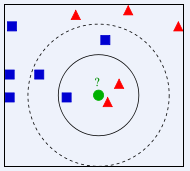
图片来自https://baike.baidu.com/item/%E9%82%BB%E8%BF%91%E7%AE%97%E6%B3%95/1151153?fr=aladdin
K值若较小,这里取内圈,红色三角占2/3,则绿色的未知物被判断为红色三角;
K值若较大,这里取外圈,蓝色方块占3/5,则绿色未知物被判断为蓝色方块。
> for(i in 1:round(sqrt(dim(train)[1]))) { + pre_result <- knn(train=train,test=test,cl=train_lab,k=i) + Freq <- table(pre_result,test_lab) + print(1-sum(diag(Freq))/sum(Freq)) #误差率 + } [1] 0.08219178 [1] 0.06849315 [1] 0.08219178 [1] 0.06849315 [1] 0.05479452 [1] 0.06849315 [1] 0.05479452 [1] 0.09589041 [1] 0.04109589
一般来说就是找误差率最小的k
k-nn中的分类决策规则往往是多数表决,即输入实例的k个近邻的训练实例中的多数类决定输入实例的类。(少数服从多数)
以UCI数据库中的鸢尾花数据集举例,UCI地址:http://archive.ics.uci.edu/ml/index.php,不过iris是R自带的数据集,可以直接加载使用。
4.1 包
R中有许多包都可以实现knn算法,以下以class包、DMwR包和kknn包为例进行说明。
4.2 class包中的knn函数
主要步骤:
knn(train, test, cl, k = 1, l = 0, prob = FALSE, use.all = TRUE)
train:指定训练样本集
test :指定测试样本集
cl :指定训练样本集中的分类变量
k :指定最邻近的k个已知分类样本点,默认为1
l :指定待判样本点属于某类的最少已知分类样本数,默认为0
prob:设为TRUE时,可以得到待判样本点属于某类的概率,默认为FALSE
use.all:控制节点的处理办法,即如果有多个第K近的点与待判样本点的距离相等,默认情况下将这些点都纳入判别样本点,当该参数设为FALSE时,则随机挑选一个样本点作为第K近的判别点
> # z-score数据标准化 > iris_scale <- scale(iris[-5]) > train <- iris_scale[c(1:25,50:75,100:125),] #训练集 > test <- iris_scale[c(26:49,76:99,126:150),] #测试集 > train_lab <- iris[c(1:25,50:75,100:125),5] > test_lab <- iris[c(26:49,76:99,126:150),5] > pre <- knn(train=train,test=test,cl=train_lab,k=round(sqrt(dim(train)[1])),prob = F) > table(pre,test_lab) test_lab pre setosa versicolor virginica setosa 24 0 0 versicolor 0 24 3 virginica 0 0 22
4.3 DMwR包中的Knn
kNN(form, train, test, norm = T, norm.stats = NULL, ...)
form:分类模型
train:指定训练样本集
test:指定测试样本集
norm:布尔值,指示是否在KNN预测前将数据标准化,默认为TRUE
norm.stats:默认FALSE,采用scale()进行标准化,也可提供其他标准化方法(标准化方法很多的,scale只是其中一种)
> train<-iris[c(1:25,50:75,100:125),] #训练集 > test<-iris[c(26:49,76:99,126:150),] #测试集 > pre2 <- kNN(Species~.,train,test,norm=T,k=round(sqrt(dim(train)[1]))) > table(pre2,test$Species) pre2 setosa versicolor virginica setosa 24 0 0 versicolor 0 24 3 virginica 0 0 22
4.4 kknn包中的kknn
kknn()函数的语法和参数如下:
kknn(formula = formula(train),train, test, na.action = na.omit(), k= 7, distance = 2, kernel = "optimal", ykernel = NULL, scale=TRUE, contrasts= c('unordered' = "contr.dummy", ordered ="contr.ordinal"))
formula一个回归模型:分类变量~特征变量;
train指定训练样本集;
test指定测试样本集;
na.action缺失值处理,默认为去掉缺失值;
k近邻数值选择,默认为7;
distance闵可夫斯基距离参数,p=2时为欧氏距离;
其他参数略,详情可以查询kknn包的说明> train<-iris[c(1:25,50:75,100:125),] #训练集 > test<-iris[c(26:49,76:99,126:150),] #测试集 > # 调用kknn > pre3 <- kknn(Species~., train, test, distance = 1, kernel = "triangular") > # 获取fitted.values > fit <- fitted(pre3) > table(fit,test$Species) fit setosa versicolor virginica setosa 24 0 0 versicolor 0 22 4 virginica 0 2 21
其实knn实现过程,每个包里面的knn函数差别不太大,参数的设置的时候有一定的差别,这可能是,写包的人的语言风格不太一样吧



【推荐】编程新体验,更懂你的AI,立即体验豆包MarsCode编程助手
【推荐】凌霞软件回馈社区,博客园 & 1Panel & Halo 联合会员上线
【推荐】抖音旗下AI助手豆包,你的智能百科全书,全免费不限次数
【推荐】轻量又高性能的 SSH 工具 IShell:AI 加持,快人一步