NGS数据格式介绍
一般情况下,从Illumina平台上得到的测序,其数据格式是Fastq格式,可以称之为原始数据(Raw data)。事实上直接的下机数据是显微拍摄得到的图像信息。但是一般都会用Bcl2Fastq软件将图像信息转化成Fastq文件。
如果测序是SE:则只有一个fastq文件,如果是PE测序,则得到两个Fastq文件。
PE的数据特点有:
(1)两个Fastq文件中分别包含数据1和2,来区分前后端;
(2)这2个文件的行数必须一致;
(3)相同的行上的数据来自同一条DNA片段双末端的测序数据;
(4)Fastq以每4行为一个单位,表示一条reads的信息。
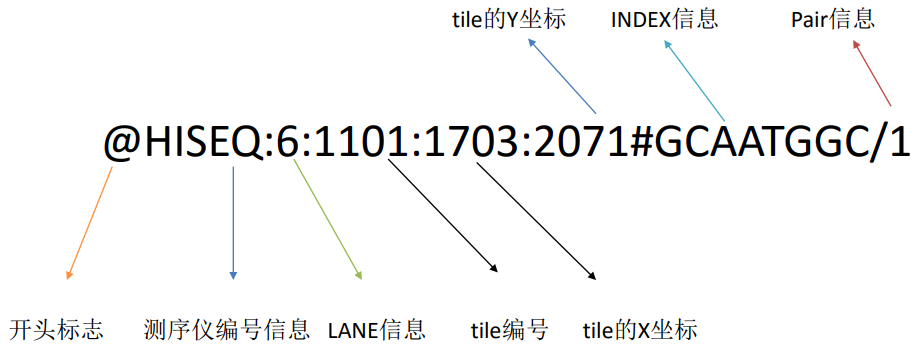
@HISEQ:6:1101:1703:2071#GCAATGGC/1AGAATGCGTCATTCTGCGGAACTCATCCGACTGAATACCGAAAAGCAGAATCTGATCCTGGTTTCTGCCATAAAGTAGCGCGAGCACACAGACGTCTGCGCGCCTGCGGTGACGGCGGTGCGGGT+`\```fdbeaeddf]d_ffNddPP\dedd]N[XPdffP\NeNdbff]faeafPdeff]PbPPP[efP^YePY\edfefO[NNNbcM_effc\OcfcOWbffffMXcaMcffa_cYcYYbccYM]b
第一行 序列名称
第二行 序列的碱基组成
第三行 序列信息,或者直接以“+”做标记
第四行 碱基的质量
现在的Illimina使用的质量格式为Phred+33,和Sanger的碱基质量基本一致;碱基质量使用Q(Phred值)表示,其计算公式为:
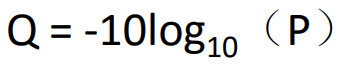
碱基质量与错误率的关系为:




 浙公网安备 33010602011771号
浙公网安备 33010602011771号