PLEK升级了:PLEKv2工具在RNA序列分析中的卓越表现
摘要:
使用 PLEKv2 识别鉴定lncRNA,只需要输入RNA的序列(fa文件)即可。
在生物信息学领域,长非编码RNA(lncRNA)和信使RNA(mRNA)的准确区分对于理解基因调控机制至关重要。随着深度学习技术的兴起,我们迎来了PLEKv2——PLEK工具的全新升级版,它在RNA序列分类精度方面取得了显著提高。这里探讨PLEKv2的技术亮点、实现细节以及在多样化数据集上的应用表现,并展望其在未来研究中的潜在影响。PLEKv2软件可以在https://sourceforge.net/projects/plek2/上免费获取。
关键词: PLEKv2, RNA序列分类,深度学习,生物信息学,跨物种预测
一、引言
随着高通量测序技术的飞速发展,生物信息学正面临着前所未有的数据量和复杂性挑战。RNA序列的分类作为基因功能研究的基础,其准确性直接影响后续的生物学解释和应用。PLEKv2的问世,正是为了应对这一挑战,通过深度学习技术提升RNA序列的分类效率和准确性。
二、PLEKv2技术亮点
高准确率: PLEKv2在人类数据集上达到了98.7%的预测准确率,这一成绩在同类工具中遥遥领先。
跨物种预测: 该工具不仅适用于人类,还能进行跨物种的RNA序列预测,显示出良好的泛化能力。
植物数据适用性: PLEKv2在植物数据集上同样表现出色,
Coding-Net模型: 该工具采用了创新的Coding-Net模型,结合k-mer频率和ORF长度特征,为RNA序列分类提供了新的视角。
三、实现细节
PLEKv2的技术实现涵盖了数据预处理、特征提取、深度学习模型构建和超参数调优等多个环节:
数据预处理: 如图1。
数据收集:首先从公共数据库如GENCODE和RefSeq获取人类lncRNA和mRNA的序列数据。
数据清洗:去除序列长度不足200个核苷酸的短序列。
替换序列中的'U'(尿嘧啶)为'T'(胸腺嘧啶),因为在DNA中通常使用'T'表示胸腺嘧啶。符号标准化:将序列中所有混合碱基符号(如'R', 'Y', 'M', 'K', 'S', 'W', 'H', 'B', 'V', 'D'和'N')替换为'N',表示不确定的碱基。
序列平衡:为了确保模型不会因为某一类别的样本数量过多而产生偏差,对lncRNA和mRNA的样本数量进行随机抽样,以保持两者数量相等。
特征计算:计算加权k-mer频率,k-mer是长度为k的核苷酸序列模式,PLEKv2中对不同长度的k-mer(通常是1到6)出现的频率进行统计和加权。
计算开放阅读框(ORF)长度:ORF是DNA或RNA序列中可能编码蛋白质的部分,PLEKv2通过寻找起始密码子(ATG)和终止密码子来确定ORF的长度,并进行归一化处理。
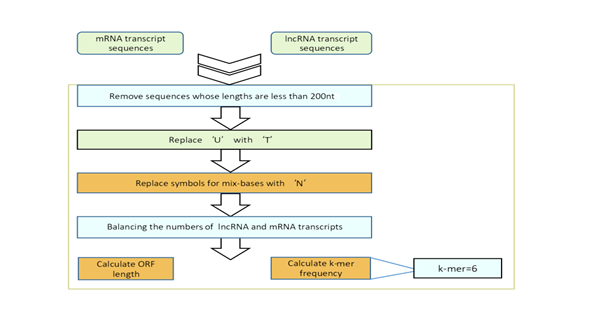
图1 数据预处理
特征提取: PLEKv2利用k-mer频率和ORF长度构建了特征向量,为深度学习模型提供了丰富的输入信息。
深度学习模型: 如图2所示,PLEKv2采用了卷积神经网络(CNN)和全连接层,有效提取并学习了RNA序列的特征。
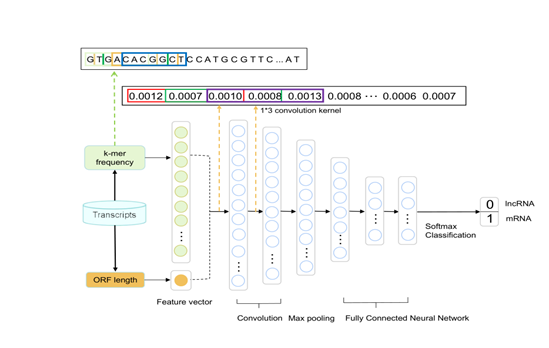
图2 网络模型
超参数调优: PLEKv2通过细致的超参数调优,进一步提升了模型的性能和泛化能力。
四、应用表现
PLEKv2在多个层面上展现了其强大的应用潜力:
如表1所示,PLEKv2在人类数据集上的预测准确率达到了98.7%,这一结果显著高于其他传统工具和一些早期的深度学习模型。这种高准确率意味着PLEKv2能够非常可靠地区分长非编码RNA(lncRNA)和信使RNA(mRNA)。
与其他现有的lncRNA和mRNA识别工具相比,如CPC2、CNCI、Wen等人的CNN、LncADeep、PLEK和NcResNet,PLEKv2在多项评估指标上均表现出更高的性能。
PLEKv2使用基于k-mer频率和校准ORF长度的特征向量,这些特征向量在人类数据集上显示出极高的区分能力。特别是当k=6时,模型的准确率显著提高。
在人类数据集上的测试表明,PLEKv2不仅在训练集上表现良好,而且在独立的测试集上也能保持高准确率,这证明了模型的泛化能力。PLEKv2在保持高准确率的同时,还展现出了较高的计算效率。它在处理时间和内存使用方面都优于许多其他工具,这使得PLEKv2在实际应用中更为实用。
表1 多个模型对比
|
Models |
Precision |
Recall |
F1score |
Accuracy |
|
CPC2 |
0.942 |
0.856 |
0.897 |
0.906 |
|
CNCI |
0.914 |
0.975 |
0.944 |
0.950 |
|
CNN |
0.792 |
0.821 |
0.806 |
0.821 |
|
LncADeep |
0.960 |
0.980 |
0.970 |
0.973 |
|
PLEK |
0.962 |
0.941 |
0.938 |
0.938 |
|
PLEKv2 |
0.986 |
0.986 |
0.986 |
0.987 |
|
NcResNet |
0.492 |
0.498 |
0.496 |
0.498 |
跨物种预测: 如表2所示,PLEKv2在灵长类动物数据集上显示出良好的泛化能力,准确率高于其他工具。
表2 灵长类动物数据集上测试结果
|
Species |
Tool |
Precision |
Recall |
F1score |
Accuracy |
|
Pan troglodytes |
CPC2 |
0.755 |
0.938 |
0.837 |
0.879 |
|
|
CNCI |
0.849 |
0.899 |
0.873 |
0.913 |
|
|
LncADeep |
0.870 |
0.939 |
0.903 |
0.934 |
|
|
PLEK |
0.842 |
0.872 |
0.856 |
0.904 |
|
|
PLEKv2 |
0.873 |
0.940 |
0.905 |
0.935 |
|
|
NcResNet |
0.343 |
0.532 |
0.417 |
0.511 |
|
Macaca mulatta |
CPC2 |
0.954 |
0.902 |
0.927 |
0.926 |
|
|
CNCI |
0.937 |
0.966 |
0.951 |
0.945 |
|
|
LncADeep |
0.968 |
0.913 |
0.944 |
0.932 |
|
|
PLEK |
0.882 |
0.885 |
0.883 |
0.873 |
|
|
PLEKv2 |
0.948 |
0.957 |
0.952 |
0.952 |
|
|
NcResNet |
0.544 |
0.489 |
0.516 |
0.503 |
|
Gorilla gorilla |
CPC2 |
0.998 |
0.917 |
0.955 |
0.918 |
|
|
CNCI |
0.998 |
0.874 |
0.932 |
0.874 |
|
|
LncADeep |
0.999 |
0.905 |
0.950 |
0.905 |
|
|
PLEK |
0.999 |
0.838 |
0.911 |
0.838 |
|
|
PLEKv2 |
0.999 |
0.922 |
0.959 |
0.922 |
|
|
NcResNet |
0.981 |
0.525 |
0.684 |
0.525 |
植物数据集: 在植物数据集上,PLEKv2的准确率超过95%,如表3所示,证明了其在植物RNA序列分类上的优越性。
表3 植物数据集上测试结果
|
Species |
Dataset type |
Number of transcripts |
CPC2 |
PLEK |
PLEKv2 |
|
Arabidopsis thaliana |
Coding |
388 |
85.90% |
60.2% |
95.7% |
|
|
Non-coding |
388 |
97.30% |
91.20% |
95.7% |
|
Arabidopsis lyrata |
Coding |
37026 |
94.20% |
62.90% |
96.9% |
|
|
Non-coding |
795 |
95.60% |
100% |
98.2% |
|
Oryza sativa |
Coding |
37389 |
96.50% |
78.90% |
95.30% |
|
|
Non-coding |
1011 |
100% |
100% |
100% |
含有短ORF的人类RNA(短肽): 使用PLEKv2来预测含有短ORF的人类RNA。PLEKv2测试使用的数据来自CPPred,包括641个编码RNA和641个lncRNA。结果表明,PLEKv2的预测准确率为89.2%,显著高于CPPred(准确率为80.66%)。这表明,PLEKv2即使在处理复杂的短RNA序列时也能保持着高性能。
五、结论与展望
PLEKv2作为PLEK工具的全新升级版,不仅在技术上实现了突破,更在实际应用中展现了卓越的性能。随着生物医学研究的不断深入,PLEKv2有望在未来的研究中发挥更大的作用,为科研人员提供更加精准的RNA序列分类工具。
六、数据和材料的可用性
PLEKv2的开放源代码可以在https://sourceforge.net/projects/plek2/上在线获取。
论文地址(开放访问):https://doi.org/10.1186/s12864-024-10662-y
七、参考文献
Aimin Li, Haotian Zhou, Siqi Xiong, Junhuai Li, Saurav Mallik, Rong Fei, Yajun Liu, Hongfang Zhou, Xiaofan Wang, Xinhong Hei, Lei Wang. PLEKv2: predicting lncRNAs and mRNAs based on intrinsic sequence features and the coding-net model. BMC Genomics 2024, 25(1):756. https://doi.org/10.1186/s12864-024-10662-y
Aimin Li, Junying Zhang*, Zhongyin Zhou. PLEK: a tool for predicting long non-coding RNAs and messenger RNAs based on an improved k-mer scheme. BMC Bioinformatics, 2014, 15(1): 311~314. https://doi.org/10.1186%2F1471-2105-15-311


 浙公网安备 33010602011771号
浙公网安备 33010602011771号