轮廓系数、方差比、DB指数(三种常见的聚类内部评价指标)
1 引言
在之前的一篇文章(https://www.cnblogs.com/emanlee/p/17742869.html)中掌柜详细介绍了聚类算法中几种常见的评估指标,包括纯度(准确率)、精确率、召回率、兰德系数和F值等。虽然这些评价指标都能很好的评估聚类结果的优劣,但是它们都有着一个共同的缺点,需要知道训练样本的真实标签(ground truth),而这在现实情况中却很难做到。
对于聚类结果的评价方法一般可以分为内部评估法(internal evaluation)与外部评估方法(external evaluation)。所谓外部评估方法是指在知道真实标签(ground truth )的情况下来评估聚类结果的好坏,也就是上面提到几种评估指标。一般来说在做论文,或者是有少量的标注数据时,都可以用外部评估法选择一个相对最优的聚类模型,然后再应用到其它未被标记的数据中。所谓内部评估法则是不借助于外部信息,仅仅只是依靠聚类结果和样本本身的属性来进行评估的办法。常见的内部评估法有轮廓系数(Silhouette Coefficient)、Calinski-Harabasz Score和Davies-Bouldin score等。在接下来的这篇文章中,将会分别就这3中内部评价指标进行介绍。
2 Silhouette Coefficient Index
所谓轮廓系数(Silhouette Coefficient)[1],它本质上衡量的是每个样本点到其簇内样本的距离与其最近簇结构之间距离的比值。如果该比值越小,则说明该样本点所在的簇结构与其最近簇结构之间的距离越远,从而说明此时的聚类结果越好。
2.1 原理
在介绍轮廓系数原理之前,首先需要明白的是对于聚类结果的轮廓系数,它实际上是每个样本点轮廓系数的平均值。这也就是说聚类样本中的每个样本点对应都有一个轮廓系数值,而总的轮廓系数则是所有样本点轮廓系数的平均值。

图 1. 轮廓系数原理图
如图1所示,数据样本中一共包含有3个簇结构,对于最左边这个簇中(圆形)的样本点 i 来说,距离其最近的簇为中间(方形)这个簇。现在定义样本 i到簇中每个样本距离的均值为 a(i),到最近簇中每个样本距离的均值为 b(i),即此时根据图1所示的结果有:

这里需要注意的一点是,对于同一个簇中的每个样本点来说,距离自己最近的簇可能并不是同一个;同时,在寻找距离当前样本点最近的簇结构时,计算的是当前样本点到各个簇中心的最短距离,而不是计算当前样本点所在簇的簇中心到其它每个簇中心的最短距离。
此时,样本 i 的轮廓系数 s(i) 定义为

从式(2) 可以看出,当 a(i) 越小而 b(i) 越大时,此时对应的情况是样本所在的簇中所有样本点之间距离都比较近(簇内距离较小),
样本所在的簇距离其最近簇的距离较远(因为此时样本所在的簇中簇内距离较小,所以样本 i 到其它簇的距离可以近似地看做样本所在簇到其它簇之间的距离),所以
s(i) 此时也就越接近于1,即聚类效果越好。
类似地,当 b(i) 越小而 a(i) 越大时,此时对应的情况是样本所在的簇中所有样本点之间距离都比较远(簇内距离较大),样本i所在的簇距离其最近簇的距离较近,所以
此时 s(i) 也就越接近于-1,即聚类效果越差。
最后,当样本 i 所在的簇中只有样本一个样本时,那么对于样本到底属于哪个簇就变得不清楚了,此时可以粗略地认为属于离它最近的簇中,所以此时有 a(i) = b(i),即
s(i) =0 表示一种中立的情况。
由此可知 s(i) 的取值范围应该是

,且进一步式(2)
可以改为为

最终,整个聚类结果的轮廓系数结果为

其中 n 表示样本点的个数。
2.2 实现
在介绍完轮廓系数的原理和计算方式后,下面掌柜就来介绍如何编程实现这一过程。具体实现如下所示:
1 def get_silhouette_coefficient(X, labels):
2 n_clusters = np.unique(labels).shape[0]
3 s = []
4 for k in range(n_clusters): # 遍历每一个簇
5 index = (labels == k) # 取对应簇所有样本的索引
6 x_in_cluster = X[index] # 去对应簇中的所有样本
7 for sample in x_in_cluster: # 计算每个样本的轮廓系数
8 a = ((sample - x_in_cluster) ** 2).sum(axis=1)
9 a = np.sqrt(a).sum() / (len(a) - 1) # 去掉当前样本点与当前样本点的组合计数
10 nearest_cluster_id = None
11 min_dist2 = np.inf
12 for c in range(n_clusters): # 寻找距离当前样本点最近的簇
13 if k == c:
14 continue
15 centroid = X[labels == c].mean(axis=0)
16 dist2 = ((sample - centroid) ** 2).sum()
17 if dist2 < min_dist2:
18 nearest_cluster_id = c
19 min_dist2 = dist2
20 x_nearest_cluster = X[labels == nearest_cluster_id]
21 b = ((sample - x_nearest_cluster) ** 2).sum(axis=1)
22 b = np.sqrt(b).mean()
23 s.append((b - a) / np.max([a, b]))
24 return np.mean(s)在上述代码中,第1行X和labels分别是原始训练集和聚类得到的标签;第2行是取聚类结果的簇数量;第4-6行是遍历每个簇并取对应中所有的样本;第7行开始是遍历每一个样本点来计算轮廓系数;第8-9行是计算当前样本距离其簇中每个样本点之间的平均距离,其中第9行分母减去1是因为要去掉当前样本与当前样本之间距离为0的情况;第10-19行是计算得到里当前样本点最近的簇,其中15行是计算簇中心;第20行是取距离当前样本点最近的簇对应的所有样本点;第21-22行是计算当前样本点到其最近簇中所有样本的平均距离;第23行是计算当前样本点对应的轮廓系数并保存;第24行是返回所有样本对应的轮廓系数的均值。
所有完整代码均可从本仓库获取:https://github.com/moon-hotel/MachineLearningWithMe
完成上述代码实现之后,便可以通过如下方式进行使用:
1 import numpy as np
2 from sklearn.cluster import KMeans
3 from sklearn.datasets import load_iris
4 from sklearn.metrics import silhouette_score
5
6 def test_silhouette_score():
7 x, y = load_iris(return_X_y=True)
8 model = KMeans(n_clusters=3)
9 model.fit(x)
10 y_pred = model.predict(x)
11 print(f"轮廓系数 by sklearn: {silhouette_score(x, y_pred)}")
12 print(f"轮廓系数 by ours: {get_silhouette_coefficient(x, y_pred)}")
13
14
15 if __name__ == '__main__':
16 test_silhouette_score()上述代码运行结束后的结果如下:
1 轮廓系数 by sklearn: 0.5528190123564091
2 轮廓系数 by ours: 0.55281901235641013 Calinski-Harabasz Index
在介绍完轮廓系数后我们再来看第2种内部评价指标Calinski-Harabasz指数[2]。由于Calinski-Harabasz指数的本质是簇间距离与簇内距离的比值,且整体计算过程与方差计算方式类似,所以又将其称之为方差比准则。
如果还有不知道怎么有效入门机器学习的朋友,掌柜强烈推荐阅读下面这篇文章,里面详细介绍了掌柜所推荐的通过五个步骤和三个阶段的方法来高效学习机器学习的路线,同时也按顺序整理了客栈了所有与机器学习相关的文章。
3.1 原理
方差比准则的计算过程相较于轮廓系数更加简单,我们只需要计算出所有簇的簇内距离和然后再比上所有簇的簇间距离和即可,原理如图2所示。
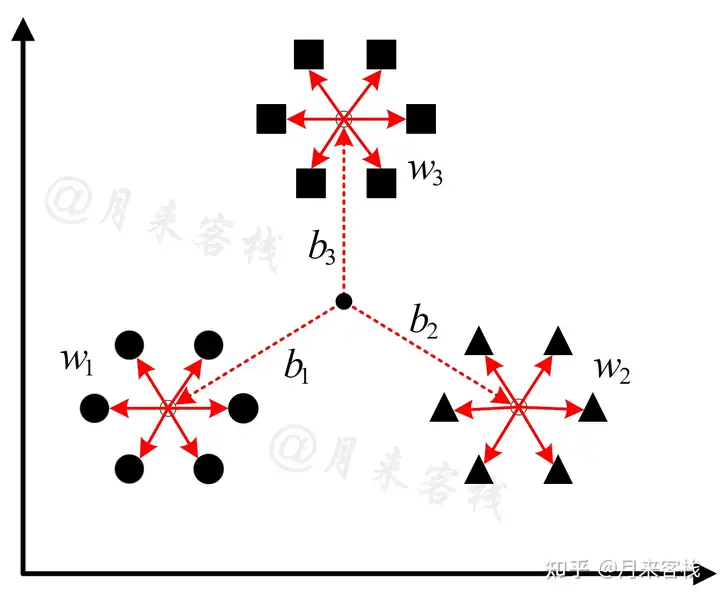
图 2. 方差比计算原理图
如图2所示一共包含有3个聚类簇,每个簇中均有6个样本点,且每个簇内部的白色圆点表示对应簇的簇中心。
表示第个簇对应的簇内距离和,即每个样本点减去簇中心的平方和;表示第个簇的簇中心到全局簇中心(即所有样本点的中心)的加权距离,而方差比则是所有簇内距离和与簇间距离和的比值,即

其中K
表示簇数量,表示第个簇中的所有样本,表示第个簇对应的簇中心,表示第个簇中样本的数量,表示所有样本的数量,
表示全局簇中心。
值得一提的是,由于每个簇的簇中心到全局簇中心的距离只会计算一次,在计算方差比时为了消除与簇内距离上的差异所以还乘上了每个簇对应的样本总数;同时,在计算方差时采用是方差的无偏估计。
根据式
可知,如果越大,则表示相对于越大,此时的簇间距离便远大于簇内距离,即簇与簇之间相距较远聚类结果好;相反,如果越小,则表示簇与簇之间相距较近聚类效果较差。同时,可以看出方差比的取值范围应该是
。
3.2 实现
在清楚了方差比的计算原理后,实现起来就比较容易了,整体实现代码如下:
1 def get_calinski_harabasz(X, labels):
2 n_samples = X.shape[0]
3 n_clusters = np.unique(labels).shape[0]
4 betw_disp = 0. # 所有的簇间距离和
5 within_disp = 0. # 所有的簇内距离和
6 global_centroid = np.mean(X, axis=0) # 全局簇中心
7 for k in range(n_clusters): # 遍历每一个簇
8 x_in_cluster = X[labels == k] # 取当前簇中的所有样本
9 centroid = np.mean(x_in_cluster, axis=0) # 计算当前簇的簇中心
10 within_disp += np.sum((x_in_cluster - centroid) ** 2)
11 betw_disp += len(x_in_cluster) * np.sum((centroid - global_centroid) ** 2)
12
13 return (1. if within_disp == 0. else
14 betw_disp * (n_samples - n_clusters) /
15 (within_disp * (n_clusters - 1.)))在上述代码中在上述代码中,第1行X和labels分别是原始训练集和聚类得到的标签;第6行是计算全局簇中心;第7-9行是开始遍历每一个簇并计算得到每个簇对应的簇中心;第10行是累计每个簇对应簇内距离的总和;第11行是累计每个簇中心到全局中心的距离总和;第13-15行则是返回最终计算得到方差比结果。
完成上述代码实现之后,便可以通过如下方式进行使用:
1 def test_calinski_harabasz_score():
2 x, y = load_iris(return_X_y=True)
3 model = KMeans(n_clusters=3)
4 model.fit(x)
5 y_pred = model.predict(x)
6 print(f"方差比 by sklearn: {calinski_harabasz_score(x, y_pred)}")
7 print(f"方差比 by ours: {get_calinski_harabasz(x, y_pred)}")
8
9 if __name__ == '__main__':
10 test_calinski_harabasz_score()上述代码运行结束后的结果如下:
1 方差比 by sklearn: 561.62775662962
2 方差比 by ours: 561.627756629624 Davies-Bouldin Index
在介绍完前面两种聚类内容部评价指标后我们再来看第3种评价方法Davies-Bouldin Index(DB指数)[3]。DB指数的核心思想是计算每个簇与之最相似簇之间相似度,然后再通过求得所有相似度的平均值来衡量整个聚类结果的优劣。如果簇与簇之间的相似度越高(DB指数偏高),也就说明簇与簇之间的距离越小(直观上理解只有相距越近的事物才会约相似),那么此时聚类结果就越差,反之亦然。
4.1 原理
在DB指数中,簇与簇之间的相似度被定义为两个簇的簇内直径和与簇间距离的比值,即图3中
与
的比值。
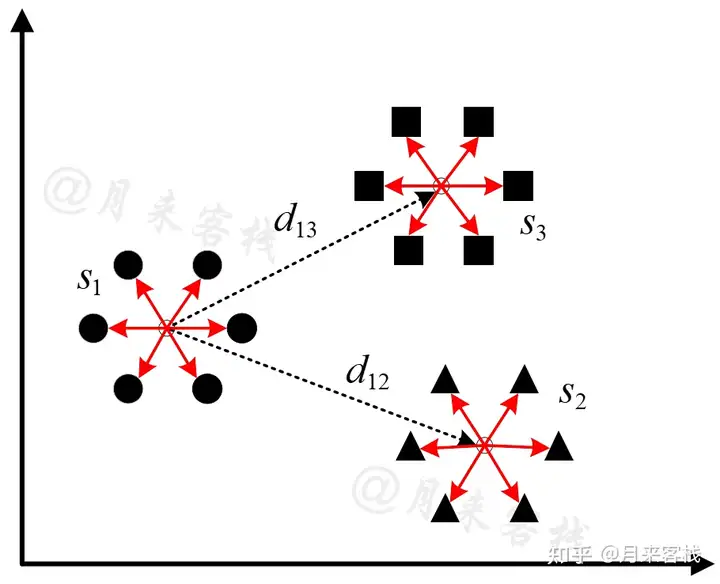
图 3. DB指数计算原理图
如图3所示一共包含有3个聚类簇,每个簇中均有6个样本点,且每个簇内部的白色圆点表示对应簇的簇中心。
表示第个簇中所有样本点到簇中心距离的平均值,又称之为簇内直径;表示第个簇与第个簇之间的距离(即两个簇中心之间的距离)。此时,定义簇与簇
之间的相似度为
进一步,DB指数的计算公式为
其中
表示总的簇数量。
从公式
可以看出,对于第个簇的相似度来说,其等于中的最大值;而DB指数则是所有簇相似度的平均值。虽然计算过程很清晰明了也容易实现,但是为什么要取最大值呢,有什么含义?如图3所示,对于第个簇来说其相似度值是还是
呢?
根据式

