eQTL
首先QTL是数量性状位点,比如身高是一个数量性状,其对应的控制基因的位点就是一个数量性状位点,而eQTL就是控制数量性状表达位点,即能控制数量性状基因(如身高基因)表达水平高低的那些基因的位点。
数量性状基因座:控制数量性状的基因在基因组中的位置称数量性状基因座。常利用DNA分子标记技术对这些区域进行定位,与连续变化的数量性状表型有密切关系
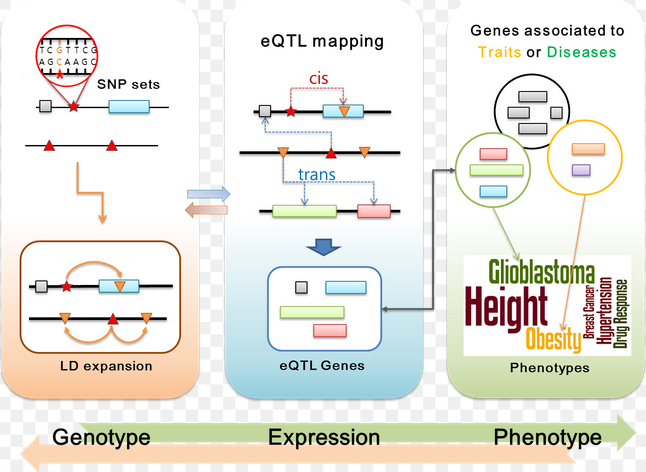
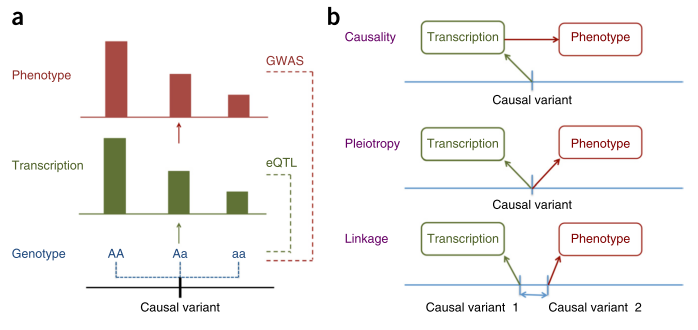
表达数量性状基因座(expression Quantitative Trait Loci,eQTL)是对上述概念的进一步深化,它指的是染色体上一些能特定调控mRNA和蛋白质表达水平的区域,其mRNA/蛋白质的表达水平量与数量性状成比例关系。eQTL可分为顺式作用eQTL和反式作用eQTL,顺式作用eQTL就是某个基因的eQTL定位到该基因所在的基因组区域,表明可能是该基因本身的差别引起的mRNA水平变化;反式作用eQTL是指某个基因的eQTL定位到其他基因组区域,表明其他基因的差别控制该基因mRNA水平的差异。
eQTL就是把基因表达作为一种性状,研究遗传突变与基因表达的相关性: 就好像研究遗传突变与身高的相关性一样
早年可以通过同时做一个个体的SNP芯片和cDNA芯片, 在全基因组尺度研究突变与表达的相关性, 这种研究需要较多个体(例如1000个); 现在随着深度测序的出现,很多人开始用RNA-Seq在较少量个体中研究allele-specific expression,本质上就是eQTL.
简单地说, 遗传学研究经常发现一些致病或易感突变, 这些突变怎样导致表型有时候不太直观; 所以用某个基因的差异表达作为过渡: 突变A-->B基因表达变化-->表型;
From https://www.cnblogs.com/Acceptyly/p/3904108.html
=====================================================
=====================================================
从基因的改变到疾病等现象的出现,中间缺失了重要的一环,那就是基因的表达。也许在测序中,我们可以看到某一个基因上某一个位置的变化(比如说SNP单核苷酸变化),但是这种变化并不一定会影响mRNA的产生或者蛋白的改变。也就有可能不会影响到疾病或其他生物学过程。于是科学家想到了另一个指标——mRNA的序列数据。因为只有被表转译到mRNA上的基因,才可能进一步表达为蛋白(图1)。

图1:eQTL是沟通基因改变与疾病的桥梁
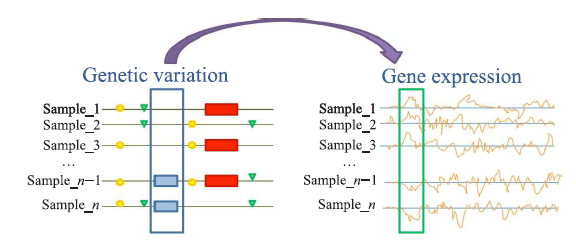
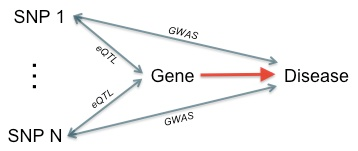
但是要怎么搞清DNA改变是怎么影响mRNA的出现呢?这一过程被称为Expression quantitative trait loci(eQTL) 分析,目的在于得到单个DNA突变与单个基因表达量之间的相关性。与单个基因mRNA表达量相关的DNA突变,就被称为eQTL。
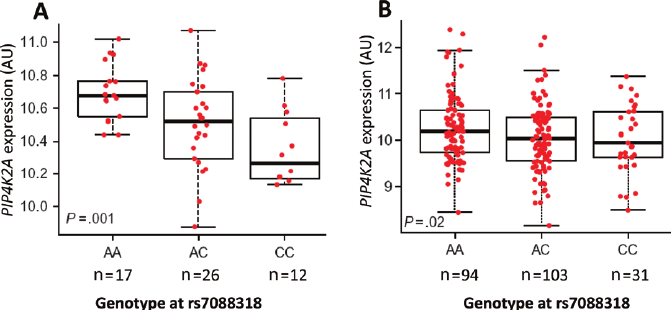
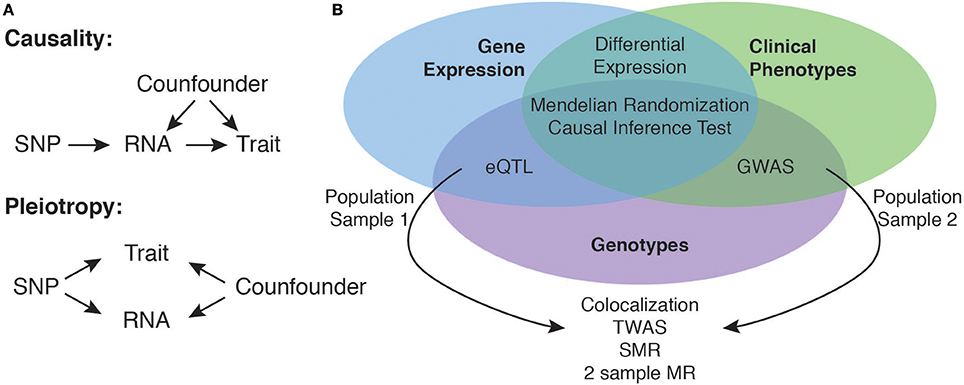
简单来讲,我们首先通过全基因组测序获得每个个体的DNA全序,然后以同种族的其他个体作为参照,标记出该个体所有的DNA变异位点, 称为SNP位点。同时,我们通过全基因组mRNA表达量测序得到该个体的特定组织样本中的基因表达量。以全部DNA变异位点为自变量,轮流以每种mRNA表达量为因变量,用大量的个体数据做样本进行线性回归,就可以得到每一个SNP位点和每一个mRNA表达量之间的关系。
GTEx是第一个收集了多个人体器官mRNA测序的数据库,并提供了跨器官的eQTL研究平台。
当前使用的GTEx v6p版本的原始数据来自于449名生前健康的遗体捐献者的44个不同的器官。图2是不同器官里面样本数的直观展示。由这个图可以看出,这一数据库中涉及的数据覆盖面非常广,数据量大,具有重要的应用潜力。
图2:GTEx 样品取材来源图示。灰色字体为 cis-eQTL 数/trans-eQTL数
链接:https://www.jianshu.com/p/2e1e9d3ccd63
=====================================================
=====================================================
常见问题
eQTL分析可以研究哪些疾病?
近年来许多科学家运用eQTL联合GWAS分析的方法,寻找致病风险位点,这些联合分析的方法已应用在类风湿性 关节炎、肾病、精神分裂等疾病的研究中。
eQTL对非编码区功能怎样进行研究?
通过eQTL分析,可以解释非编码区基因突变与基因表达之间的调控关系。系统的了解基因转录的调控机制,构建 基因表达调控网络。
eQTL分析怎样进行药物处理前后反应的研究?
对药物处理前后的机体/细胞进行eQTL的差异分析,可以很好的研究药物处理前后反应的差异,为寻找药物作用靶 点,以及药物作用机制提供基因层面的证据。
怎样利用eQTL分析进行人群队列研究?
在进行人群队列研究中,可以进行eQTL分析,通过比较不同人群中eQTL的差异,充实人群队列研究结果,寻找不同 人群中基因差异与基因表达调控的关系。
from http://www.novogene.com/tech/service/eQTL/QA/
=====================================================
=====================================================
eQTLs, the discovery of genetic variants that explain variation in gene expression levels.
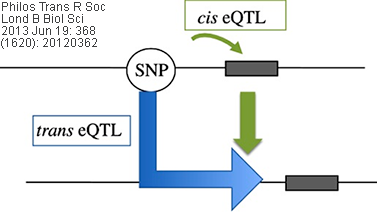
An eQTL is a locus that explains a fraction of the genetic variance of a gene expression phenotype. Standard eQTL analysis involves a direct association test between markers of genetic variation with gene expression levels typically measured in tens or hundreds of individuals. This association analysis can be performed proximally or distally to the gene. One of the major advantages of eQTL mapping using the GWAS approach is that it permits the identification of new functional loci without requiring any previous knowledge about specific cis or trans regulatory regions. Typically in the eQTL mapping literature, regulatory variants have been characterized as either cis or trans acting, reflecting the predicted nature of interactions and of course depending on the physical distance from the gene they regulate. In this review, conventionally, variants within 1 Mb (megabase) on either side of a gene's TSS were called cis (figure 1), while those at least 5 Mb downstream or upstream of the TSS or on a different chromosome were considered trans acting.
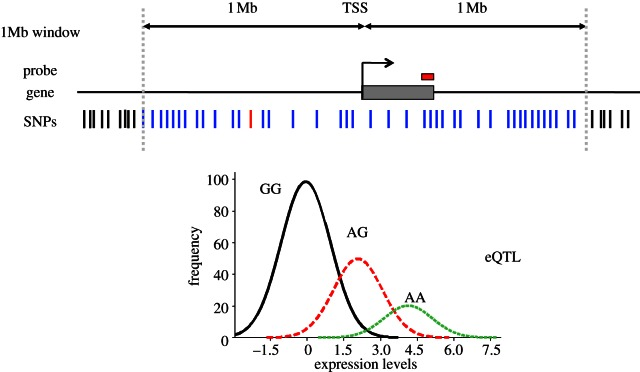
A typical eQTL; many SNPs tested against levels of expression measured by a probe or by other means. The panel below illustrates the difference in distributions of expression values stratified by the SNP genotype of the most significant SNP. (Online version in colour.)
From:
https://www.ncbi.nlm.nih.gov/pmc/articles/PMC3682727/
=====================================================

=====================================================
Web resources
GTEx Portal: https://www.gtexportal.org/
GWAS Catalog: https://www.ebi.ac.uk/gwas/
1000 Genomes Project: http://www.internationalgenome.org/
PLINK 1.9: https://www.cog-genomics.org/plink2
HaploReg v4.1: https://pubs.broadinstitute.org/mammals/haploreg/haploreg.php
=====================================================
=====================================================
=====================================================
=====================================================
=====================================================
=====================================================

=====================================================
=====================================================

=====================================================
=====================================================
=====================================================


 浙公网安备 33010602011771号
浙公网安备 33010602011771号